KARAKTERISTIK FENOTIF ISOLAT KLINIK Escherichia coli O157:H7 PADA MEDIA SORBITOL MAC CONKEY AGAR (SMAC)
on
J. Agric. Sci. and Biotechnol.
ISSN: 2302-0113
Vol. 7, No. 1, Juli 2018
KARAKTERISTIK FENOTIF ISOLAT KLINIK Escherichia coli O157:H7 PADA MEDIA SORBITOL MAC CONKEY AGAR (SMAC)
Wahyu Hidayati1*), I Gede Rai Maya Temaja2, Ni Nengah Dwi Fatmawati3
-
1 Mahasiswa Magister Bioteknologi Pertanian Fakultas Pertanian Universitas Udayana
-
2. Fakultas Pertanian Universitas Udayana,
-
3. Departemen Mikrobiologi Klinik Fakultas Kedokteran Universitas Udayana Denpasar
*)Email: hidayatiwahyu.fkunud@gmail.com
Abstract
Phenotypic Characteristic of Escherichia coli O157: H7 Clinical Isolates on MacConkey Agar with Sorbitol (SMAC)
Escherichia coli is a Gram-negative, rod-shaped bacterium, and commonly has flagella. Most of E. coli strains are normal flora in digestive tract of human, but some serotypes are pathogen for human and animal. One of pathogenic E. coli strain that causes a severe infection in humans (hemorrhagic colitis and hemolytic uremic syndrome) is known as Enterohemorrhagic Escherichia coli (EHEC) with cattle as their natural reservoir. E. coli O157:H7 is the most important and pathogenic serotype of EHEC which responsible for outbreak of Hemorrhagic Colitis and Hemolytic Uremia Syndrome. Accurate and cheap method detection of E. coli O157:H7 is needed to help early detection diagnosis and therapy. Therefore, the aim of this study is investigate phenotypic characteristics of E. coli O157:H7 isolated from clinical specimens using MacConkey Agar with Sorbitol (SMAC) media. Three E. coli clinical isolates from Sanglah General Hospital showed colorless non-sorbitol fermenting colony in SMAC media as phenotypic characteristic of E. coli O17:H7, therefore SMAC may beused on are of confirmation methods for E. coli O157:H7 detection from clinical isolats.
Keyword: E. coli O157:H7, Phenotype, SMAC
Escherichia coli (E. coli) adalah bakteri berbentuk batang, Gram negatif, dan biasanya mempunyai flagella. Bakteri ini termasuk dalam famili Enterobactericeae genus Escherichia dan bersifat aerob. Kebanyakan bersifat flora normal pada usus manusia. Namun demikian beberapa serotipe dari bakteri ini dapat menjadi patogen dalam tubuh manusia maupun hewan (Nataro & Kaper, 1998). Strain patogen memiliki beberapa faktor virulensi yang tidak dimiliki oleh strain non patogen (Aranda et al, 2004).
Enterohemorragic Escherichia coli (EHEC) merupakan salah satu bakteri E. coli patogen yang sangat berbahaya bagi kesehatan manusia karena dapat menyebabkan penyakit yang fatal seperti Hemorrhagic colitis (HC) yang ditandai dengan gejala khas yaitu diare berdarah dan Hemolytic Uremic Syndrome (HUS) yaitu infeksi saluran kencing yang menyebabkan gangguan fungsi ginjal. Diantara beberapa serotipe EHEC, yang paling berbahaya adalah serotipe O157:H7 (Suwito, 2009). Bakteri ini masih menjadi perhatian dunia karena sering menyebabkan wabah. Insiden O157:H7 pertama kali terjadi pada tahun 1982 yaitu terjadinya diare berdarah hingga Hemolytic Uremic Syndrome (HUS) (CDC, 1982). Tahun 2000 wabah E.coli O157:H7 terjadi di Amerika dan diperkirakan ada 78.480 kasus setiap tahunnya yang menyebabkan 2.168 pasien rawat inap, 61 mengalami kematian dan banyak anak yang mengalami gagal ginjal akut (Chad. et al, 2012).
Di Indonesia belum pernah dilaporkan adanya wabah diare yang disebabkan oleh bakteri ini. Namun demikian Indonesia juga masih menghadapi risiko terinfeksi bakteri ini. Hal ini disebabkan karena masih banyaknya penggunaan produk susu sapi, daging sapi, air yang terkontaminasi oleh tinja sapi maupun terkontaminasi oleh tinja manusia yang terinfeksi oleh bakteri ini (Rey et al, 2006). Bakri dkk pernah melakukan identifikasi bakteri E. coli O157:H7 pada tahun 2010, di wilayah Makasar, dimana sampel diambil dari penderita diare. Sebanyak 28 sampel yang diperiksa didapatkan 9 sampel (32,14%) positif E. coli O157:H7 (Bakri. et al, 2010)
Di bidang mikrobiologi identifikasi secara fenotif dapat dilakukan dengan beberapa metode antara lain menggunakan mesin Vitek 2 Compact ( Biomereux) yang merupakan teknologi mesin otomatis di bidang mikrobiologi. Program mesin Vitek 2 compact ( Biomereux) ini menentukan nilai positif atau negatif dengan cara mengukur intensitas cahaya dengan scanner optical (Shetty. et al, 1998; Blondel. et al, 2002).
Kelemahan mesin ini pada identifikasi E. coli O157:H7 adalah hasil analisa dari mesin ini hanya menyebutkan E. coli O157 saja, tanpa mengidentifikasi antigen H. Untuk dapat mengidentifikasi E. coli O157:H7 secara fenotif dapat dilakukan menggunakan media Sorbitol Mac Conkey Agar (SMAC), yaitu media selektif dan diferensial untuk E. coli O157:H7. Bakteri ini tidak dapat memfermentasi sorbitol sehingga koloni akan tampak berwarna jernih atau tidak berwarna. Hal ini yang membedakan O157:H7 dengan non O157:H7 (Nataro, 1998). Identifikasi menggunakan media SMAC ini merupakan identifikasi yang cepat dan dapat diandalkan, sehingga biaya identifikasi menjadi lebih murah, diagnosa klinis juga dapat ditegakkan dengan cepat (March, 1986)
Identifikasi O157:H7 yang lebih cepat dan murah diperlukan terutama pada spesimen klinis, sehingga tujuan penelitian ini adalah mengetahui karakteristik fenotif E. coli O157 dari isolat klinis RSUP Sanglah Denpasar
menggunakan media Sorbitol Mac Conkey Agar (SMAC), sehingga dapat mempercepat diagnosis dan terapi.
Sampel dalam penelitian ini adalah isolat klinis E. coli O157 yang terisolasi dari spesimen klinis tahun 2017, yang teridentifikasi secara fenotif menggunakan mesin Vitek 2 compact (Biomerieux). E. coli ATCC 8739 digunakan sebagai kontrol.
-
2.2 Metode
Sampel yang secara fenotif sudah teridentifikasi O157 menggunakan Vitek 2 Compact (Biomerieux) dikultur pada media Sorbitol Mac Conkey Agar (SMAC) dan Mac Conkey Agar (MC). Diinkubasi pada suhu 37oC selama 24 jam. Pengamatan dilakukan setelah inkubasi selesai.
Berdasarkan data laboratorium Mikrobiologi RSUP Sanglah selama tahun 2017, didapatkan 3 isolat klinis yang positif E. coli O157. Isolat klinis didapatkan dari sampel urin RSUP Sanglah Denpasar. Data isolat tersebut terangkum dalam tabel1:
Tabel 1. Hasil Vitek 2 Compact pada isolat klinis O157
|
No |
Kode Hasil Keterangan Sampel |
|
1 2 3 |
U141 Escherichia coli O157 Highly Pathogenic Organism U47 Escherichia coli O157 Highly Pathogenic Organism L39 Escherichia coli O157 Highly Pathogenic Organism |
-
3.1.2 Identifikasi Menggunakan Media Mac Conkey Agar dan Sorbitol Mac Conkey Agar
Hasil identifikasi E. coli dari mesin Vitek 2 Compact (Biomerieux) dikonfirmasi menggunakan media Mac Conkey Agar sebagai pembanding dan Sorbitol Mac Conkey Agar. E.coli ATCC 8739 digunakan sebagai kontrol untuk penelitian ini. Hasil kultur pada MC dan SMAC ditunjukkan pada gambar 1 dan tabel 2
A
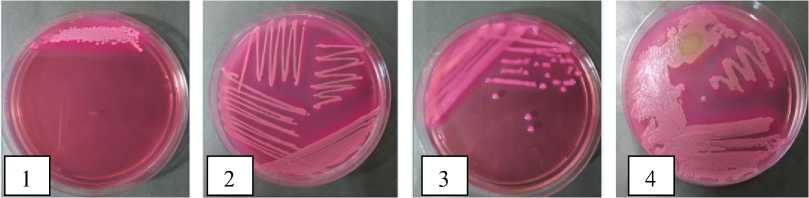
B
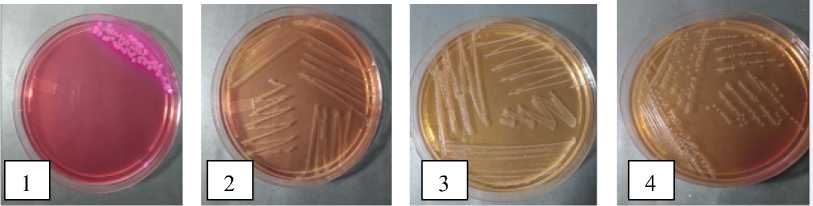
Gambar 1. Karakteristik Escherichia coli pada media agar. Pertumbuhan Escherichia coli pada media Mac Conkey Agar (A), koloni Escherichia coli ATCC 8739 (A1), isolat klinis U141 (A2), isolat klinis U47 (A3), isolat klinis L39 (A4). Pertumbuhan pada media Sorbitol Mac Conkey Agar (B), Escherichia coli ATCC 8739 (B1), isolat klinis U141 (B2), isolat klinis U47 (B3), dan isolat klinis L39 (B4)
Hasil pengamatan menunjukkan bahwa E. coli O157 dari Vitek 2 Compact yang dikultur pada media MC menunjukkan warna merah muda/pink pada semua isolat sehingga sulit dibedakan. Sedangkan pada media SMAC koloni isolat klinis berwarna jernih/bening, sementara pada kontrol berwarna merah muda/pink.
Tabel 2. Hasil kultur Escherichia coli O157:H7 pada media Mac Conkey Agar dan
|
Sorbitol Mac Conkey Agar | |
|
No. |
Hasil Kode Sampel Mac Conkey Sorbitol Mac Conkey Agar |
|
ATCC berwarna merah muda Berwarna merah muda U141 berwarna merah muda Jernih (tidak berwarna) U47 berwarna merah muda Jernih (tidak berwarna) L39 berwarna merah muda Jernih (tidak berwarna) |
|
3.2 |
Pembahasan |
Penelitian ini dilakukan untuk mengetahui apakah E. coli O157 yang berasal dari isolat klinis yang sudah diidentifikasi menggunakan mesin Vitek 2 Compact ( Biomeurex) tersebut adalah E. coli O157:H7 dengan menggunakan media SMAC. Penanaman pada media Mac Conkey Agar (MC) baik kontrol maupun isolat klinis menunjukkan warna merah muda/pink, sehingga tidak dapat dibedakan antara kontrol dan isolat klinik Escherichia coli O157.
Hasil dari media SMAC menunjukkan koloni warna jernih pada isolat klinis yang menandakan bahwa isolat tersebut tidak memfermentasi sorbitol dan bersifat motil (O157:H7). Tidak seperti kebanyakan strain Escherichia coli, serotipe O157:H7 hampir 95% tidak memfermentasi sorbitol, sehingga cara identifikasi sederhana untuk strain ini adalah dengan melakukan kultur pada media Sorbitol Mac Conkey Agar. Laktosa yang ada di dalam media Mac conkey diganti dengan sorbitol. Media SMAC merupakan media yang selektif untuk mendeteksi Escherichia coli serotipe O157:H7. Sorbitol digunakan sebagai penanda atau media isolasi utama dalam mendeteksi Escherichia coli O157:H7 (Rappaport, 1952).
Meskipun hampir seluruh isolat E. coli O157 tidak memfermentasi sorbitol, sebanyak 5% adalah strain Escherichia coli O157 non motil (O157:NM) yang mampu memfermentasi sorbitol. Meskipun bersifat non motil, E. coli strain ini juga bersifat patogen dan mampu menginfeksi manusia sejak teridentifikasi di Jerman tahun 1988. Namun demikian Escherichia coli O157:NM lebih jarang diisolasi dari sampel manusia dibandingkan Escherichia coli O157:H7 (Karch, et al. 2001: Weber, 2011: Alpers, et al, 2009).
SMAC memiliki sensitifitas 100% dan spesifitas 85% untuk mendeteksi Escherichia coli O157:H7, dan terbukti menjadi cara yang murah, cepat dan sederhana untuk bakteri serotipe tersebut (March, 1986).
Secara fenotif seluruh isolat klinis E. coli O157 yang digunakan dalam penelitian ini adalah E. coli O157:H7 yang diidentifikasi menggunakan media SMAC, sehingga SMAC dapat digunakan sebagai media untuk konfirmasi E. coli O157:H7 dari isolat klinis yang ditunjukkan dengan warna jernih/bening.
Terimakasih kepada Departemen/ KSM Mikrobiologi Klinik FK Unud/RSUP Sanglah dan Instalasi Mikrobiologi Klinik RSUP Sanglah yang telah memberikan tempat untuk melakukan penelitian dan mengijinkan penggunaan sampel dari isolat klinis E. coli O157.
Daftar Pustaka
Alpers, K, Werber, D, Frank, C et al. 2009. Sorbitol‐fermenting
enterohaemorrhagic Escherichia coliO157:H‐ causes another outbreak of haemolytic uraemic syndrome in children. Epidemiol Infect. ;137: 389– 395
Aranda K.R.S, Fagundes-Neto U, and Scaletskyl I.C.A. 2004: Evaluation of Multiplex PCRs for Diagnosis of Infection with Diarrheagenic Escherichia coli and Shigella spp. JCM.42(12):5849-5853
Bakri Z; Hatta M; Nasrum M. 2010. Deteksi Keberadaan Bakteri Escherichia coli O157:H7 pada Feses penderita diare dengan Metode Kultur dan PCR. JST Kesehatan; (5), 2:184-192
Blondel., HE, Hetchler., D, el al, 2003, Evaluation of Vitek 2 for Analysis of Enterobactericeae using the Advance Expert System (AES) versus Interpretive Susceptibility Guideline used at Dynacare Kasper Medical Laboratories, Edmonton, Alberta, Clinical Microbiology and Infection, 9(11): 1091-1103
Chad., I, et al, 2012, Shiga Toxins and the Pathophysiology of Hemolytic Uremic Syndrome in Human and Animal, Toxin Journal, 4: 1261-1287
Center for Disease Control, 1982. Isolation of E.coli O157:H7 from sporadic cases of hemorrhagic colitis-United States. WMWR Morb Mortal Wlky Rep;31:580-585
Karch, H and Bielaszewska, M. 2001 Sorbitol‐fermenting Shiga
toxin‐producing Escherichia coli O157:H(‐) strains: epidemiology,
phenotypic and molecular characteristics, and microbiological diagnosis. J Clin Microbiol; 39: 2043–2049
March., SB, Ratnam.,S, et al. 1986. Sorbitol Mac Conkey Medium for Detection of Escherichia coli O157:H7 Associated with Hemorrhagic Colitis. J.Clin.Microbiol. 23:869-872
Nataro JP,Kaper JB,1998. Diarrheagenic Escherichia coli. Clin Microbial Rev 11:142-201
Rappaport F., and Henig.E.1952. Media For The Isolation and Defferntiation of Pathogenic Escherichia coli Serotypes O111 & O55. J.Clin.Pathol; 5:361362
Rey, JS. Sanchez, J.E. Blanco. 2006. Prevalence, Serotypes and Virulance genes of Shiga Toxin Producing Escherichia coli Isolated From Ovine and Caprine milk and other Dairy Product in Spain. International Journal of Food Microbiology. 107(2): 212-217
Shetty.,N, Hill.,G, Ridgway.,GL, 1998. The Vitek analyser for routine Bacterial Identification and Susceptibility testing: Protocol, Problem and Pitfalls, J Clin Pathol; 51:316-323
Suwito W, 2009. Escherichia coli Verotoxigenik diisolasi dari susu sapi. JIVT 14(3):237-242
Werber, D, Bielaszewska, M, Frank, C, Stark, K, and Karch, H. 2011. Watch out for the even eviler cousin‐sorbitol‐fermenting E.
coli O157. Lancet. ; 377: 298–299
https://ojs.unud.ac.id/index.php/JASB
40
Discussion and feedback