DETEKSI GEN PspC PADA ISOLAT KLINIS BAKTERI Streptococcus pneumoniae DI RUMAH SAKIT UMUM PUSAT SANGLAH DENPASAR DARI JANUARI 2012 – MARET 2017 DENGAN MENGGUNAKAN POLYMERASE CHAIN REACTION (PCR)
on
ISSN: 2597-8012 JURNAL MEDIKA UDAYANA, VOL. 8 NO.6,JUNI, 2019
∏∩Λ l≈≈ OsTnta
journals ........"
DETEKSI GEN PspC PADA ISOLAT KLINIS BAKTERI Streptococcus pneumoniae DI RUMAH SAKIT UMUM PUSAT SANGLAH DENPASAR DARI JANUARI 2012 – MARET 2017 DENGAN MENGGUNAKAN POLYMERASE CHAIN REACTION (PCR)
Kadek Anggie Wigundwipayana1*, Ni Nyoman Sri Budayanti2, Ni Nengah Dwi Fatmawati2
1Program Studi Pendidikan Dokter, Fakultas Kedokteran, Universitas Udayana 2DepartemenMikrobiologi KlinikFakultas Kedokteran Universitas Udayana/RS Sanglah E-mail: wigundwipayana@gmail.com
ABSTRAK
Bakteri Streptococcus pneumoniae merupakan penyebab berbagai penyakitnon-invasif seperti otitis media, maupun penyakit invasif seperti meningitis, bakteremia, dan pneumonia.S. pneumoniae memiliki beberapa faktor virulensi, salah satunya PspC yang merupakan CBP dengan karakteristik adhesin yang berinteraksi dengan vitronectin, laminin receptor, polymeric immunoglobulin receptor (plgR), dan penghambat komplemen faktor H dan C4b-binding protein. Adapun tujuan dari penelitian ini adalah untuk mendeteksi gen PspC pada isolat klinis bakteri S. pneumoniae di RSUP Sanglah Denpasar dengan menggunakan PCR.Penelitian ini menggunakan desain cross sectional dengan menggunakan 21sampel yang diambil dari sputum (42,9%), luka (23,8%), dan darah (33,3%). Sebanyak 10 (47,6%) dari 21 isolat klinis S. pneumoniae memiliki gen PspC.
Kata Kunci: Streptococcus pneumoniae, faktor virulensi, PspC, PCR
ABSTRACT
Streptococcus pneumoniae is an infectious agent of various non-invasive diseases such as otitis media, as well as invasive diseases such as meningitis, bacteremia, and pneumonia. S. pneumoniae has several virulence factors, one of them is PspC which is a CBP with adhesin characteristics that interact with vitronectin, laminin receptor, polymeric immunoglobulin receptor (plgR), and complement inhibitor factor H and C4b-binding protein. The purpose of this study was to detect the PspC gene in clinical isolates of S. pneumoniae bacteria at Sanglah Hospital Denpasar using PCR. This study used cross sectional design using 21 samples taken from sputum (42.9%), wound (23.8%), and blood (33.3%). A total of 10 (47.6%) of 21 clinical isolates of S. pneumoniae had the PspC gene.
Keywords: Streptococcus pneumoniae, virulence factors, PspC, PCR

PENDAHULUAN
Streptococcus pneumoniae
(peumokokus) merupakan bakteri gram positif penyebab infeksi yang menimbulkan angka kesakitan dan kematian yang signifikan di seluruh dunia, baik pada anak-anak maupun pada orang dewasa.1
S. pneumoniae merupakan flora normal pada nasofaring manusia, namun pada kondisi tertentu bakteri ini dapat menyebabkan berbagai penyakit non-invasif seperti otitis media maupun penyakit invasif seperti meningitis, bakteremia, dan pneumonia.Penyakit
pneumokokus invasif/IPD (Invasive
Pneumococcal Disease) dideskripsikan sebagai kondisi dimana S. pneumoniae ditemukan di dalam darah, cairan serebrospinal, atau tempat steril lainnya di dalam tubuh.2
Dalam proses terjadinya penyakit, S. pneumoniae memiliki beberapa faktor virulensi, diantaranya; kapsul polisakarida, protein-protein permukaan, enzim, toksin pneumolisin, autolisin, dan beberapa faktor virulensi minor lainnya.2Penelitian di Bali yang menggunakan 21 sampel menunjukkan distribusi dari serotipe 19F, 23F, 6A/B, 7F dan 15B/C.4
Sistem kekebalan tubuh manusia terdiri dari berbagai mekanisme pertahanan, seperti komplemen, pembersihan mukosiliar, fagosit, berbagai antibodi, dan T-sel efektor.5Sistem ini akan menjadi pertahanan dari infeksi pneumokokus. Pertahanan hospes dan virulensi dari pneumokokus mempengaruhi apakah akan menjadi penyakit atau tidak. Selain itu ada faktor lingkungan yang juga berpengaruh dalam patogenesis penyakit.8
Infeksi pneumokokus bertanggung jawab untuk sekitar 1 juta kematian per tahun pada anak usia di bawah 5 tahun, dimana lebih
dari 90% pada masyarakat kelas bawah, khususnya di Sub-Sahara Afrika, Amerika Latin, dan Asia. Selain faktor geografis, beberapa faktor tambahan lainnya adalah usia, kebiasaan merokok, dan adanya penyakit pada individu misalnya HIV/AIDS, yang juga mempengaruhi terjadinya infeksi pneumokokus.3
Banyak penelitian sudah dilakukan sebelumnya untuk mendapatkan penjelasan mengenai bakteri S. pneumoniae, tetapi masih belum bisa menjawab semua pertanyaan tentang faktor virulensinya.8
Penelitian ini bertujuan untuk mengetahui gen PspC yang terdeteksi dari isolat klinis S. pneumoniae yang terisolasi di Laboratorium Mikrobiologi Klinis RSUP Sanglah Denpasar dari Januari 2012 – Maret 2017.
BAHAN DAN METODE
Peremajaan Bakteri
Stok isolat klinis bakteri S. pneumoniae yang terisolasi dari spesimen klinis (darah, sputum, dan luka)di Laboratorium Mikrobiologi Klinik RSUP Sanglah dari Januari 2012 – Maret 2017 disimpan pada suhu -70°C.Isolat kemudian direjuvenasi pada media agar darah dan diinkubasi pada suhu 37°C, selama 24 jam.Pengujiandrug susceptibility testisolat dilakukan dengan Vitek-2 (Biomereux) berdasarkan CLSI di Laboratorium Mikrobiologi Klinik RSUP Sanglah Denpasar.
Pengambilan Koloni dan Isolasi DNA
Koloni bakteri S. pneumoniae yang tumbuh pada media agar darah kemudian dipanen dan dilakukan isolasi DNA. Proses isolasi DNA menggunakan metode Boil Cell
DIRECTORY OF OPEN ACCESS JOURNALS

Exctraction (BCE) dengan terlebih dahulu mensuspensi koloni bakteri pada TE dengan pH 8. Suspensi kemudian dipanaskan selama 10 menit pada suhu 100°C.Selanjutnya didinginkan menggunakan es selama 1 menit, kemudian disentrifius 8.000 rpm selama 1 menit.Sebanyak 75 μl supernatan disimpan pada suhu -20°C hingga digunakan sebagai cetakan DNA.7
PCR untuk Mendeteksi Gen PspC
DNA bakteri S. pneumoniae terisolasi digunakan untuk PCR.PCR untuk mendeteksi gen penyandi faktor virulensi PspC dikerjakan dengan menggunakanGo Taq®Green Master Mix. Uniplex PCR dengan konsentrasi PCR Master Mix (Promega) 5 μL, primer PspC-F dan PspC-R masing-masing 0,3 μL, nuclease free water 3,6 μL, dan DNA template 0,8 μL sehingga volume total menjadi 10 μL.7
Tabel 1. Sekuens primer gen penyandi faktor virulensi PspC.7
|
Primer |
Sekuens (5’-3’) |
Amplikon(b p) |
|
PspC-F |
TCCAATTAGATA GAAGAAAACAT ACCC |
160 |
|
PspC-R |
TGCTTAAAAGCT GCGGTTAA |
Tabel 2. Program PCR.
Tahapan Suhu Waktu Siklus
Pre-
95°C 2 menit 1 kali
denaturasi
Denaturasi
95°C 30 detik 35 kali
|
Annealing |
50°C |
30 detik |
35 kali |
|
Ekstensi |
72°C |
30 detik |
35 kali |
|
Final ekstensi |
72°C |
5 menit |
1 kali |
Elektroforesis
Amplikon yang dihasilkan kemudian dilakukan elektroforesis menggunakan gel agarose 2% yang ditambahkan 1 μL Biotium Gel Red™ Nucleic Acid .Elektroforesis menggunakan TBE 0,5xpada tegangan sebesar 100 volt selama 35 menit.Selanjutnya, DNA divisualisasikan menggunakan Gel Doc ™ XR (Bio-Rad). Ukuran pita yang terbentuk dianggap positif bila memiliki ukuran 160bp.9,10
DNA Sequencing
Sequencing dilakukan untuk mengkonfirmasi bahwa pita amplikon yang diperoleh merupakan pita PspC yang diharapkan.Sequencing dilakukan dengan menggunakan BigDye Terminator v3.1 Cycle Sequencing Kit yang dilakukan di First Base (Malaysia). Hasil sequencing divisualisasi dan diedit dengan Mega version 7.0, kemudian dibandingkan dengan data pada GenBank (www.ncbi.gov/BLAST).
HASIL
Dalam penelitian ini, sebanyak 21 isolat klinis bakteri S. pneumoniae digunakan sebagai sampel.Seluruh sampel didapatkan dari Laboratorium Mikrobiologi Klinik RSUP Sanglah dari Januari 2012 – Maret 2017. Sampel diambil dari sputum (n=9; 42,9%), luka
DIRECTORY OF OPEN ACCESS JOURNALS

(n=5; 23,8%), darah (n=7; 33,3%).Stok isolatS. pneumoniae tumbuh homogen dan tidak ditemukan adanya kontaminasi saat ditumbuhkan pada media agar darah (Gambar 1).
Gambar 1.Koloni S. pneumoniae pada media agar darah
|
Tabel 3.Distribusi Sampel Berdasarkan Tahun | |
|
Jenis Sampel |
Tahun Jumlah (%) 2012 2013 2014 2015 2016 2017 |
|
Sputum |
0 0 0 6 2 1 9 (42,9) |
|
Luka |
0 0 1 1 2 1 5 (23,8) |
|
Darah |
4 0 0 1 2 0 7 (33,3) |
|
Jumlah( %) |
4 (19) 0 (0) 1 (4,8) 8 (38,1) 6 (28,6) 2 (9,5) 21 (100) |
Teknik PCR digunakan untuk mengamplifikasitarget gen PspC dari seluruh sampel S.
pneumoniae dengan menggunakan primer spesifik.
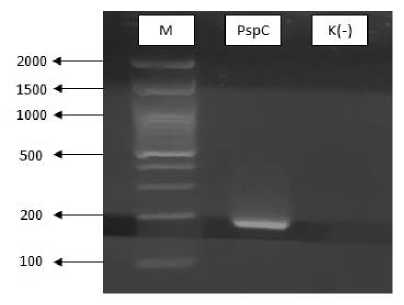
Gambar 2.Elektroforegram PCR untuk mendeteksi gen PspC pada suhu annelaling 50°C. Keterangan : M = Marker 100 bp DNA ladder, K(-) = Kontrol negatif
DIRECTORY OF OPEN ACCESS JOURNALS

Ukuran produk PCR untuk gen PspC adalah 160 bp, gen PspC dinyatakan positif jika pada elektroforesis hasil PCR ditemukan pita pada 160 bp dan dinyatakan negatif jika pada elekroforeis hasil PCR tidak ditemukan pita pada 160 bp.
Setelah tahap PCR, dari 21 sampel S. pneumoniae ditemukan 10 sampel (47,6%) positif terhadap gen PspC dan 11 (52,4%) sampel dinyatakan negatif karena tidak mencapai target 160 bp(Gambar 2).Sampel yang positif berasal dari jenis sampel sputum (n=5; 23,8%), luka (n=1; 4,8%), dan darah (n=4; 19,0%)yang ditemukan pada tahun 2012, 2015, 2016, dan 2017 (Tabel 4).
Tabel 4. Hasil PCR PspC dari Berbagai Jenis Sampel S. pneumoniae di RSUP Sanglah Denpasar dari Januari 2012 - Maret 2017.
|
Jenis Sampel |
Hasil Jumlah PspC[+] PspC[-] (%) |
|
Sputum |
5 4 9 (42,8) |
|
Luka |
1 3 4 (19,2) |
|
Darah |
4 4 8 (38,0) |
|
Jumlah (%) |
10 (47,6) 11 (52,4) 21 (100) |
Untuk mengkonfirmasi bahwa pita yang teramplifikasi benar merupakan segmen gen PspC, maka dilakukan DNAsequencing dengan BLAST (LT669720.1). Hasil sequencing produk PCR menunjukkan bahwa pita yang teramplifikasi merupakan segmen gen PspC.
PEMBAHASAN
Bakteri S. pneumoniae merupakan agen infeksi penyebab berbagai penyakit; non-invasif seperti otitis media, maupun penyakit invasif seperti meningitis, bakteremia, dan pneumonia.2S. pneumoniae memiliki beberapa faktor virulensi, salah satunya PspC yang merupakan CBP dengan karakteristik adhesin yang berinteraksi dengan vitronectin, laminin receptor, polymeric immunoglobulin receptor (plgR), dan penghambat komplemen faktor H
dan C4b-binding protein. Karakteristik yang dimiliki PspC ini sangat berperan penting dalam patogenesis pneumokokus untuk terjadinya suatu infeksi.6
Sebuah studi untuk mendeteksi gen PspC pada bakteri S. pneumoniae sudah dilakukan sebelumnya di Mashhad, Iran. Penelitian tersebut menyatakan bahwa dari 59 sampel terdeteksi hanya 2 sampel (3,38%) positif memiliki gen PspC.7 Sedangkan pada penelitian ini, 10 (47,6%) dari 21 sampel memiliki gen PspC. Adanya perbedaan hasil penelitian yang signifikan disebabkan oleh perbedaan asal sampel, dimana pada penelitian sebelumnya didapatkan dari kolonisasi nasofaring anak sehat umur dibawah 6 tahun7, sedangkan pada penelitian ini sampel diperoleh dari sputum, luka dan darah pada pasien di RSUP Sanglah.

Metode pada penelitian sebelumnya melakukan isolasi DNA dengan DNAase Tissue kit (KIAGEN, Tehran, Iran), sedangkan pada penelitian ini isolasi DNA dilakukan dengan metode Boil Cell Exctraction pada suhu 100oC selama 10 menit. Setelah itu dimasukkan ke dalam es suhu 0oC selama 1 menit(heat-shock) untuk melisiskan sel dan mengeluarkan DNA pada permukaan.
Proses PCR penelitian sebelumnya, pre-denaturasi dengan suhu 94oC selama 2 menit, denaturasi 94oC selama 10 detik, annealing 58oC selama 15 detik, ekstensi selama 1 menit (denaturasi, annealing, dan ekstensi menggunakan 25 siklus pengulangan). Final ekstensi pada suhu 72oC selama 5 menit.7PCR pada penelitian ini dikerjakan pre-denaturasi dengan suhu 95oC selama 2 menit, denaturasi 95oC selama 30 detik, annealing 50oC selama 30 detik, ekstensi selama 30 detik (denaturasi, annealing, dan ekstensi
menggunakan 35 siklus penguulangan). Final ekstensi pada suhu 72oC selama 5 menit.
Penelitian yang menggunakan 4 sampel serotipe yaitu 1, 7F, 19F, dan 23F
menunjukkan gen PspC pada isolat serotipe 7F, 19F, dan 23F memiliki homologi tertinggi (98%) dengan varian alel PspC-3.9, sementara isolat 1 serotipe memiliki kesamaan tertinggi (98%) pada varian alel PspC-5.1, PspC-5.2, dan PspC-5.3.7
Adapun tujuan dari penelitian ini adalah untuk mengetahui distribusi gen PspC pada isolat klinis S. pneumoniae di Laboratorium Mikrobiologi Klinik RSUP Sanglah Denpasar, akan tetapi belum dapat menjelaskan faktor-faktor yang mempengaruhi distribusinya tersebut. Oleh sebab itu,
diperlukan penelitian lebih lanjut untuk mengetahui hubungan antar keduanya.
SIMPULAN
Berdasarkan hasil penelitian ini dapat disimpulkan bahwa 10 (47,6 %) dari 21 (100 %) sampel memiliki gen penyandi faktor virulensi PspC, sedangkan 11 (52,4%) sampel lainya
menunjukkan hasil yang negatif terhadap gen PspC.
SARAN
Pada penelitian selanjutnya diharapkan dapat menggunakan jumlah sampel yang lebih besar sehingga memberikan pemahaman yang lebih baik mengenai faktor virulensi yang dimiliki oleh bakteri S. pneumoniae.
UCAPAN TERIMA KASIH
Penulis mengucapkan terimakasih kepada Fakultas Kedoteran Universitas Udayana dan Rumah Sakit Umum Pusat Sanglah Denpasar telah memberi dukungan, dan Wahyu Hidayati selaku staf Molekuler Mikrobiologi Departemen Mikrobiologi Klinik FK Unudserta Ni Wayan Nilawati selaku staf di Instalasi Mikrobiologi Klinik RSUP Sanglah telah membantu secara teknis.
DAFTAR PUSTAKA
-
1. Adegbola, R.A., et all.Carriage of Streptococcus pneumoniae and Other Respiratory Bacterial Pathogens in Low and Lower- Middle Income Countries: A Systematic Review and Meta-Analysis. 2014;9(8).
-
2. AM Mitchell, TJ Mitchell. Streptococcus pneumoniae: virulence factors and
variation. European Society of Clinical Microbiology and Infectious Disease. 2010;16:411-418.
DIRECTORY OF OPEN ACCESS JOURNALS

-
3. Chaguza, C., Cornick, J.E. and Everett, 7.
D.B. Mechanisms and impact of genetic recombination in the evolution of Streptococcus pneumoniae. CSBJ.
2015;13: 241–247. Available at:
http://dx.doi.org/10.1016/j.csbj.2015.03.00 7.8.
-
4. Fatmawati, N.N.D., Tarini, N,M,A., and Mayura I,P,B. Multiplex PCR for
Biosciences and Biotechnology
Consortium. 2015;2(2):93–99.
Access.2011;15(7):1057–1063.
Streptococcus pneumoniae and formation of choline binding protein-DNA complexes. Environmental Microbiology Reports.2015;7(5):715-727.
Moghaddam TG, Rad M, Mousavi SF, Ghazvini K. Detection of lytA, pspC and rrgA genes in Strepococcus pneumoniae isolated from healthy children. IranianJournal of Microbiology. 2015;7(3): 156-160.
Velasco, E.A., Verheul, A.F.M., Verhoef, J. and Snippe, H. Streptococcus pneumoniae: Virulence Factors , Pathogenesis , and Vaccines. 1995;59(4): 591–603.
Maestro B and Sanz JM. Choline Binding Protein from Streptococcus pneumoniae : A Dualrole as Enzybiotics and Targets for the Design of New Antimicrobials. MDPI. 2016;5: 2-33.
Desa MNM, Parasakthi N, Vadivelu and Sekaran. Expression analysis of adherence-associated genes in pneumococcal clinical isolates during adherence to human respiratory epithelial cells (in vitro) by real time PCR. FEMS Microbiol Lett. 2008; 288: 125-130.
Discussion and feedback