DESAIN PELACAR DNA SECARA IN SILICO SEBAGAI PENDETEKSI RESISTENSI FLUOROQUINOLONE PADA ISOLAT MULTI DRUG RESISTANT TUBERCULOSIS
on
Cakra Kimia (Indonesian E-Journal of Applied Chemistry)
Volume 7 Nomor 2, Oktober 2019
DESAIN PELACAR DNA SECARA IN SILICO SEBAGAI PENDETEKSI RESISTENSI FLUOROQUINOLONE PADA ISOLAT MULTI DRUG RESISTANT TUBERCULOSIS
Sagung Chandra Yowani1,2, Jennifer Tamara2, Ayu Nyoman Chandra Yustiana2, Putu Sanna Yustiantara1,2
-
1 Kelompok Studi MDR-TB & XDR-TB, FMIPA, Universitas Udayana, Bukit Jimbaran, Bali-Indonesia, 80361 cyowani@gmail.com
-
2 Program Studi Farmasi, FMIPA, Universitas Udayana, Bukit Jimbaran, Bali-Indonesia, 80361 jenniferrtamara@gmail.com
ABSTRAK: Resistensi fluoroquinolone (FQ) pada Multi Drug Resistant Tuberculosis (MDR-TB) umumnya disebabkan oleh adanya sejumlah mutasi pada beberapa gen yang mengkode sensitivitas Mycobacterium tuberculosis dan sebagian besar terjadi pada gen gyrA. Mutasi pada kodon 94 gen gyrA merupakan mutasi yang paling sering terjadi dengan 7 variasi perubahan asam amino. Resistensi FQ dapat dideteksi menggunakan DNA probe yang spesifik agar dapat memberi terapi yang tepat pada pasien. Penelitian ini akan mendesain urutan nukleotida TaqMan probe menggunakan program Clone Manager Suite 9.2. Hasil rancangan DNA probe kemudian dianalisis dalam 2 tahap. Tahap pertama berdasarkan kriteria probe secara umum yaitu panjang (18-30 basa), %GC (40-60%), Tm (5-10°C lebih tinggi dibanding Tm primer), runs (≤ 4), repeats (≤ 4), dimer (≤ 4), dan tidak terbentuk hairpin. Selanjutnya tahap kedua berdasarkan kriteria pelabelan TaqMan probe, yaitu tidak terdapat basa G pada 2 nukleotida di ujung 5’ dan jumlah basa C ≥ G. Rancangan DNA probe mutan menggunakan program menghasilkan 1 probe untuk mutasi spesifik D94G. Probe tersebut dianalisa dengan kriteria probe secara umum dan kriteria pelabelan TaqMan probe. Kesimpulan dari penelitian ini yaitu hasil rancangan probe mutan A94MG1 dengan urutan 5’ – TCGATCTACGGCAGCCTGGT – 3’ telah memenuhi kriteria pelabelan TaqMan probe dan dapat digunakan untuk mendeteksi adanya mutasi pada kodon 94 gen gyrA Mycobacterium tuberculosis.
Kata kunci: MDR-TB, gen gyrA, in silico, TaqMan probe, Real-Time PCR
ABSTRACT: Fluoroquinolone (FQ) resistance in Multi Drug Resistant Tuberculosis (MDR-TB is generally caused by some mutations in several genes that encode the sensitivity of Mycobacterium tuberculosis and most of them occur in gyrA gene. Mutations in codon 94 gyrA gene are the most common mutations with 7 variations in amino acid changes. FQ resistance can be detected using a spesific DNA probe to provide the precise therapy for the patient. This research will design the TaqMan probe nucleotide sequence using the Clone Manager Suite 9.2. program. The results of designing DNA probes were then analyzed by 2 stages. The first stage is based on criteria of the probe in general which is length (18-30 bases), GC% (40-60%), Tm (5-10°C higher than primer Tm), runs (≤ 4), repeats (≤ 4), dimer (≤ 4), and hairpin is not formed. Second stage is examination based on the labeling criteria for TaqMan, that is no G base at 2 nucleotides at the end of 5’ and the amount of bases C ≥ G. The mutant DNA probe design using the program produced 1 probe for the D94G specific mutation. The probe was analyzed with general criteria and TaqMan probe labeling. The conclusion of this study is A94MG1 mutant probe design have met the TaqMan probe labeling criteria and can be used to detect mutations in the Mycobacterium tuberculosis gyrA gene codon 94.
Keywords: MDR-TB, gyrA gene, in silico, TaqMan probe, Real-Time PCR
Multi Drug Resistant Tuberculosis (MDR-TB) didefinisikan sebagai penyakit TB yang resisten terhadap setidaknya dua Obat Anti Tuberkulosis (OAT) lini pertama yaitu isoniazid dan rifampisin. Data World Health Organization (WHO) menunjukkan bahwa 3,5% kasus baru TB dan 18% kasus yang sudah ditangani mengalami MDR-TB. Pada kasus MDR-TB tahun 2017, 8,5% diantaranya diperkirakan berkembang menjadi Extensively Drug Resistant Tuberculosis (XDR-TB) [1].
Pasien MDR-TB telah resisten terhadap OAT lini pertama sehingga digunakan OAT lini kedua. Menurut WHO, golongan fluoroquinolone (FQ) merupakan obat inti lini kedua yang digunakan pada terapi MDR-TB, sehingga resistensi terhadap FQ dapat menyebabkan kegagalan terapi dan menjadi penanda kondisi XDR-TB [2].
Resistensi pada MDR-TB umumnya disebabkan oleh terjadinya sejumlah mutasi pada beberapa gen yang mengkode sensitivitas Mycobacterium tuberculosis terhadap OAT. FQ bekerja dengan menghambat DNA gyrase (salah satu DNA topoisomerase tipe II) dan topoisomerase IV yang diperlukan oleh bakteri untuk melakukan replikasi [3]. DNA gyrase merupakan target utama ikatan FQ pada bakteri karena tidak adanya topisomerase IV dalam genom bakteri tersebut [4, 5]. DNA gyrase terdiri dari 2 subunit A dan 2 subunit B yang masing-masing dikode oleh gen gyrA dan gen gyrB [6]. M. tuberculosis resisten terhadap FQ terutama melalui mutasi di daerah penentu mutasi yang disebut dengan Quinolone Resistance Determining Regions (QRDR) [7,8]. Daerah QRDR gen gyrA berada diantara kodon 74-113 [4,9], sedangkan daerah gen gyrB berada diantara kodon 500-540 [8].
Pada kasus TB, resistensi FQ sebagian besar disebabkan oleh mutasi yang terjadi pada gen gyrA, sedangkan mutasi pada gen
gyrB jarang terjadi dengan prevalensi sebesar 1,6-4,1% [5,10,11]. Mutasi pada gen gyrA menyebabkan terjadinya perubahan asam amino seperti G88A, D89G, A90V, S91P, D94N, D94Y, D94G, D94A, D94H, D94C dan D94V [5,6,8]. Berdasarkan penelitian sebelumnya, daerah yang paling sering terjadi mutasi ditemukan pada gen gyrA kodon 94 dengan mutasi D94G sebesar 11,6-42,6%.
Salah satu cara mengetahui resistensi pada MDR-TB dilakukan dengan pendeteksian cepat agar dapat memberikan terapi yang tepat pada pasien. Deteksi M. tuberculosis yang resisten terhadap FQ dapat dilakukan dengan metode molekuler, yaitu metode Real Time Polymerase Chain Reaction (RT-PCR). RT-PCR memiliki komponen yang sama dengan PCR ditambah dengan DNA probe berupa molekul asam nukleat yang memiliki afinitas kuat dan dapat berikatan spesifik dengan target DNA. Jenis probe yang sering digunakan pada metode RT-PCR yaitu TaqMan probe. Probe jenis ini digunakan untuk mendeteksi adanya mutasi dan skrining subtipe spesifik dari mikroba patogen [12]. TaqMan probe merupakan urutan oligonukleotida yang memiliki pewarna reporter fluoresen pada ujung 5’ dan pewarna quencher pada ujung 3’ [13]. TaqMan probe mudah didesain dan disintesis, tetapi jika tidak dirancang dengan baik maka dapat terbentuk struktur sekunder seperti dimer, hairpins, runs, dan repeat selama proses RT-PCR [14].
Desain DNA probe atau pelacak DNA merupakan tahap yang kritis dan harus spesifik terhadap urutan DNA target untuk menghindari hibridisasi silang pada urutan yang memiliki banyak persamaan [15]. Desain DNA probe akan dibuat dengan perangkat lunak (software) Clone Manager Suite 9.2.. Penelitian ini difokuskan untuk mendesain DNA probe yang memenuhi kriteria secara umum dan kriteria pelabelan
TaqMan probe. Daerah mutasi yang dipilih dalam perancangan DNA probe yaitu daerah QRDR gen gyrA M. tuberculosis pada kodon 94 dengan jenis mutasi D94G. Kodon tersebut dipilih sebagai target perancangan DNA probe didasarkan pada penelitian sebelumnya yaitu memiliki frekuensi mutasi yang paling sering terjadi dan berkaitan dengan resistensi FQ.
Bahan yang digunakan untuk mendesain probe yaitu urutan nukleotida gen gyrA M. tuberculosis H37Rv dari database URL://www.ncbi.nlm.nih.gov dengan Accession No. NC_000962.3. Satu pasang primer gen gyrA terdiri dari primer forward (gen-gyrA-F) dengan urutan 5’-GCAGCTACATCGACTATGC-3’ dan
primer reverse (gen-gyrA-R) dengan urutan 5’-GGCTTCGGTGTACCTCATC-3’.
Primer gen gyrA tersebut merupakan desain terbaik secara in silico dan setelah dilakukan optimasi, suhu annealing yang paling optimal untuk gen gyrA adalah suhu 58°C. Primer tersebut telah terbukti mampu mengamplifikasi gen gyrA [16]. Penelitian desain DNA probe gen gyrA M. tuberculosis menggunakan data panjang dan Tm dari primer secara in silico sebagai acuan untuk menentukan kriteria perancangan DNA probe.
Peralatan
Peralatan yang digunakan dalam penelitian ini diantaranya yaitu komputer dengan spesifikasi Windows 10 32 bit,
koneksivitas jaringan internet, dan software Clone Manager Suite 9.2.
Daerah mutasi yang menjadi target perancangan DNA probe yaitu daerah QRDR gen gyrA M. tuberculosis yang terdiri dari 2517 pb. DNA probe untuk gen gyrA M. tuberculosis akan dirancang di
dalam daerah amplifikasi pada nukleotida 77 – 396. Pada Gambar 1, dapat dilihat bahwa daerah amplifikasi berada di dalam garis berwarna hitam dan sepasang primer yang membatasi daerah amplifikasi yang ditandai dengan warna hijau dan mencakup posisi terjadinya mutasi pada gen gyrA M. tuberculosis. Pemilihan kodon pada daerah QRDR gen gyrA sebagai target perancangan DNA probe mutan didasarkan pada frekuensi mutasi yang paling sering terjadi dan berkaitan erat dengan resistensi FQ yaitu pada kodon 94. Posisi kodon 94 ditandai dengan warna merah pada Gambar 1. Perancangan urutan DNA probe mutan mengacu pada perubahan nukleotida dari mutasi D94G dimana terjadi perubahan basa di posisi nukleotida ke 281 dari GAC menjadi GGC pada kodon 94.

Gambar 1. Urutan Nukleotida Gen gyrA M. tuberculosis Target Perancangan DNA Probe
Perancangan DNA Probe
Perancangan DNA probe dilakukan secara in silico menggunakan program Clone Manager Suite 9.2. Langkah awal yang dilakukan yaitu mengunduh urutan nukleotida gen gyrA M. tuberculosis H37Rv dengan Accession No.
NC_000962.3 dalam format data FASTA dan disimpan dalam notepad sebagai template. Probe dirancang untuk
mendeteksi mutasi D94G sehingga dilakukan penggantian basa pada template untuk memperoleh urutan probe mutan yang spesifik terhadap mutasi. Program Clone Manager Suite 9.2 dijalankan dan diklik ‘Primer’ lalu pilih ‘Design’ lalu dimasukkan data sekuens gen gyrA M. tuberculosis dan klik ‘OK’. Kemudian pada ‘Primer Type’ pilih ‘Probe’ dan pada kolom ‘Design Detail’ dimasukkan panjang
DNA probe yang divariasikan dari 18-30 basa pada bagian ‘length’ dan diatur daerah target yang diinginkan, lalu klik ‘OK’. Daerah target pada gen gyrA berada pada kodon 94 posisi nukleotida ke 281. Selanjutnya program akan memberikan beberapa data sekuens DNA probe dan diklik ‘Primer report’ untuk menampilkan urutan nukleotida, panjang, komposisi dan kriteria dari probe.
-
3. HASIL DAN PEMBAHASAN
Pemilihan Sekuens Target Rancangan DNA Probe
Pemilihan sekuens target rancangan DNA probe dilakukan penelusuran pustaka untuk mengetahui daerah pada gen gyrA M. tuberculosis yang paling sering terjadi mutasi dan menyebabkan resistensi FQ. Perancangan DNA probe dilakukan pada daerah QRDR gen gyrA M. tuberculosis dimana mutasi yang terjadi akan mengubah struktur QBP dan menyebabkan FQ tidak dapat berikatan dengan DNA gyrase. DNA probe akan dirancang pada kodon 94 karena kodon tersebut merupakan daerah kodon yang paling sering terjadi mutasi dan menghasilkan variasi asam amino sebanyak 7 jenis. Penelitian ini akan merancang DNA probe untuk mendeteksi mutasi dengan prevalensi tertinggi yaitu D94G. Perancangan DNA probe mutan dilakukan menggunakan urutan nukleotida wild-type dari gen gyrA M. tuberculosis yang dapat diperoleh secara online dari database URL://www.ncbi.nlm.nih.gov dengan Accession No. NC_000962.3.
Hasil Perancangan DNA Probe
Probe merupakan urutan nukleotida yang memiliki afinitas kuat dengan target spesifik sehingga dapat digunakan untuk
mendeteksi adanya mutasi. Penelitian ini akan mendesain probe berjenis TaqMan. Keuntungan utama penggunaan TaqMan probe meliputi spesifisitas tinggi, rasio signal-to-noise yang tinggi, dan memiliki kemampuan untuk reaksi multipel. Sedangkan kelemahannya yaitu biaya probe yang tinggi [17]. Perancangan DNA probe mutan dapat dilakukan secara in silico menggunakan beberapa program diantaranya yaitu Alleleid, Beacon Designer, Clone Manager Suite 9.2., Probe Maker, dan Oligo7. Penelitian ini menggunakan program Clone Manager Suite 9.2. karena ketersediaan program dan kode lisensi yang diperlukan untuk mengaktivasi program sehingga dapat digunakan secara lengkap. Program akan menghasilkan probe yang memenuhi kriteria yang telah ditetapkan. Pada penelitian sebelumnya, program Clone Manager Suite 9.2. telah digunakan untuk perancangan DNA probe mutan sehingga program tersebut terbukti dapat menghasilkan urutan DNA probe [18].
Perancangan DNA probe dilakukan dengan menjalankan program Clone Manager Suite 9.2. lalu dilakukan desain probe dengan memasukkan kriteria probe secara umum diantaranya yaitu memiliki panjang 18-30 basa, %GC 40-60%, Tm probe lebih tinggi 5-10°C daripada Tm primer, dimer ≤ 4, runs ≤ 4, repeats ≤ 4, dan tidak ada hairpins. Selanjutnya diatur daerah target terjadinya mutasi. Posisi mutasi berpengaruh terhadap hibridisasi dari probe dimana mismatch antara probe dan target akan menyebabkan efisiensi hibridisasi probe menurun. Pada kondisi tersebut, enzim Taq polymerase akan memanjangkan target probe tanpa mendegradasi probe, sehingga fluoresensi reporter menjadi tidak terdeteksi [19].
Tabel 1. Kriteria Hasil Perancangan DNA Probe Mutan Untuk Deteksi Mutasi Spesifik D94G
|
Nama |
Umtan |
Kriteria |
Desain DNA Probe Secara In Silico | |
|
Probe |
Nukleotida Probe |
%GC |
Dimer Runs Repeat Haijpm | |
|
Desain probe spesifik untuk mutasi D94G | ||||
|
A94MG1 |
Tcgatctacg Gcagcctggt |
20 67 |
60 |
4 2 - - |
Pada tahap ini, program akan menghasilkan beberapa data sekuens DNA probe mutan dan diklik ‘Primer report’ untuk menampilkan urutan nukleotida, panjang, komposisi dan kriteria lainnya dari probe yang dihasilkan. Probe diberi nama dan dimasukkan ke dalam tabel beserta kriterianya. Penamaan probe dilakukan dengan huruf dan angka, misal A94MG1 artinya A menunjukkan gen gyrA M. tuberculosis, 94 menunjukkan kodon target, M menunjukkan jenis rancangan probe mutan, G menunjukkan singkatan asam amino yang terbentuk akibat mutasi, dan 1 menunjukkan nomor probe yang dihasilkan. Hasil perancangan probe mutan untuk mutasi spesifik D94G terdapat sebanyak 1 sekuens yang ditunjukkan pada Tabel 1.
Hasil Analisis Rancangan DNA Probe
Hasil desain probe mutan dianalisa menggunakan menu ‘Direct entry’. Sekuens probe yang memenuhi seluruh kriteria umum akan ditampilkan dengan tanda centang (√), sedangkan sekuens probe yang tidak memenuhi kriteria akan ditampilkan dengan tanda peringatan (!). Contoh tampilan hasil analisis probe A94MG1 menggunakan program ditunjukkan pada Gambar 2.
Analisis tahap awal dilakukan berdasarkan kriteria probe secara umum yang meliputi panjang nukleotida, kandungan %GC, Tm, runs, repeats, dimer, dan hairpin. Kriteria probe secara umum yang pertama yaitu memiliki panjang nukleotida yang berkisar antara 18-30 basa dengan panjang optimal 20 basa [13]. Panjang dari probe dapat mempengaruhi spesifisitas PCR, dimana probe yang terlalu panjang dapat meningkatkan peluang terjadinya mismatch antara probe dan DNA target. Probe akan bersifat kurang spesifik untuk membedakan mismatch sehingga dapat menyebabkan hasil positif yang palsu terutama ketika perbedaan antara 2 atau lebih probe hanya sebuah nukleotida tunggal [20]. Probe yang lebih pendek dapat mengurangi spesifisitas [21] dan
membutuhkan waktu yang lebih sedikit untuk hibridisasi secara spesifik ke DNA targetnya [22]. Hasil rancangan probe mutan memiliki panjang dari 20 nukleotida sehingga memenuhi kriteria.
Sequence:
Length:
iCGATcτAcggcagcctGGT 20
Composition:
Molar absorbance:
A: 3 C: € G: 6 T: 5
33.1 μg∕ml = 1 OD2 60
©%GC Tm0C Dimers Stability GC damp Runs Hairpin Hom 60 67 OK n/a n/a OK OK-
Sequence:
Length:
Tcgatctacggcagcctggc 20
Composition:
Molar absorbance:
A: 3 C: 7 G: 6 T: 4 33.4 μg∕ml = 1 OD260
Analysis criteria: Probe
I a j ⅞GC Tm0C Dimers Stability GC damp Runs Hairpin Hom
∖~ √ 65 69 OK n/a n/a OK OK -
Gambar 2. Contoh tampilan hasil analisis pada program Clone Manager Suite 9.2.
Kriteria kedua yaitu memiliki kandungan %GC sebesar 40-60% [12,22, 23]. Nilai %GC berhubungan dengan Tm dimana semakin tinggi GC maka nilai Tm juga semakin tinggi. Hal tersebut disebabkan oleh pasangan basa G-C dihubungkan oleh ikatan hidroden rangkap 3 dan pasangan basa A-T yang dihubungkan oleh ikatan hidrogen rangkap 2 [24]. Hasil rancangan probe mutan memiliki kandungan %GC sebesar 60% sehingga masih memenuhi kriteria.
Kriteria ketiga dari probe yaitu memiliki Tm 5-10°C lebih tinggi dari Tm dari primer [17, 25]. Tm probe harus lebih besar dari Tm primer untuk memastikan ikatan yang terbentuk kuat selama fase penempelan. Tm probe bergantung dari panjang dan kandungan %GC [21]. Perbedaan nilai Tm probe dengan primer akan menjamin probe dapat terhibridisasi dengan target selama proses ekstensi primer terjadi, sehingga akan meningkatkan sinyal fluoresens pada proses amplifikasi [13]. Tm probe yang lebih tinggi memungkinkan probe tetap terhibridisasi
dengan DNA target sebelum proses pemanjangan primer [26]. Nilai Tm probe yang melebihi 80°C dapat menghambat ekstensi polimerase karena polimerase tidak dapat melepaskan probe yang telah terhibridisasi [25]. Tm dari hasil rancangan probe mutan yaitu 67°C telah sesuai dengan kriteria yaitu 5-10°C diatas Tm primer (60°C dan 61°C).
Kriteria keempat dan kelima yaitu DNA probe memiliki runs dan repeats maksimal sebanyak 4 basa. Runs yang melebihi 4 basa khususnya basa G dan C akan menyebabkan mispriming dan oligonukleotida terlipat satu sama lain sehingga membentuk struktur sekunder [13, 23]. Probe diharapkan tidak mengandung repeats karena pengulangan 2 nukleotida yang sama berturut-turut (dinukleotida) memungkinkan probe tidak dapat berhibridisasi dengan benar pada target [27]. Pengulangan dinukleotida dapat menghasilkan urutan yang komplemen dan membentuk struktur sekunder [28]. Pada hasil rancangan, probe memiliki runs sebanyak 2 dan tidak ada repeats sehingga masih memenuhi kriteria.
Kriteria selanjutnya yang perlu diperhatikan yaitu probe harus memiliki dimer sedikit. Dimer terbentuk oleh interaksi intermolekuler antara dua probe [23]. Adanya dimer pada probe dapat mengurangi efisiensi hibridisasi sehingga menimbulkan penurunan sensitivitas deteksi [29]. Pada hasil rancangan terbentuk dimer sebanyak 4 yang dapat dilihat pada Gambar 3. Dimer yang terbentuk yaitu dimers any dimana dimer berada pada daerah selain di ujung 3’. Pada program Clone Manager Suite 9.2., dimers any yang diperbolehkan yaitu maksimal 5 basa, sehingga hasil rancangan masih memenuhi kriteria. Kriteria terakhir yaitu probe tidak boleh terbentuk hairpin yang dapat menghambat hibridisasi dengan target [12,23]. Keberadaan struktur sekunder akan mempengaruhi probe dikarenakan adanya interaksi intermolekuler maupun intramolekuler pada sekuens [23]. Hasil rancangan probe
tidak mengandung hairpins dan memenuhi kriteria. Hasil analisis runs, repeats, dan hairpins ditunjukkan pada Gambar 4.
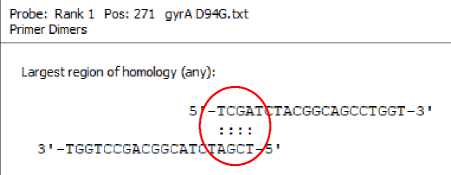
Gambar 3. Hasil analisis dimer pada program Clone Manager Suite 9.2.
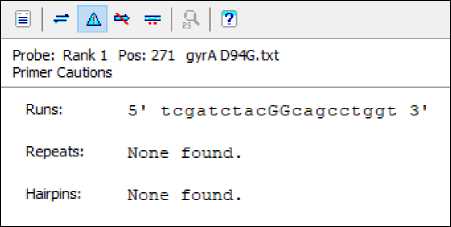
Gambar 4. Hasil analisis runs, repeats, dan hairpins pada program Clone Manager Suite 9.2.
Hasil desain probe mutan telah memenuhi kriteria probe secara umum sehingga dapat dilanjutkan dengan analisis berdasarkan kriteria pelabelan TaqMan probe. TaqMan probe merupakan probe yang memiliki pewarna reporter fluoresen yang melekat pada ujung 5’ dan pewarna quencher pada ujung 3’ [30]. Dalam RT-PCR, sebuah molekul reporter fluoresens digunakan untuk memantau saat proses PCR berlangsung. Fluoresensi yang dipancarkan oleh molekul reporter menunjukkan produk PCR dan terakumulasi pada setiap siklus amplifikasi. Jika pewarna reporter berada di dekat pewarna quencher, sinyal fluoresens yang dihasilkan oleh reporter akan diserap atau dipadamkan oleh pewarna quencher sehingga tidak ada sinyal fluoresens. Hal ini disebut sebagai fenomena FRET (Fluorescence Resonance Energy Transfer) [14]. FRET merupakan fenomena kuantum
yang terjadi antara dua molekul pewarna. Energi eksitasi ditransfer diantara kedua pewarna fluorofor melalui interaksi dipol-dipol tanpa emisi foton sehingga mengakibatkan fluoresensi reporter akan padam [31].
Kriteria pertama dari pelabelan TaqMan probe yaitu tidak boleh ada basa G pada posisi nukleotida pertama dan kedua dari ujung 5’ [27]. Basa G tidak dianjurkan berada pada ujung 5’ karena basa G dapat memadamkan fluoresens dari pewarna reporter [13,26,31]. Kemampuan basa G mengabsorbsi fluoresensi terjadi melalui transfer elektron yang diinduksi oleh cahaya [32]. Hasil desain probe A94MG1 memenuhi kriteria tersebut.
A94MG1 --► 5' - TCgATCTACGGCAGCCTGGT - 3’
Keterangan :
Il : Posisi basa G
Gambar 5. Posisi basa G pada probe A94MG1, A94MA1, dan A94MY9.
Kriteria kedua yaitu probe mengandung jumlah basa C lebih banyak atau sama dengan basa G [13]. Jumlah kandungan basa G yang lebih banyak berpengaruh pada penurunan sinyal fluoresens, sedangkan jumlah kandungan basa C yang lebih banyak akan meningkatkan fluoresens yang dihasilkan selama proses PCR. Hal tersebut berkaitan dengan sifat alami basa G yang dapat memadamkan sinyal fluoresens dari pewarna reporter [31]. Probe A94MG1 memenuhi kedua kriteria dari pelabelan TaqMan probe tersebut.
Probe A94MG1 yang telah memenuhi kriteria pelabelan TaqMan dapat ditempelkan label yang terdiri dari reporter dan quencher. Pemilihan kedua pewarna penting dalam mendesain TaqMan probe agar dapat terdeteksi pada instrumen [33]. Label yang biasanya digunakan untuk TaqMan probe adalah FAM sebagai reporter dan TAMRA sebagai quencher [29]. Label reporter FAM merupakan pewarna donor sebagai derivat fluorescein dengan emisi fluoresens kuat yang
digunakan untuk memonitoring amplikon hasil amplifikasi pada setiap siklus PCR dan dapat berikatan spesifik dengan DNA probe [17,25,29]. Penggunaan label FAM untuk reporter dianjurkan saat mendesain reaksi tunggal karena lebih murah, berkinerja baik, dan dapat dideteksi oleh semua instrumen yang ada di pasaran [33]. Label quencher TAMRA merupakan pewarna akseptor sebagai derivat rodamin yang dapat mengabsorbsi emisi dari reporter saat kedua pewarna berada dalam posisi yang berdekatan [17,25,29].
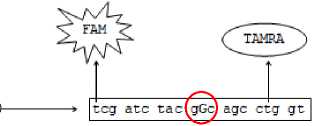
Kedua label reporter dan quencher ditempelkan pada ujung berlawanan pada urutan nukleotida probe mutan. Pelabelan pewarna direkomendasikan agar dilekatkan pada basa C atau T, tetapi umumnya dilekatkan pada basa T [26]. Penelitian Proudnikov et al. (2003) melaporkan bahwa pelabelan quencher TAMRA yang ditempatkan di posisi sentral dari oligonukleotida memberikan sensitivitas yang lebih tinggi sebanyak 30 kali lipat daripada di posisi ujung 3’ [34]. Pemasangan label pada desain probe mutan A94MG1 dapat dilihat pada Gambar 6. Label reporter ditempelkan pada basa T di ujung 5’ dan label quencher ditempelkan di basa T pada posisi basa ke 17 dari ujung 5’.
Q2)—* gac gcg teg ate tac gAc agc ctg gtg cgc atg (T)→ 89 90 91 92 93 94 95 96 97 98 99
Keterangan : 1 : Kodon gen ^rA A/ tuberculosis
2 : Nukleotida Wildtxpe gen g»rA J/ tuberculosis
3 : Nukleotida pro be mutan A 94MG1
Gambar 6. Pelabelan desain probe mutan A94MG1 yang spesifik terhadap perubahan A → G pada kodon 94.
Pada penelitian ini, hasil desain TaqMan probe dapat digunakan secara spesifik untuk mendeteksi mutasi D94G pada gen gyrA M. tuberculosis kodon 94.
Probe A94MG1 merupakan probe terpilih yang telah melewati analisis kriteria probe secara umum dan kriteria pelabelan [4]
TaqMan probe. Pada saat probe tersebut berhibridisasi dengan urutan target, probe akan mengalami hidrolisis oleh enzim Taq polimerase dan menghasilkan sinyal fluoresens yang dapat ditangkap oleh detektor. Sinyal yang ditangkap detektor sebanding dengan jumlah amplikon dari hasil amplifikasi pada reaksi PCR. Probe yang tidak terhibridisasi dengan urutan [5]
target menandakan tidak adanya mutasi pada isolat, sehingga probe tidak menghasilkan sinyal fluoresens.
Penggunaan hasil desain probe mutan dapat memberikan hasil positif palsu dengan adanya mutasi pada kodon lain. Maka dari itu, perlu dilakukan pengujian lebih lanjut secara eksperimental di laboratorium untuk membuktikan efisiensi kerja dari desain [6]
TaqMan probe A94MG1.
-
4. KESIMPULAN
TaqMan probe yaitu probe A94MG1 dengan urutan 5’–
untuk deteksi spesifik mutasi D94G pada kodon 94 gen gyrA M. tuberculosis.
-
5.
DAFTAR PUSTAKA
[8]
-
[1] World Health Organization. 2018,
Global Tuberculosis Report 2018,
Perancis: World Health Organization.
-
[2] Malik, S., Willby, M., Sikes, D.,
Tsodikov, O. V. and Posey, J. E. 2012, New Insights into
Fluoroquinolone Resistance in Mycobacterium tuberculosis:
Functional Genetic Analysis of gyrA and gyrB Mutations, Plos One, 7: 110.
-
[3] Rinanda, T. 2015, Kajian Molekuler Mekanisme Resistensi
Mycobacterium tuberculosis, Jurnal
[9]
Kedokteran Syiah Kuala, 15: 162
167.
Maruri, F., Sterlin, T. R., Kaiga, A. W., Blackman, A., Heijden, Y. F., Mayer, C., et al. 2012, A systematic review of gyrase mutations associatef with fluoroquinolone-resistant
Mycobacterium tuberculosis and a proposed gyrase numbering system, Journal of Antimicrobiology
Chemotherapy, 67: 819-831.
Disratthakit, A., Prammananan, T., Tribuddharat, C., Thaipisuttikul, I., Doi, N., Leechawengwongs, M., et al. 2016, Role of gyrB Mutations in Pre-extensively and Extensively Drug-Resistant Tuberculosis in Thai Clinical Isolates, Antimicrobial Agents and Chemotherapy, 60: 51895197.
Zhang, Z., Lu, J., Wang, Y., Pang, Y., and Zhao, Y. 2014, Prevalence and Molecular Characterization of Fluoroquinolone-Resistant Mycobacterium tuberculosis Isolates in China, Antimicrobial Agents and Chemotherapy, 58: 364-369.
Da Silva, P. E. A. and Palomino, J. C. 2011, Molecular basis and mechanisms of drug resistance in Mycobacterium tuberculosis:
classical and new drugs, Journal of Antimicrobiology Chemotherapy, 66: 1417-1430.
Li, J., Gao, X., Luo, T., Wu, J., Sun, G., Liu, G., et al. 2014, Association of gyrA/B mutations and resistance levels to fluoroquinolones in clinical isolates of Mycobacterium
tuberculosis, Emerging Microbes and Infections, 3: 1-5.
Avalos, E., Catanzaro, D., Catanzaro, A., Ganiats, T., Brodine, S., Alcaraz, J., et al. 2015, Frequency and Geographic Distribution of gyrA and gyrB Mutations Associated with Fluoroquinolone Resistance in Clinical Mycobacterium tuberculosis Isolates: A Systematic Review, Plos One, 10: 1-24.
-
[10] Yin, X. and Yu, Z. 2010, Mutation characterization of gyrA and gyrB genes in levofloxacin-resistant Mycobacterium tuberculosis clinical isolates from Guangdong Province in China, Journal of Infection, 61: 150154.
-
[11] Cui, Z., Wang, J., Lu, J., Huang, X., and Hu, Z. 2011, Association of mutation patterns in gyrA/B genes and ofloxacin resistance levels in Mycobacterium tuberculosis isolates from East China in 2009, BMC Infections Diseases, 11: 1-5.
-
[12] Walker, J. M. and Rapley, R. 2005, Medical Biomethods Handbook,
Humana Press Inc., New Jersey.
-
[13] McPherson, M. J. and Moller, S. G. 2006, PCR Second Edition, Taylor & Francis Group, New York.
-
[14] Navarro, E., Serrano-Heras, G., Castano, M. J., and Solera, J. 2015, Real-time PCR detection chemistry, Clinica Chimica Acta, 439: 231-250.
-
[15] Chou, C., Chen, C., Lee, T., and Peck, K. 2004, Optimization of probe length and the number of probes per gene for optimal microarray analysis of gene expression, Nucleic Acids Research, 32: 1-8.
-
[16] Sasmitha, L. V. 2017, Eksplorasi Mutasi Gen gyrA Sebagai Penanda Extensively Drugs Resistant
Tuberculosis pada Isolat Klinik Multidrug-Resistant Tuberculosis di Bali dengan Metode Polymerase Chain Reaction, Skripsi, Program
Studi Farmasi, Fakultas Matematika dan Ilmu Pengetahuan Alam, Universitas Udayana.
-
[17] Anonim a. 2006, Real-Time PCR Applications Guide. Bulletin 5279, Bio-Rad Laboratories Inc., USA. Available At: http://www.bio-
rad.com/webroot/web/pdf/lsr/literatur e/Bulletin_5279.pdf
-
[18] Pebriani, N. N. 2019, Desain DNA Probe secara In Silico Sebagai Pendeteksi Mutasi Gen gyrA dan gyrB Mycobacterium tuberculosis
untuk Metode Real Time Polymerase Chain Reaction, Skripsi, Program Studi Farmasi, Fakultas Matematika dan Ilmu Pengetahuan Alam, Universitas Udayana.
-
[19] Anonim b. 2001, Allelic
Discrimination Using The 5’ Nuclease Assay, Applied Biosystems, USA, Available At:
http://www.austincc.edu/mlt/mdfund/ mdfund_Unit11AllelicDiscrimination .pdf
-
[20] Alvandi, E. and Koohdani, F. 2014, Zip Nucleic Acid: A New Reliable Method To Increase The Melting Temperature of Real-Time PCR Probes, Journal of Diabetes & Metabolic Disorders, 13: 1-4.
-
[21] Rodriguez, A., Rodriguez, M., Cordoba, J. J., and Andrade, M. J. 2015, ‘Design of Primers and Probes for Quantitative Real-Time PCR Methods’ in Basu, C., PCR Primer Design, 2nd ed, Humana Press, New York, pp. 31-53.
-
[22] Narayanan, S. 1992, Overview of Principles and Current uses of DNA Probes in Clinical and Laboratory Medicine, Annals of Clinical and Laboratory Science, 22: 353-376.
-
[23] Borah, P. 2011, Primer Designing for PCR, Science Vision, 11: 134-136.
-
[24] Patel, N. K., and Prakash, N. 2013, Principle and Tools For Primer Design, Atmiya Spandan Biological Sciences, 1: 79-95.
-
[25] Meuer, S. C., Wittwer, and Nakagawara, K. 2001, Rapid Cycle Real-Time PCR: Method and
Applications, Springer, Berlin.
-
[26] Bishop, J. L., Campbell, S. A., Farrell, P., Fitzgerald, M., Haugen, M., Kocmond, W., Madden, D. E., Murray, W. E., and Persing, D. H. 2015, Designing Real Time Assays on the SmartCycler® II System, Cepheid Technical Support, United States, pp 1-8, Available At: http://www.cepheid.com/en/compone
nt/phocadownload/category/5-support?download=87:smart-note-6-1
-
[27] Rychlik, W. 2010, OLIGO Primer Analysis Software Version 7,
Molecular Biology Insights, Inc., USA.
-
[28] Yuwono, T. 2008, Biologi Molekuler, Penerbit Erlangga, Jakarta. pp. 49-74.
-
[29] Dorak, M. T. 2007, Real-Time PCR, Taylor and Francis Group, New York.
-
[30] Arya, M., Shergill, I. S., Williamson, M., Gommersall, L., Arya, N., and Patel, H. R. H. 2005, Basic principles of real-time quantitative PCR, Experts Rev. Mol. Diagn., 5: 209219.
-
[31] Rodriguez-Lazaro, D. and
Hernandez, M. 2013, Real-Time PCR in Food Science: Introduction, Curr. Issues Mol. Biol., 15: 25-38.
-
[32] Murray, J. L., Hu, P., and Shafer, D. A. 2014, Seven Novel Probe Systems for Real-Time PCR Provide Absolute Single-Base Discrimination, Higher Signaling, and Generic Components, The Journal of Molecular Diagnostics, 16: 627-628.
-
[33] Seifi, M., Ghasemi, A., Heidarzadeh, S., Khosravi, M., Namipashaki, A., Soofiany, V. M., et al. 2012,
‘Overiew of Real-Time PCR Principles’ in Hernandez-Rodriguez, P., Polymerase Chain Reaction, InTechOpen, London.
-
[34] Proudnikov, D., Yuferov, V., Zhou, Y., LaForge, K. S., Ho, A., and Kreek, M. J. 2003, Optimizing
primer-probe design for fluorescent PCR, Journal of Neuroscience
Methods, 123: 31-45.
121

Discussion and feedback