ANALISIS REDUKSI DIMENSI PADA KLASIFIKASI MICROARRAY MENGGUNAKAN MBP POWELL BEALE
on
E-Jurnal Matematika, Vol. 7 (1), Januari 2018, pp.17-24
DOI: https://doi.org/10.24843/MTK.2018.v07.i01.p179
ISSN: 2303-1751
ANALISIS REDUKSI DIMENSI PADA KLASIFIKASI MICROARRAY MENGGUNAKAN MBP POWELL BEALE
Andi Futri Hafsah Munzir1,∗, Adiwijaya2, Annisa Aditsania3
1Ilmu Komputasi, Fakultas Informatika – Universitas Telkom [Email: andifutri@student.telkomuniversity.ac.id] 2lmu Komputasi, Fakultas Informatika – Universitas Telkom [Email: adiwijaya@telkomuniversity.ac.id] 3lmu Komputasi, Fakultas Informatika – Universitas Telkom [Email: aaditsania@telkomuniversity.ac.id] ∗ Corresponding Author
ABSTRACT
Cancer is the second leading cause of death in the world based on World Health Organization (WHO) survey in 2015. It took DNA microarray technology to analyze and diagnose cancer. DNA microarray has large dimensions so it influences the process of cancer ‘s classification. GA and PCA are used as reduction method and MBP Powell Beale as classification method. The testing of MBP classification without dimension reduction results 70, 59% - 100% accuracy. MBP+PCA results 76, 47% - 100% accuracy. MBP + GA results 76, 47% - 92, 31% accuracy.
Keywords: cancer, microarray, ga, pca, mbp.
Kanker merupakan salah satu penyebab kematian di negara berkembang. Berdasarkan survei yang dilakukan WHO (2012), kanker dianggap sebagai penyebab utama kematian di dunia. Sekitar 8, 2 juta orang meninggal karena kanker dan jumlah ini diperkirakan akan meningkat setiap tahun karena gaya hidup yang tidak sehat. Kanker adalah penyakit akibat pertumbuhan tidak normal dari sel-sel jaringan tubuh yang berubah menjadi sel kanker.
Dalam beberapa dekade terakhir, microarray data memegang peran penting dalam mendiagnosis kanker. Microarray dapat digunakan untuk menganalisis ribuan gen secara bersamaan. DNA mengandung sifat dan informasi suatu makhluk hidup. Diagnosis kanker hasil olah data microarray sering dibandingkan dengan teknik diagnosis tradisional, seperti yang telah dijelaskan Van’t Veer et al. (2002).
Dalam penelitian ini dibangun suatu sistem yang dapat menganalisis dan memprediksi se-
orang pasien apakah mengidap kanker atau tidak berdasarkan data microarray pasien. Dimensi data microarray sangatlah besar. Oleh karena itu, diperlukan proses reduksi dimensi. Reduksi dimensi data merupakan suatu teknik yang dilakukan untuk mengurangi ukuran data yang sangat besar karena dimensi pengamatan yang sangat besar menjadi menjadi kendala dalam perhitungan komputasi sehingga solusi yang dihasilkan tidak stabil seperti yang telah dijelaskan Næs et al. (2002). Nurfalah (2016) telah berhasil menggunakan perpaduan analisis komponen utama dan algoritma conjugate gradient pada backpro-pogation untuk menghasilkan akurasi sekitar 76% - 97%.
Penelitian ini membandingkan performansi sistem klasifikasi data microarray menggunakan klasifier MBP Powell Beale yang telah melalui proses reduksi dimensi, baik seleksi fitur maupun ekstraksi fitur. PCA dipilih sebagai metode ekstraksi fitur, sedangkan GA dipilih sebagai metode seleksi fitur pada penelitian ini.
PCA merupakan teknik untuk mereduksi dimensi, PCA menghasilkan sekumpulan data berdimensi baru yang biasa dinamakan Principal Component (PC). PC merupakan kombinasi linier data actual berdimensi rendah seperti yang telah dijelaskan Nurfalah (2016).
Pada penelitian ini, pemilihan jumlah vektor eigen yang akan digunakan berdasarkan proporsi kumulatif varians (nilai eigen). Proporsi varians menunjukkan besarnya persentase informasi variabel-variabel asal yang terkandung dalam setiap vektor ciri (vektor eigen) berdasarkan nilai eigen dan memberikan interpretasi mengenai seberapa besar data dapat terwakili dalam dimensi yang telah direduksi yang telah dijelaskan Maharani et al. (2014).
Tahapan untuk melakukan reduksi dimensi dengan menggunakan PCA berdasarkan Nur-falah (2016), yaitu:
-
1. Data diatur sebagai satu set |n| data vektor A 1...A |n|, dimana matriks input untuk PCA adalah A(i,j) untuk proses training, dengan i merupakan baris dan j merupakan kolom.
-
2. Hitung mean (A¯) dari data:
1n
A = - ∑ Am (1)
nm=1
dengan :
n=jumlah sampel, A=data observasi.
-
3. Menghitung matriks kovariansi (C):
C (A) = τ(Am - A )(Am - A) T (2)
n- 1
dengan :
n=jumlah sampel, A=data observasi, A¯=mean data.
-
4. Menghitung nilai eigen dan vektor eigen:
C (A ) Vm = λ Vm
|C(A) - λ11Vm = 0 ()
dengan :
vm = vektor eigen,
λ = nilai eigen,
C (A) = matriks kovariansi dari A, m = jumlah atribut
-
5. Ekstrak nilai diagonal dari nilai eigen dan urutkan nilai diagonal tersebut secara descending.
Ada beberapa cara dalam menentukan jumlah vektor eigen yang akan dipilih berdasarkan nilai eigen, diantaranya:
-
(a) menggunakan scree plot. Banyaknya jumlah vektor eigen yang dipilih adalah berdasarkan pada titik kurva yang tidak lagi menurun tajam atau mulai melandai.
-
(b) menggunakan proporsi kumulatif varians (nilai eigen) terhadap total varians (nilai eigen).
Adapun Perhitungan Proporsi Varians (PPV) untuk setiap komponen utama (vektor eigen) adalah sebagai berikut:
kλ
PPV = ∑≡0√ x 100% (4)
∑ i=0 λ i
dengan :
λ = nilai eigen,
m = jumlah atribut data awal, k = jumlah atribut yang akan dipilih.
-
6. Principal Component (PC) dipilih dari k kolom eigen vector yang memiliki nilai diagonal tertinggi.
-
7. Kemudian didapat data baru dengan mengalikan PC dengan data.
data baru = PC x data (5)
-
2.2 Seleksi Fitur Menggunakan GA
Genetic Algorithm adalah cabang dari algoritma evolusi yang merupakan metode adaptive yang biasa digunakan untuk memecahkan suatu pencarian nilai dalam sebuah masalah optimasi. Berdasarkan Suyanto (2008) komponen utama Genetic Algorithm, yaitu:
-
1. Teknik Pengkodean
Teknik pengkodean adalah bagaimana mengkodekan gen dari kromosom. Satu gen biasanya akan mewakili satu variabel. Gen dipresentasikan dalam bentuk bit.
-
2. Membangkitkan Populasi Awal
Membangkitkan populasi awal adalah proses membangkitkan sejumlah individu secara acak atau melalui prosedur tertentu. Teknik yang digunakan dalam pembangkitan populasi awal ini yaitu random generator.
-
3. Evaluasi Fitness
Dalam penelitian ini, fungsi Fitness didapatkan dari nilai akurasi dari klasifikasi backpropagation termodifikasi dengan algoritma MBP.
Fitness (x) = Akurasi (x) (6)
s TP + TN Akurasi (x) =---------------------
v 7 TP + FN + FP + TN
(7)
TP (True Positive) adalah proporsi positif dalam data set yang diklasifikasikan positif, FP (False Positive) adalah proporsi negatif dalam data set yang diklasifikasikan positif, TN (True Negative) adalah proporsi negatif dalam data set yang diklasifikasikan negatif, dan FN (False Negative) proporsi negatif dalam data set yang diklasifikasikan negatif. Data positif merupakan data yang ter-kategorikan kanker dan data negatif merupakan data yang terkategorikan bukan kanker.
-
4. Seleksi Orang Tua
Seleksi orang tua dilakukan dengan memilih dua individu secara proporsional berdasarkan nilai fitness yang dihasilkan. Metode yang digunakan yaitu roulettewheel.
-
5. Crossover
Crossover adalah operator dari algoritma genetika yang melibatkan dua orang tua untuk membentuk individu baru berdasarkan titik Crossover yang ditentukan secara acak. Penelitian ini menggunakan Crossover jenis single point crossover.
-
6. Mutasi
Mutasi diperlukan untuk mengembalikan informasi bit yang hilang akibat Crossover, namun dengan probabilitas mutasi (Pm) yang sangat kecil. Sebab jika mutasi terlalu sering dilakukan maka akan menghasilkan individu yang lemah karena konjugusi gen pada individu yang unggul akan dirusak.
-
7. Seleksi Survivor
Seleksi Survivor menghasilkan dampak yang signifikan terhadap performansi dan kemampuan GA untuk menemukan solusi dalam ruang pencarian yang sangat besar. Seleksi Survivor atau pergantian populasi dilakukan setelah melewati crossover dan mutasi.
Powell Beale
Metode yang digunakan untuk mendeteksi kanker pada microarray adalah modified back-propagation (MBP) conjugate gradient Powell Beale. Arsitektur yang digunakan adalah multi layer perceptron dan fungsi ak-tivasi yang digunakan yaitu sigmoid biner.
Backpropagation adalah sebuah metode sistematik untuk pelatihan multi layer jaringan syaraf tiruan (MLP-ANN). Conjugate Gradient (CG) merupakan algoritma pencarian yang arah pencariannya tidak selalu menurun tapi berdasarkan pada arah konjungsinya
yang telah dijelaskan Nurfalah (2016) Adiwi-jaya et al. (2013). Secara umum algoritma ini lebih cepat konvergen daripada metode penurunan tercepat standar backpropagation Nur-falah (2016) Adiwijaya et al. (2014).
Hasil keluaran pada proses ini adalah bobot-bobot terbaik dari arsitektur selama proses pelatihan. Adapun tahapan propagasi maju dan propagasi mundur yaitu, data yang telah melalui tahapan preprocessing dimasuk-an kedalam arsitektur JST. Tahap propagasi disini merupakan salah satu proses untuk mengeluarkan bobot yang optimum. Nilai error yang diperolah digunakan sebagai parameter dalam propagasi mundur. Tahap pelatihan akan berhenti jika MSE < minimum MSE. Error tersebut dipropagasikan balik untuk memperbaiki bobot-bobot dari semua neuron pada hidden layer dan output layer. Selanjutnya menghitung gradient di output layer dan dihidden layer. Kemudian menghitung parameter β pada semua output layer dan hidden layer. Lalu menghitung direction pada iterasi saat ini dan Teknik line search digunakan sebagai proses pencarian parameter α yang bertujuan untuk meminimumkan kinerja selama arah pencarian, karena parameter α merepresentasikan besar langkah yang diambil untuk setiap direction. Adapun Algoritma pelatihan yang digunakan dapat dilihat pada paper Nurfalah (2016).
Penelitian ini bertujuan untuk mendiagnosis kanker berdasarkan microarray data menggunakan perpaduan analisis komponen utama PCA, genetic algorithm (GA) dan metode Conjugate Gradient Propagation (MBP) Powell Beale.
Berdasarkan Gambar 1 untuk menghasilkan akurasi dari sistem ini akan melakukan tahap normalisasi data agar data dapat diproses. Setelah itu data yang telah dinormalisasi akan dibagi menjadi dua data yaitu data Training dan data Testing. Selanjutnya data Training dan Testing akan digunakan untuk reduksi dimensi menggunakan ekstraksi fitur PCA dan seleksi fitur GA kemudian di-
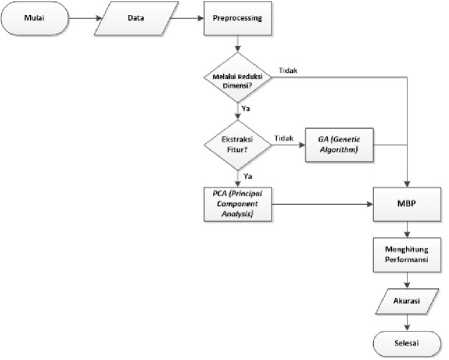
Gambar 1. Flowchart Umum Sistem
lakukan pelatihan modified backpropagation (MBP) Powell Beale. Hasil dari tahapan ini akan menghasilkan bobot terbaik. Kemudian akan dilakukan pengujian sistem dengan menggunakan data Testing untuk mengetahui hasil akurasi dari sistem.
Data set yang digunakan dalam pengerjaan penelitian ini berupa data microarray, terdiri dari tiga dataset DNA microarray yaitu colon cancer, lung cancer, ovarian cancer. Data set tersebut diperoleh dari Ridge Bio-medical Data Set Repository WHO (2012).
Tabel 1. Dataset Microarray
|
Data |
Atribute |
Record |
Train |
Test |
|
Colon Cancer |
2000 |
62 |
43 |
19 |
|
Ovarian Cancer |
15154 |
253 |
177 |
76 |
|
Lung Cancer |
12533 |
181 |
127 |
54 |
Proses pengujian menggunakan beberapa skenario klasifikasi diantaranya itu MBP, MBP+PCA, dan MBP+GA. Setiap skema klasifikasi dilakukan percobaan masing-masing sebanyak 9 kali. Pengulangan ini dilakukan karena adanya proses random number saat awal proses MBP Powell Beale.
Pada proses pengujian, proporsi data
yang digunakan sebesar 70% untuk data training dan 30% untuk data testing.
-
4.1 Pengaruh Arsitektur Jaringan Terhadap Performansi Sistem
Pada proses pengujian ini dilakukan perbandingan akurasi hasil klasifikasi MBP Powell Beale menggunakan berbagai jumlah hidden neuron. Perbandingan dilakukan menggunakan 2, 3, dan 4 Neuron. Hal ini dilakukan untuk mengetahui jumlah Hidden Neuron yang optimal untuk model sistem klasifikasi modified backpropagation (MBP) Conjugate Gradient Powell Beale.
Tabel 2. Hasil Perbandingan Akurasi Berdasarkan Neuron Hidden Layer
|
Neuron Hidden Layer |
Ovarian Cancer (%) |
Lung Cancer (%) |
Colon Cancer (%) |
Rata-Rata m |
|
2 |
97,29 |
IOO |
70,59 |
89,29 |
|
3 |
98,65 |
100 |
70,59 |
89,75 |
|
4 |
98,64 |
100 |
70,59 |
89,74 |
Berdasarkan Tabel 2 hasil perbandingan neuron hidden layer di atas didapatkan jumlah neuron terbaik pada hidden layer sebesar 3 neuron dengan akurasi rata-rata sebesar 89, 75%. Oleh karena itu, arsitektur jaringan tersebut akan digunakan sebagai arsitektur klasifikasi di skema selanjutnya.
-
4.2 Pengaruh Ekstraksi Fitur Terhadap Performansi Sistem
Pada penelitian ini, adapun pemilihan jumlah vektor eigen yang akan digunakan yaitu dengan menggunakan proporsi kumulatif varians (nilai eigen) sesuai dengan Næs et al. (2002). Adapun Perhitungan Proporsi Varians (PPV) untuk setiap komponen utama (vektor eigen) adalah menggunakan persamaan 4.
Tabel 3 menyajikan jumlah atribut masing-masing dataset kanker berdasarkan nilai threshold PPV.
Tabel 3. Jumlah Atribut berdasarkan Nilai
Threshold PPV
|
Data |
PC 80% |
PC 90% |
PC 100% |
|
Colon Cancer |
7 |
14 |
44 |
|
Ovarian Cancer |
5 |
12 |
178 |
|
Lung Cancer |
53 |
80 |
128 |
Tabel 4. Hasil Pengujian Skema Klasifikasi MBP dengan PCA
|
Data |
Threshold (%) |
Akurasi (%) |
Training Time(s) |
|
Colon |
80 |
76,47 |
6,37 |
|
Cancer |
90 |
76,47 |
6,75 |
|
100 |
76,47 |
7,37 | |
|
Ovarian |
80 |
95,94 |
7,08 |
|
Cancer |
90 |
95,95 |
7,27 |
|
100 |
100 |
7,36 | |
|
Lung |
80 |
94,23 |
5,77 |
|
Cancer |
90 |
98,08 |
5,83 |
|
100 |
100 |
5,93 |
Tabel 4 menunjukkan bahwa menggunakan batas threshold 100% pada PPV menghasilkan akurasi tertinggi bila dibandingkan dengan batas threshold lainnya.
-
4.3 Pengaruh Seleksi Fitur Terhadap Performansi Sistem
Pada sub bab ini, pengaruh jumlah atribut dari hasil seleksi fitur akan dikaji. Terdapat 2 skenario pengujian, yaitu:
-
1. Jumlah atribut yang digunakan sesuai dengan jumlah atribut hasil PPV yaitu individu yang akan digunakan untuk data colon cancer sebanyak 7,14 dan 44. Untuk data lung cancer yaitu 53, 80, dan
128. Untuk data ovarian cancer yaitu 5, 12, dan 178.
Tabel 6. Hasil Pengaruh Jumlah Atribut Terhadap Nilai Akurasi
-
2. Jumlah atribut yang digunakan sesuai dengan persentase proporsi masing-masing data awal (50%, 25% dan 10%).
Ukuran populasi merupakan salah satu parameter yang penting dalam penelitian ini, sehingga ukuran populasi yang telah ditentukan akan dilihat pengaruhnya terhadap kinerja klasifikasi.
Tabel 5. Pengaruh Ukuran Populasi Terhadap Nilai Akurasi
|
Data |
Ukuran Populasi |
Max Gen |
Jumlah Individu |
Akurasi (%) |
Training Time (s) |
|
Colon Cancer |
50 |
10 |
7 |
64,71 |
58 |
|
14 |
64,71 |
58 | |||
|
44 |
35,29 |
58 | |||
|
IOO |
5 |
7 |
64,71 |
58 | |
|
14 |
64,71 |
58 | |||
|
44 |
64,71 |
58 | |||
|
Ovarian Cancer |
50 |
10 |
5 |
64,86 |
76 |
|
12 |
64,86 |
7 5 | |||
|
178 |
64,86 |
79 | |||
|
IOO |
5 |
5 |
64,86 |
77 | |
|
12 |
64,86 |
77 | |||
|
178 |
62,16 |
81 | |||
|
Lung Cancer |
50 |
10 |
53 |
80,77 |
77 |
|
80 |
84,62 |
75 | |||
|
128 |
84,62 |
77 | |||
|
100 |
5 |
53 |
84,62 |
82 | |
|
80 |
73,08 |
74 | |||
|
128 |
84,62 |
79 |
Berdasarkan Tabel 5 dapat diamati untuk proporsi ukuran populasi yang mengikuti proporsi PCA mampu menghasilkan nilai akurasi untuk colon cancer 64, 71% dengan training time 58 detik, ovarian cancer 64, 86% dengan training time 75 detik, dan lung cancer 84, 62% dengan training time 75 detik.
Berdasarkan Tabel 6 dapat diamati bahwa untuk proporsi data berdasarkan jumlah atribut mampu memberikan hasil untuk colon cancer 64, 71% dengan training time 60 detik, ovarian cancer 64, 86% dengan training time 133 detik, dan lung cancer 84, 62% dengan training time 98 detik.
Berdasarkan proporsi data yang digunakan dapat diamati bahwa seleksi fitur GA+MBP mampu memprediksi dengan baik pada jenis data lung cancer yaitu sebesar 84, 62%. Hal
|
Data |
Ukuran Populasi |
IVlax Gen |
Jumlah Individu |
Akurasi (%) |
Training Time (s) |
|
Colon Cancer |
50 |
IO |
IOOO |
58,82 |
71 |
|
500 |
58,82 |
64 | |||
|
200 |
58,82 |
60 | |||
|
IOO |
5 |
IOOO |
64,71 |
72 | |
|
500 |
64,71 |
67 | |||
|
200 |
64,71 |
60 | |||
|
Ovarian Cancer |
50 |
IO |
75 77 |
64,86 |
222 |
|
3788 |
64,86 |
149 | |||
|
1515 |
64,86 |
202 | |||
|
IOO |
5 |
7577 |
64,86 |
218 | |
|
3788 |
64,86 |
153 | |||
|
1515 |
64,86 |
133 | |||
|
Lung Cancer |
50 |
IO |
5266 |
76,92 |
211 |
|
3133 |
84,62 |
135 | |||
|
1253 |
84,62 |
98 | |||
|
IOO |
5 |
6266 |
84,62 |
195 | |
|
3133 |
84,62 |
137 | |||
|
1253 |
84,62 |
108 |
ini disebabkan karena pemilihan jumlah individu yang digunakan akan memberikan hasil performansi yang optimum.
-
4.4 Perbandingan Performansi dengan atau tanpa Proses Reduksi Dimensi
Berdasarkan hasil pengujian yang telah didapatkan, maka didapatkan akurasi untuk setiap jenis data.
Tabel 7. Perbandingan Akurasi Berdasarkan Skema Klasifikasi
|
Data |
Skema Klasifikasi |
Akurasi(%) |
|
Colon |
MBP |
76,47 |
|
Cancer |
MBP+PCA |
76,47 |
|
MBP+GA |
76,47 | |
|
Ovarian |
MBP |
100 |
|
Cancer |
MBP+PCA |
100 |
|
MBP+GA |
81,03 | |
|
Lung |
MBP |
100 |
|
Cancer |
MBP+PCA |
100 |
|
MBP+GA |
92,31 |
Berdasarkan skema pengujian pada subbab sebelumnya, akurasi untuk setiap jenis data microarray menggunakan skema klasifikasi MBP menghasilkan akurasi 70, 59% -100%, skema klasifikasi MBP+PCA menghasilkan akurasi 76, 47% - 100%, dan ske-
ma klasifikasi MBP+GA menghasilkan akurasi 35, 26% - 84, 62%. Hal ini disebabkan karena pengaruh pengambilan vektor eigen (PPV) pada MBP+PCA serta pengaruh ukuran populasi dan max gen pada MBP+GA yang mempengaruhi klasifikasi pelatihan dan pengujian.
Pada modified backpropagation (MBP) Powell Beale tanpa menggunakan reduksi dimensi maupun menggunakan reduksi dimensi menghasilkan akurasi lebih dari 70%. Tetapi training time pada MBP 20 kali lipat dari training time MBP+PCA sehingga diperlukan skema yang didalamnya terdapat proses reduksi dimensi dan proses klasifikasi. Dari hasil pengujian, skema klasifikasi MBP+PCA menghasilkan rata-rata akurasi sebesar 92, 15% dengan training time 2 - 4 detik untuk pelatihan masing-masing data sedangkan untuk skema klasifikasi MBP+GA menghasilkan rata-rata akurasi sebesar 70% yang membuthkan waktu training time 58 -222 detik. Hal ini disebabkan karena seleksi fitur GA memilih atribut yang nilai fitness-nya paling besar dan prinsipnya menggunakan korelasi padahal tidak semua aribut berkorelasi, sedangkan pada ekstraksi fitur PCA menggunakan semua atribut dan membangun dimensi baru dari kombinasi linier dimensi asli.
Pada penelitian ini penulis merekomendasikan skema klasifikasi menggunakan MBP+PCA karena pada skema klasfikasi MBP+PCA menghasilkan akurasi tertinggi yaitu pada jenis data ovarian cancer dan lung cancer menghasilkan akurasi sebesar 100%, sedangkan pada jenis data colon cancer menghasilkan akurasi sebesar 76, 47% dengan waktu training time 2 detik.
Skema klasifikasi MBP tanpa reduksi dimensi menghasilkan akurasi 70, 59% -100%, skema klasifikasi MBP+PCA menghasilkan akurasi 76, 47% - 100%, dan skema klasifikasi MBP+GA menghasilkan akurasi 35, 26% - 84, 62%. Hal ini disebabkan karena pengaruh pengambilan vektor ei-
gen (PPV) pada MBP+PCA serta pengaruh ukuran populasi dan max gen pada MBP+GA yang mempengaruhi klasifikasi pelatihan dan pengujian. Penulis menggunakan PPV 80%, 90% dan 100% karena dari hasil pengujian yang telah dilakukan perbandingan jumlah PC yang terpilih menghasilkan akurasi yang tidak jauh beda seperti pada data ovarian cancer jumlah PC yang terpilih untuk threshold 90% yaitu 12 menghasilkan akurasi 95, 95% sedangkan jumlah pc yang terpilih untuk threshold 100% yaitu 178 menghasilkan akurasi 100%.
MBP+PCA menghasilkan akurasi tertinggi yaitu pada jenis data ovarian cancer dan lung cancer menghasilkan akurasi sebesar 100% dan pada jenis data colon cancer menghasilkan akurasi sebesar 76, 47% dengan waktu training time 2 detik.
Daftar Pustaka
Adiwijaya, U., Wirayuda, T., Baizal, Z., and Haryoko, U. (2013). An improvement of backpropagation performance by using conjugate gradient on forecasting of air temperature and humidity in indonesia. Far East J. Math. Sci, 1:57–67.
Adiwijaya, Wisesty, U., Nhita, F., et al. (2014). Study of line search techniques on the modified backpropagation for forecasting of weather data in indonesia. Far East Journal of Mathematical Sciences, 86(2):139.
Maharani, Adiwijaya, W., Gozali, A. A., et al. (2014). Degree centrality and eigenvector centrality in twitter. In Telecommunication Systems Services and Applications (TSSA), 2014 8th International Conference on, pages 1–5. IEEE.
Næs, T., Isaksson, T., Fearn, T., and Davies, T. (2002). A user friendly guide to multivariate calibration and classification. NIR publications.
Nurfalah, Adiwijaya, S. A. A. (2016). Cancer detection based on microarray data classification using pca and modified back pro-
pagation. Far East Journal of Electronics and Communications, 16(2):269.
Suyanto, S. C. (2008). Membangun mesin ber-iq tinggi. Bandung: Informatika.
Van’t Veer, L. J., Dai, H., Van De Vijver, M. J., He, Y. D., Hart, A. A., Mao, M., Peterse, H. L., Van Der Kooy, K., Marton, M. J., Witteveen, A. T., et al. (2002). Gene expression profiling predicts clinical outcome of breast cancer. nature,
415(6871):530–536.
WHO (2012). World health day 2012: ageing and health: toolkit for event organizers.
24
Discussion and feedback