Analisis Filogenetik Ikan Tuna (Thunnus spp.) yang didaratkan di Pelabuhan Benoa, Bali
on
JMRT, Volume 4 No 2 Tahun 2021, Halaman: 37-41
JMRT
JOURNAL OF MARINE RESEARCH AND TECHNOLOGY
journal homepage: https://ojs.unud.ac.id/index.php/JMRT
ISSN: 2621-0096 (electronic); 2621-0088 (print)
Analisis Filogenetik Ikan Tuna (Thunnus spp.) yang didaratkan di Pelabuhan Benoa, Bali
Paul Helga Fernandeza, IGB Sila Dharma*a, I Nyoman Giri Putraa, Andrianus Sembiring b, Astria
Yusmalindab, Danie Al Malik b, Putu Dian Pertiwi b
aProgram Studi Ilmu Kelautan, Fakultas Kelautan dan Perikanan, Universitas Udayana, Bali, Indonesia
bYayasan Biodiversitas Indonesia (Bionesia) Denpasar, Bali, Indonesia
*Corresponding author, email: siladharma@unud.ac.id
ARTICLE INFO
ABSTRACT
Article history:
Received: August 25th 2020
Received in revised form: December 24th 2020
Accepted: February 2th 2021
Available online: August 31th 2021
Keywords:
Molecular genetic Tuna
Benoa
Phylogenetic
Tuna is one of the largest fisheries commodities in Indonesia after shrimp and demersal fish. The genus Thunnus is a type of tuna that dominates the international market. The genus Thunnus consisted of seven species of tuna. In some cases, the same morphological character has caused misidentification and data collection on tuna species. Therefore, this study aimed to identify tuna species that are landed at Benoa Harbor and analyzed their phylogenetic relationships. Species identification and phylogenetic analysed in this study used the mtDNA control region locus. The results of this study indicated that there are five tuna species landed at Benoa Harbor, namely yellowfin tuna (T. albacares), longtail tuna (T. tonggol), bigeye tuna (T. obesus), southern bluefin tuna (T. maccoyii), and albacore tuna (T. alalunga). Based on phylogenetic tree reconstruction, all samples were divided into five according to the number of tuna species resulted from molecular identification. Reconstruction of phylogenetic trees is supported by genetic distance between clades has a value of 0.075 - 0.212, with the closest kinship found in yellowfin tuna (T. albacares) with bigeye tuna (T. obesus) and the farthest found in yelowfin tuna (T. albacares) with albacore tuna (T. alalunga).
2021 JMRT. All rights reserved.
Tuna termasuk dalam perdaganan perikanan yang paling besar di Indonesia (Habibi et al., 2011). Tuna termasuk dalam Famili Scombridae dan dibagi menjadi empat genus, yaitu Euthynnus, Thunnus, Auxis, dan Katsuwonus (Majkowski, 2007). Genus Thunnus adalah jenis tuna yang mendominasi di pasar internasional. Spesies tuna dari Thunnus yang sering mendominasi dalam pasar internasional yaitu tuna mata besar (T. obesus), tuna sirip biru pasifik (T. orientalis), tuna sirip biru atlantik (T. Thynnus), tuna albacore (T. alalunga), tuna sirip kuning (T. albacares) dan tuna sirip biru selatan (T. maccoyii) (Majkowski, 2007).
Setiap spesies dari Thunnus mempunyai ciri morfologinya masing-masing sehingga dapat mempermudah untuk melakukan identifikasi. Beberapa spesies seperti tuna mata besar (T. obesus) dan tuna sirip kuning (T. albacares) yang panjangnya tidak lebih dari 40 cm mempunyai mirip sehingga bisa menyebabkan kekeliruan dalam identifikasi serta estimasi dari hasil tangkapan (Gerasmio, 2012). Maka dari itu diperlukan kajian molekuler dengan teknik deoxyribonucleic acid (DNA) barcoding yang berguna dalam pengelolaan tuna.
DNA barcoding adalah sebuah teknik yang digunakan untuk mengidentifikasi organisme dengan menggunakan sekuens gen tertentu yang sudah terbukti kemampuannya untuk menemukan perbedaan tingkat spesies.
Tidak seperti dengan teknik identifikasi pada umumnya, hanya bisa dilakukan pada organisme yang dewasa serta utuh. DNA barcoding bisa digunakan untuk identifikasi segala macam organisme mulai dari telur hingga dewasa serta bisa juga dilakukan pada potongan tubuh organisme. (Zein dan Prawiradilaga, 2013).
Dalam teknik DNA barcoding, control region (CR) mitokondria telah divalidasi sebagai penanda molekuler untuk membedakan spesies Thunnus dan merupakan penanda yang lebih kuat, daripada penanda standar DNA barcoding yaitu CO1 mitokondria, karena CO1 mitokondria tidak dapat membedakan semua spesies Thunnus termasuk yang dalam subgenus NeoThunnus (Thunnus albacares, T. atlanticus, dan T. tonggol) yang sangat terkait erat (Vinãs and Tudela, 2009). Filogenetik adalah suatu teknik untuk memodelkan dan melihat kedekatan atau kekerabatan suatu spesies dengan spesies lainnya serta memprediksi perbedaan dari satu nenek moyang terhadap keturunannya (Li et al., 1999).
Identifikasi tuna dengan menggunakan DNA barcoding perlu dilakukan karena dalam penanganan dan penyimpanan tangkapan diatas kapal, kondisi tangkapan tuna sudah dalam keadaan beku dan sirip sudah dipotong yang dapat mengakibatkan kesalahan dalam identifikasi spesies tangkapan serta estimasi hasi tangkapan. Penangan dan penyimpanan
tangkapan seperti itu dilakukan di beberapa perusahaan tuna yang ada di Pelabuhan Benoa, Bali.
Pelabuhan Benoa adalah salah satu pelabuhan di Indonesia yang menjadi lokasi pendaratan tuna. Industri perikanan tuna yang ada di Pelabuhan Benoa yaitu mulai dari agen perusahaan penangkapan, eksportir, pengolahan serta jassa coldstorage (Miazwir, 2012). Oleh karena itu penelitian ini akan mengidentifikasi spesies Thunnus apa saja yang didaratkan di Pelabuhan Benoa, Bali dan mencari hubungan kekerabatan (filogenetik) antar spesiesnya.
Pengambilan sampel ikan tuna (Thunnus spp.) dilakukan secara langsung di dua perusahaan tuna di Pelabuhan Benoa, Bali yaitu Bali Tuna Segar dan Jaya Kota (Gambar 1). Pengambilan sampel hanya dilakukan sekali di setiap perusahaan dan dilakukan saat perusahaan tersebut sedang melakukan pemindahan hasil tangkapan dari kapal ke tempat penyimpanan beku. Tuna yang akan dijadikan sampel diidentifikasi secara morfologi terlebih dahulu. Sampel yang diambil adalah bagian sirip dan dipotong berukuran ± 5 cm. Sirip dipilih karena bagian tubuh tersebut merupakan bagian yang lebih yang lebih mudah dipotong dibandingkan tubuh lainnya karena tuna yang didapatkan sudah dalam keadaan beku. Setiap sampel sirip yang telah didapatkan kemudian dimasukkan kedalam microtube 1,6 ml yang telah diisi dengan larutan etanol 96%. Penggunaan bahan etalo ini dimaksudkan untuk mencegah rusaknya komposisi DNA pada sampel. Selanjutnya sampel diberi label pada masing-masing tube.
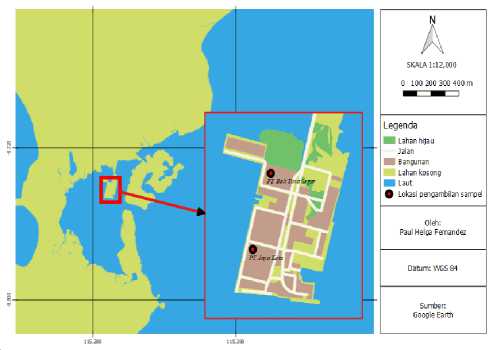
Gambar 1. Peta penelitian lokasi pengambilan sampel
-
2.2 . Analisis Molekuler
-
2.2.1 Ekstraksi DNA
-
Ekstraksi DNA dilakukan dengan menggunakan larutan Chelex 10% (Walsh et al., 1991). Jaringan sampel diambil sampel diambil ± 2 mm dan dimasukkan kedalam tube berisi larutan chelex 10%, kemudian divortex selama ± 15 detik dan centrifuge selama ± 1 menit. Setelah itu, sampel dipanaskan didalam heating block dengan suhu 95°C selama 60 menit. Walsh et al (1991) menyebutkan bahwa metode ini banyak digunakan karena praktis, lebih cepat, dan tidak melibatkan pelarut organik serta tidak memerlukan banyak transfer tube untuk prosesnya sehingga mendapatkan genome sampel yang menjadi tujuan penelitian dibandingkan metode lainnya. Metode chelex juga telah berhasil diterapkan pada sampel
JMRT, Volume 4 No 2 Tahun 2021, Halaman: 37-41 Tuna (Jackson et al., 2014; Iranawati et al., 2016; Pertiwi et al., 2017; Malik et al., 2020)..
-
.2.2.2 Polymerase Chain Reaction (PCR)
Proses amplifikasi DNA dilakukan pada mtDNA control region dan primer yang digunakan yaitu primer forward CRK 5’-AGC TCA GCG CCA GAG CGC CGG TCT TGT AAA-3’, primer reverse CRE 5’-CCT GAA GTA GGA ACC AGA TG-3’ (Lee et al., 1995). Primer ini digunakan secara universal terkait identifikasi genetik ataupun populasi Tuna (Pertiwi et al., 2017; Malik et al., 2020). Pengaturan tahapan PCR meliputi denaturasi awal pada suhu 80°C selama 10 detik, denaturasi pada suhu 94°C selama 30 detik, annealing pada suhu 50°C selama 30 detik, extension pada suhu 72°C selama 45 detik (Barber et al., 2006). Tahapan PCR ini diulang sebanyak 38 siklus.
-
2.2.3 Elektroforesis
Pengecekan kualitas produk DNA dengan menggunakan media gel agarosa. Bahan pembuatan gel agarosa adalah 0.75 gram bubuk agarosa dan 75 ml larutan buffer dimasukkan ke dalam gelas beker. Kemudian dipanaskan di dalam microwave selama 4 menit. Gel agarosa dituangkan ke dalam cetakan yang telah didiamkan selama 60 menit. Setelah mengeras, gel dimasukkan ke dalam mesin elektroforesis, kemudian ditambahkan 2 µl biotium, 1 µl loading dye ke masing – masing sampel. Kemudian mesin elektroforesis dinyalakan selama 30 menit dengan tegangan 100 V dan kecepatan arus 200 A. 2.4 Analisis Data
-
2.3 Analisis Data
-
2.3.1 Identifikasi Spesies
-
Analisis molekuler sekuen mtDNA control region menggunakan program MEGA5. Sekuen hasil penjajaran (alignment) dianalis dengan menggunakan Clustal W kemudian dibandingkan dengan sekuen yang ada di GenBank melalui program BLAST (Basic Local Alignment Search Tool) dengan situs web (http://www.ncbi.nih.gov). Hasil prosentase tertinggi dari nilai identifikasi dan nilai coery cover dari NCBI diambil sebagai acuan validasi identifikasi species secara genetik.
-
2.3.2 Analisis Filogenetik
Analisis filogenetik menggunakan program MEGA 5, dengan metode Neighbor Joining (kimura 2- parameter) dengan replikasi bootstraps 1000 (Tamura et al., 2011). Penentuan clade atau kelompok pada filogenetik ditentukan berdasarkan species yang mengelompok berdasarkan hasil identifikasi genetik atau jarak genetik yang dekat satu sama lain.
Sampel yang berhasil dikoleksi di Pelabuhan Benoa berjumlah 47 spesimen dan merupakan hasil tangkapan dari perairan Samudra Hindia dan Laut Flores Jumlah perkiraan spesies yang didapatkan berdasarkan pengamatan secara morfologi yaitu lima spesies. Adapun jumlah dari masing-masing sampel tersebut yaitu 7 sampel T. tonggol, 12 sampel T. alalunga, 8 sampel T. obesus, 13 sampel T. maccoyii, dan 7 sampel T. albacares. Dari 47 sampel tuna yang dikoleksi, hanya DNA dari 17 sampel yang berhasil diamplifikasi dengan metode PCR. Sebagian sampel yang tidak berhasil diamplifikasi mungkin dapat disebabkan karena sampel tuna
yang didapatkan merupakan hasil tangkapan yang sudah lama disimpan dalam keadaan beku, sehingga pada bagian sirip yang didapatkan sudah dalam keadaan kering dan kemungkinan kualitas dari DNA pada sirip tuna tersebut sudah tidak bagus. Kemudian dari 17 sampel tersebut dilakukan proses sekuensing lalu hasilnya dianalisis menggunakan program MEGA 5.2. Analisis dengan MEGA 5.2 menunjukkan ada 2 sampel yang memiliki kualitas kromatogram yang kurang baik sehingga dua sampel tersebut tidak diikutkan pada analisis berikutnya. Dengan demikian, dari 47 sampel yang dikumpulkan, hanya 15 sampel yang memenuhi syarat untuk dianalisis secara molekuler.
Pada saat pengambilan sampel terdapat lima spesies Thunnus yang teridentifikasi secara morfologi. Setelah dilakukan analisis molekuler terdapat satu spesies yang gagal dianalisis hingga tahap amplifikasi dengan metode PCR, yaitu T. albacares (tuna sirip kuning). Namun setelah dilakukan analisis lebih lanjut, hasil identifikasi BLAST menunjukkan terdapat lima spesies yang teridentifikasi. Berikut beberapa hasil perbedaan identifikasi secara morfologi dan molekular ikan tuna yang didapatkan di pelabuhan benoa (Tabel 1
Tabel 1. Hasil BLAST sampel tuna
|
No. |
ID |
Ident. Morfologi |
Ident. Blast |
Per. Ident. |
Acc. No. |
|
1 |
BIO06.035.002 |
T. tonggol |
T. tonggol |
97.57% |
HQ425780 |
|
2 |
BIO06.035.004 |
T. tonggol |
T. albacares |
95.90% |
KM588080 |
|
3 |
BIO06.035.005 |
T. tonggol |
T. albacares |
96.45% |
KT724724 |
|
4 |
BIO06.035.006 |
T. tonggol |
T. albacares |
96.26% |
KM588080 |
|
5 |
BIO06.035.007 |
T. tonggol |
T. albacares |
95.79% |
KM261642 |
|
6 |
BIO06.056.002 |
T. alalunga |
T. alalunga |
95.65% |
JN086151 |
|
7 |
BIO06.056.004 |
T. alalunga |
T. alalunga |
96.26% |
JN086151 |
|
8 |
BIO06.056.006 |
T. alalunga |
T. alalunga |
99.67% |
KP412763 |
|
9 |
BIO06.056.010 |
T. alalunga |
T. alalunga |
95.61% |
JN086151 |
|
10 |
BIO06.056.016 |
T. obesus |
T. obesus |
99.37% |
KM871985 |
|
11 |
BIO06.056.018 |
T. obesus |
T. obesus |
99.56% |
KM872028 |
|
12 |
BIO06.056.020 |
T. obesus |
T. obesus |
99.78% |
KM261661 |
|
13 |
BIO06.056.024 |
T. maccoyii |
T. maccoyii |
92.95% |
AB536519 |
|
14 |
BIO06.056.026 |
T. maccoyii |
T. maccoyii |
95.42% |
AB536518 |
|
15 |
BIO06.056.031 |
T. maccoyii |
T. maccoyii |
95.42% |
AB536517 |
Berdasarkan Tabel 1, dari 15 sampel tuna yang berhasil dianalisis terdiri dari 1 individu T. tonggol, 4 T. albacares, 4 T. alalunga, 3 T. obesus, dan 3 T. maccoyii. Hasil BLAST tersebut juga menunjukkan adanya perbedaan dengan hasil identifikasi morfologi. Sebagai contoh identifikasi secara morfologi menunjukkan ada 5 spesies T. tonggol tetapi setelah dilakukan BLAST ternyata hanya terdapat 1 individu T. tonggol. Hal tersebut dikarenakan beberapa sampel sulit dibedakan karena memiliki morfologi tampak sama terutama pada spesies T. tonggol dan T. albacares.
Selain itu, disebabkan juga karena keadaan sampel yang tidak utuh karena sudah dipotong pada bagian sirip dan
sebagian tubuh sampel sudah dilapisi es karena disimpan dalam penyimpanan beku. Beberapa jenis tuna yang sering didaratkan di Benoa berdasarkan validasi morfologi biasanya tuna sirip biru selatan (Thunnus maccoyii), tuna mata besar (Thunnus obesus), dan tuna sirip kuning (Thunnus albacares) (Proctor et al., 2006).
Perbedaan hasil identifikasi antara mofologi dan molekular ini akan mengganggu pengimplementasian perikanan berkelanjutan terutama dalam pencatatan jenis di tingkat spesies (Pauly and Froese, 2012). Saat ini, eksploitasi tuna di perairan Samudera Hindia setiap tahunnya mengalami peningkatan sehingga terindikasi over fishing (Nugraha dan Hufiadi, 2013).
-
3.2 Hubungan Filogenetik
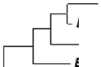
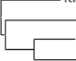
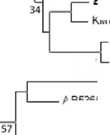
Filogenetik dianalisis dengan menggunakan metode Neighbor-joining dan model yang digunakan yaitu Kimura 2-parameter diperoleh lima clade (Gambar 2).
32
BIO06.035.005
----BIO06.035.007
BK>06.035.004
BIO05.035.005
KT724724.1 lħurι∩us albacares
KM261655.1 TTiunnus albacares
KM 261642.1 Thunnus albacares
Clade I
KU588080 1 Thunnus albacares
BIO05.055.016
HQ425780.1 TTiunnus Ionggol
---BIO06.035.002
JN086154.1 TTiunnus Ionggol
KM871985 1 Thunnus Obesus
Chde ∏
63
BIO06.056.031
B/O06.055.020 ]
KM261661.1 Thunnus obesus
BIO06.056.018
KM872028.1 TTiunnus obesus
AB536517.1 Thunnus maccoyii
85
100
Clade IΠ
BIO06.056.024
BK>06.05δ.026
Clade W
----------A0536519 1 Thunnus maccoyii
AB536518.1 Thunnus maccoyi
- AY302574 2 Thunnus thynnus
AB 106306.1 Thunnus thynnus
-------------JN086151.1 TTiunnus alalunga lBI006.056.006
1KP412763.1 Thunnus alalunga
---BIO06.056.002
Clade V
-------BIO06.056.004
KJ535757.1 Thunnus alalunga
BIO06.056.010
Gambar 2. Hasil pohon filogenetik ikan tuna menggunakan metode Neighbor Joining dengan model Kimura 2 parameter
Data pembanding dari GenBank menunjukkan bahwa clade I berkerabat dekat atau dapat dikatakan satu spesies dengan tuna sirip kuning (T. albacares), clade II dengan spesies tongkol abu-abu (T. tonggol), clade III dengan spesies tuna mata besar (T. obesus), clade IV dengan spesies tuna sirip biru selatan (T. maccoyii), dan clade V dengan spesies tuna albacore (T. alalunga).
Berdasarkan pohon filogenetik, clade tongkol abu-abu (T. tonggol) memiliki kekerabatan dekat dengan clade tuna sirip kuning (T. albacares) yang berdekatan, sedangkan clade tuna albacore (T. alalunga) terpisah jauh dengan clade T. tonggol yang menandakan kekerabatan yang jauh. Secara morfologi spesies tuna sirip kuning (T. albacares) dan tongkol abu-abu (T. tonggol) memiliki morfologi yang mirip dan menjadi sulit dibedakan karena pada saat pengambilan sampel di lapangan, keadaan kedua spesies ini sudah dalam keadaan sebagian sirip terpotong dan tubuh dilapisi es.
Jika dalam keadaan utuh kedua spesies tertebut memiliki ciri pembeda pada bagian perut dan sirip dada. Pada tuna sirip kuning (T. albacares) pada bagian perut terdapat bintik-bintik putih tesusun secara vertikal dan panjang sirip dada melewati sirip dorsal pertama sedangkan pada tongkol abu-abu sedangkan (T. tonggol) bintik-bintik tersusun secara horizontal dan panjang sirip dada tidak melewati sirip dorsal pertama. Secara morfologi tuna sirip kuning (T. albacares) lebih mudah dibedakan dengan tuna albacore (T. alalunga) karena tuna albacore (T. alalunga) tidak memiliki bintik-bintik pada
perutnya melainkan pita berwarna biru pudar disepanjang sisi tubuh dan memiliki sirip dada yang sangat panjang dengan ujung yang runcing (Collette and Graves, 2019). Rekontruksi dari pohon filogenetik ini didukung oleh hasil dari analisis nilai jarak genetik antar spesies.
Tabel 2. Matriks jarak genetik antar clade
|
1 |
2 |
3 |
4 |
5 |
6 | ||
|
T. albacares |
1 | ||||||
|
T. tonggol |
2 |
0.075 | |||||
|
T. obesus |
3 |
0.100 |
0.091 | ||||
|
T. maccoyii |
4 |
0.172 |
0.176 |
0.115 | |||
|
T. Thynnus |
5 |
0.186 |
0.180 |
0.142 |
0.149 | ||
|
T. alalunga |
6 |
0.212 |
0.197 |
0.172 |
0.200 |
0.172 | |
Berdasarkan Tabel 2, jarak genetik paling rendah (hubungan kekerabatan terdekat) terdapat pada clade T. albacares dengan T. tonggol yaitu 0.075, jarak genetik paling tinggi (hubungan kekerabatan terjauh) terdapat pada clade T. albacares dengan T. alalunga. Spesies tropis, tuna sirip hitam (T. atlanticus), tongkol abu-abu (T. tonggol), dan tuna sirip kuning (T. albacares) adalah anggota kelompok sirip kuning. Spesies yang mendiami perairan yang lebih dingin, tuna sirip biru selatan (T. maccoyii), tuna mata besar (T. obesus), tuna sirip biru utara (T. Thynnus) dan tuna albacore (T. alalunga) adalah
adalah anggota kelompok sirip biru (Chow, 1995). Collette (2001) mengusulkan bahwa Thunnus harus dibagi menjadi dua yang terdiri dari subgenus NeoThunnus tropis (T. albacares, T. tonggol, dan T. atlanticus) dan subgenus Thunnus yang lebih toleran dingin (T. Thynnus, T. maccoyii, T. alalunga, dan T. obesus).
Dalam hasil ini jarak genetik tongkol abu-abu dan tuna sirip kuning, memiliki jarak yang dekat dibandingkan jenis lainnya. Griffiths et al., (2007), menyebutkan bahwa hubungan karakteristik kedua jenis tuna tersebut memiliki kesamaan mirip dibandingkan jenis lainnya. Secara morfologi saat di tahap juvenile sampai remaja memiliki karakteristik yang sama dan sering tertukar identifikasi satu sama lain (Collette and Graves, 2019).
Benoa merupakan salah satu pelabuhan utama untuk pendaratan perikanan tuna di Indonesia (Ahmad et al., 2009). Hasil penelitian ini dapat menjadi acuan tambahan untuk validasi informasi data untuk mengontrol target spesies tuna yang didaratkan di Pelabuhan Benoa, Bali.
Terdapat lima spesies ikan tuna (Thunnus spp.) yang teridentifikasi secara molekuler di Pelabuhan Benoa, Bali yaitu tuna sirip kuning (T. albacares), tongkol abu-abu (T. tonggol), tuna mata besar (T. obesus), tuna sirip biru selatan (T. maccoyii), dan tuna albacore (T. alalunga). Hubungan filogenetik yang terdekat terdapat pada spesies tuna sirip kuning (T. albacares) dengan tongkol abu-abu (T. tonggol) yang mempunyai nilai jarak genetik sebesar 0.075 dan terjauh pada tuna sirip kuning (T. albacares) dengan tongkol abu-abu (T. alalunga) yang mempunyai nilai jarak genetik sebesar 0.212.
Ucapan Terima Kasih
Penulis mengucapkan terima kasih kepada dosen pembimbing pembimbing, penguji dan Biodiversitas Indonesia (BIONESIA) yang telah menyediakan fasilitas dan laboratorium serta semua pihak yang telah membantu dan membimbing dalam proses penyelesaian penelitian ini.
Daftar Pustaka
Ahmad, F., Dewanti, L.P., Arnenda, G.L., Rizal, A. 2019. Lengthweight relationship and catch size of bigeye tuna (Thunnus obesus) landed in Benoa, Bali, Indonesia. World News of Natural Sciences, 23, 34-42.
Barber, P.H., Erdmann, M.V., Palumbi, S.R. 2006. Comparative phylogeography of three codistributed stomatopods: Origins and timing of regional lineage disversification in the Coral Triangle. Evolution. 60(9): 1825–1839.
Chow S, Kishino H. 1995. Phylogenetic relationships between tuna species of the genus Thunnus (Scombridae: Teleostei): inconsistent implications from morphology, nuclear and mitochondrial genomes. Journal of Molecular Evolution, 41(6), 741-748.
Collette, B.B., Graves, J. 2019. Tuna and billfishes of the world.
Maryland : John Hopkins University Press. 352 pp
Collette B.B., Reeb C., Block B.A. 2001. Systematics of the tunas and mackerels (Scombridae). Fish physiology, 19, 1-33.
Gerasmio, I.R.P., Babaran, R.P., Santos, MD. 2012. Discrimination of Juvenile Yellowfin (Thunnus albacares) and Bigeye (T. obesus) Tunas
JMRT, Volume 4 No 2 Tahun 2021, Halaman: 37-41 using Mitochondrial DNA Control Region and Liver Morphology. PLoS ONE 7(4).
Griffiths, S.P., Fry, G.C., Manson, F.J., Pillans, R.D. 2007. Feeding dynamics, consumption rates and daily ration of longtail tuna (Thunnus tonggol) in Australian waters, with emphasis on the consumption of commercially important prawns. Marine and Freshwater Research, 58(4), 376-397.
Habibi, A., Anyogagautama, D., Sugiyanta. 2011. Perikanan Tuna-Panduan Penangkapan dan Penanganan. Jakarta (ID): WWF-Indonesia. 27 hal.
Iranawati, F., Nazifah, L., Harlyan, L.I., Sari, S.H.J., Arfiati, D. 2016. Determination on yellow fin tuna stock (Thunnus albacares) in South Java Sea based on genetic variation by restriction fragment length polymorphism (RFLP) method. Research Journal of Life Science, 3(1):06-15.
Itano, D.G., Fukofuka, S. 2007. Handbooks for the Identification of Yellowfin and Bigeye Tunas in (1) Fresh, (2) Frozen and (3) Fresh but Less than Ideal Condition. Pelagic Fisheries Research Program. Amerika Serikat (US): University of Hawaii. 27 pp.
Jackson, A.M., Erdmann, M.V., Toha, A.H.A., Stevens, L.A, & Barber, P.H. 2014. Phylogeography of commercial tuna and mackerel in the Indonesian Archipelago. Bulletin of Marine Science, 90(1):471-492.
Lee, W.J., Conroy, J., Howell, W.H, Kocher TD. 1995. Structure and evolution of teleost mitochondrial control regions. Moleculer Evolution, 41: 54-66.
Li, S., Pearl D.K., Doss H. 1999. Phylogenetic tree construction using Markov ChainMonte Carlo.Fred Huntchinson Cancer Research Center Washington. Fred Hutchinson Cancer Research Center Washington. 29 pp.
Majkowski, J. 2007. Global Fishery Resources of Tuna and Tuna-Like Species. FAO Fisheries Technical Paper. Rome (IT): FAO. 483:54
Malik, M.D.A., Pertiwi, N.P.D., Sembiring, A., Yusmalinda, N.L.A, Ningsih, E.Y., Astarini, I.A. 2020. Genetic structure of Longtail Tuna Thunnus tonggol (Bleeker, 1851) in Java Sea, Indonesia. Biodiversitas Journal of Biological Diversity, 21(8).
Miazwir. 2012. Analisis aspek biologi reproduksi ikan tuna sirip kuning (Thunnus albacares) yang tertangkap di Samudera Hindia. Universitas Indonesia. Jakarta.
Nugraha, B., Hufiadi, H. 2013. Efisiensi Teknis Perikanan Rawai Tuna Di Benoa (Studi Kasus: Pt. Perikanan Nusantara). Jurnal Penelitian Perikanan Indonesia, 19(1), 25-30.
Pauly, D., Froese, R. 2012. Comments on FAO’s State of Fisheries and Aquaculture, or ‘SOFIA 2010’. Mar. Policy 36, 746–752.
Pertiwi, N.P.D., Nugraha, B., Sulistyaningsih R.K., Jatmiko, R., Sembiring, A., Mahardini, A., Cahyani, N.K.D., Anggoro, A.W., Madduppa, H.H., Ambariyanto, A., Barber, P.H., Mahardika, G.N. 2017. Short Communication: Lack of differentiation within the bigeye tuna population of Indonesia. Biodiversitas 18 (4): 1406-1413.
Proctor, C.H., Andamari, R., Retnowati, D., Herrera, M., Poisson, F., Fujiwara, S., Davis, T.L. 2006. The catch of SBT by the Indonesian longline fishery operating out of Benoa, Bali in 2005. In CCSBT 7th Meeting of the Stock Assessment Group and the 11th Meeting of the Extended Scientific Committee, Tokyo, Japan., 4-11.
Tamura, K., Peterson D., Peterson, N., Stecher, G., Nei, M., Kumar, S. 2011. MEGA5: Molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance and maximum parsimony method. Molecular Biology Evolution, 28(10): 2731–2739.
Viñas, J., Tudela, S., 2009. A Validated Methodology for Genetic Identification of Tuna Species (Genus Thunnus). PLoS ONE4(10):e7606.https://doi.org/10.1371/journal.pone.0007606
Walsh, P.S., Metzger, D.A., Higuchi, R. 1991. Chelex-100 as a medium for Simple extraction of DNA for PCR based typing from forensic material. Biotechniques, 10: 506-513.
Zein, M.S.A., Prawiradilaga D.M., 2013. DNA Barcode Fauna Indonesia. Jakarta: Kharisma Putra Utama.
41
Discussion and feedback