Uji Aktivitas SOD (Superoxide Dismutase) Bakteri Limbah Cair Tahu dengan Metode WST-1 (Water Soluble Tetrazolium Salt-1) dan Identifikasi Molekuler Bakteri Menggunakan PCR 16S rDNA
on

JURNAL FARMASI UDAYANA | pISSN: 2301-7716; eISSN: 2622-4607 | VOL. 12, NO. 2, 2023
https://doi.org/10.24843/JFU.2023.v12.i02.p03
Uji Aktivitas SOD (Superoxide Dismutase) Bakteri Limbah
Cair Tahu Dengan Metode WST-1 (Water Soluble Tetrazolium Salt-1) dan Identifikasi Molekuler Bakteri Menggunakan Pcr 16S rDNA
Arum Pinesti1, Ana Indrayati1 dan Lukito Mindi Cahyo1
-
1 Program Studi S1 Farmasi Fakultas Farmasi, Universitas Setia Budi, Jl. Letjen Sutoyo, Mojosongo, Jebres, Surakarta, Jawa Tengah
57127
Reception date of the manuscript: 17 Januari 2023
Acceptance date of the manuscript: 23 November 2023
Publication date: 31 Januari 2024
Abstract— Superoxide dismutase (SOD) is an endogenous antioxidant enzyme that can be produced by several microorganisms such as bacteria which are used to neutralize free radicals. The aims of this study were to isolate and identify SOD-producing bacteria from tofu wastewater, test SOD activity and molecularly identify by PCR 16S rDNA the bacteria with the highest SOD activity. The stages of the research included: isolation of bacteria from tofu liquid waste using NA media. Five bacterial isolates were selected and identified based on differences in morphology. The next stage was SOD extraction, determination of protein content with the BCA Assay and activity testing using the WST-1 Assay Kit. Bacteria with the highest SOD activity were identified molecularly using 16S rDNA PCR. Five bacterial isolates were successfully isolated. LCT 1, 2 and 3 are Gram positive while LCT 4 and 5 are Gram negative. Enzyme extraction was carried out on the five isolates and then precipitated with 80 % ammonium sulfate. LCT 5 isolate had the highest SOD activity, namely 87,90 %. The results of the identification of the 16S rDNA PCR were Pseudomonas aeruginosa strain HSS-6.
Keywords—tafu liquid waste, SOD, WST-1, PCR 16 S rDNA, bacteria
Abstrak— Superoksida dismutase (SOD) merupakan enzim antioksidan endogen yang dapat dihasilkan oleh beberapa mikroorganisme seperti bakteri yang digunakan untuk menetralisir radikal bebas. Tujuan penelitian ini adalah isolasi dan identifikasi bakteri penghasil SOD dari limbah cair tahu, menguji aktivitas SOD dan identifikasi molekuler dengan PCR 16S rDNA bakteri yang memiliki aktivitas SOD tertinggi. Tahapan penelitian meliputi: isolasi bakteri dari limbah cair tahu menggunakan media NA. Lima isolat bakteri dipilih dan diidentifikasi berdasarkan perbedaan morfologi. Tahap selanjutnya ekstraksi SOD, penetapan kadar protein dengan BCA Assay dan uji aktivitas menggunakan WST-1 Assay Kit. Bakteri dengan aktivitas SOD tertinggi didentifikasi molekuler menggunakan PCR 16S rDNA. Lima isolat bakteri berhasil diisolasi. LCT 1, 2 dan 3 termasuk Gram positif sedangkan LCT 4 dan 5 termasuk Gram negatif. Ekstraksi enzim dilakukan pada kelima isolat lalu dipresipitasi dengan amonium sulfat 80 %. Isolat LCT 5 memiliki aktivitas SOD tertinggi yaitu 87,90 %. Hasil identifikasi PCR 16S rDNA adalah Pseudomonas aeruginosa strain HSS-6.
Kata Kunci—limbah cair tahu, SOD, WST-1, PCR 16S rDNA, bakteri
Limbah cair tahu yang merupakan bahan sisa dari proses produksi tahu mengandung senyawa organik berupa protein 40 hingga 60 %, karbohidrat 25 hingga 50 % dan lemak 10 % (Sugiharto, 1994). Senyawa organik yang terdapat dalam limbah cair tahu dapat diuraikan oleh mikroorganisme salah satunya yaitu bakteri. Bakteri termasuk mikroorganisme prokariotik yang memiliki bentuk bervariasi, tidak memiliki membran inti serta memiliki informasi genetik beru-
Penulis koresponden: Panesti, 25195954a@mhs.setiabudi.ac.id
pa DNA namun tidak terlokasikan di nukleus (Jean B et al., 2017). Superoxide dismutase (SOD) adalah jenis metaloen-zim yang diproduksi di sitosol dan mengandung atom tembaga (Cu), seng (Zn) atau besi (Fe). SOD yang mengandung mangan (Mn) diproduksi dalam matrik mitokondria dengan mengkatalisis dismutase pada superoxide menjadi peroxide dan oksigen. Enzim superoxide dismutase (SOD) merupakan suatu antioksidan endogen yang mengatur kadar ROS di dalam tubuh. Enzim superoxide dismudase memiliki manfaat dalam berbagai bidang, diantaranya dalam bidang kosmetik dapat digunakan sebagai agen anti penuaan dini (Gou-tam Brahmachari, 2013). Dewasa ini untuk mengidentifikasi DNA suatu bakteri analisis yang paling umum digunakan
yaitu gen 16S rRNA (16S Ribonucleic acid). Analisis se-kuensing gen 16S rRNA (16S Ribonucleic acid) merupakan teknik identifikasi molekuler yang lebih cepat, lebih sensitif dan tepat sedangkan untuk gen lain seperti 5S dan 23S lebih sulit untuk identifikasi karena panjang rantai urutan gen 5S terlalu pendek hanya 120 bp dan 23S terlalu panjang sekitar 2900 bp (Akihary dan Kolondam., 2020). Panjang rantai urutan gen 1,550 bp 16S rRNA terdiri dari bagian-bagian yang dilestarikan (conserved regions). Gen ini memiliki ukuran yang memadai dan polimorfisme interspesifik, untuk menunjukkan perbedaan dan menghasilkan data yang signifikan secara statistik. Penelitian yang telah dilakukan oleh Lestari (2019) menyatakan bahwa bakteri yang memiliki aktivitas superoksida dismutase dari hasil isolasi tanah hutan mangrove di Semarang yaitu Bacillus altitudinis dengan aktivitas SOD jenis Cu-SOD. Pada penelitian ini akan diisolasi bakteri penghasil SOD limbah cair tahu, menguji aktivitas SOD serta identifikasi molekuler dengan PCR 16S rDNA bakteri yang memiliki aktivitas SOD tertinggi.
Sampel bakteri limbah cair tahu, media NA, BHI, pewarna Gram, PBS, NaCl, seperangkat reagen uji protein BCA (Pierce, nomor katalog: 23227), larutan standar protein BSA (Bovine Serum Albumin), air suling ganda, kit SOD (19160). Alat yang digunakan adalah seperangkat alat gelas, cawan petri setril, jarum ose, inkas, lampu spiritus, objek glass, mikroskop binokuler Leica DM500, inkubator, autoclave, oven, alat sentrifugasi, timbangan analitik, shaker, mikro-pipet, spektrofotometer, seperangkat alat PCR, seperangkat alat elektroforesis gel dan gel doc, Erlenmeyer, 96 well microplate.
Instrumen kaca disterilkan selama 60-120 menit pada suhu 150-170°C dalam oven. Media yang digunakan disterilkan menggunakan autoklaf pada suhu 120°C selama 15 menit pada tekanan 1 atmosfer (1 atm) sedangkan jarum ose disterilkan dengan menggunakan pembakaran di atas spiritus.
Sampel diambil langsung menggunakan botol kaca steril dengan kedalaman 5 cm sebanyak 150 mL.
Isolasi bakteri dilakukan dengan metode gores. Sampel diambil 1 mL, kemudian disuspensikan dalam 9 mL NaCl lalu dihomogenkan menggunakan vortex. Sebanyak 1 mL dari suspensi diambil lalu dicampurkan ke dalam 9 mL NaCl pada tabung reaksi lainnya. Suspensi ini adalah faktor pengenceran 10-1. Langkah yang sama dilakukan untuk membuat pengenceran 10-2 dan 10-3. Isolasi bakteri dilakukan pada tingkat pengenceran 10-3. Secara aseptis digoreskan menggunakan ose steril ke dalam media NA yang sebelumnya sudah dipadatkan lalu dinkubasi selama 24 jam pada suhu 37 °C sampai diperoleh koloni yang tumbuh (Farmakope Indonesia edisi VI, 2020).
Pewarnaan Gram dilakukan dengan membuat preparat ulas (smear), lalu difiksasi selanjutnya ditetesi pewarna Gram A (kristal violet) sebagai pewarna utama. Preparat didiamkan selama ±1 menit, kemudian dicuci menggunakan aquadest mengalir. Preparat yang sudah dicuci, ditetesi dengan mordant sebagai pewarna Gram B. Pewarna mordant berisi lugol dan iodin dan berfungsi sebagai penguat warna. Setelah ditetesi mordant, preparat didiamkan selama ±1 menit kemudian cuci kembali dengan air mengalir lalu dikering anginkan. Preparat yang sudah kering dilunturkan dengan alkohol (Gram C) dan didiamkan selama ±45 detik, lalu dicuci dengan aquadest mengalir dan dikeringkan. Langkah berikutnya yaitu dengan memberikan larutan safranin (Gram D) pada preparat selama 30 detik kemudian dicuci kembali dengan air mengalir dan dikeringkan. Preparat yang akan diamati diba-wah mikroskop, diberi minyak emersi diatasnya, kemudian diamati dengan perbesaran 10× atau 100×. Jika bakteri Gram positif hasil pewarnaan Gram akan berwarna ungu namun jika berwarna merah bakteri tersebut termasuk Gram negatif (Amaliah et al., 2018).
Ekstraksi enzim dilakukan dengan menyiapkan suspensi bakteri sebanyak 2 ose biakan dari media agar miring dimasukkan ke dalam media BHI 10 ml dan dihomogenkan menggunakan vortex. Suspensi bakteri distandarkan menggunakan larutan standar McFarland 0,5. Pengenceran dapat dilakukan jika suspensi yang dibuat masih terlalu pekat atau belum setara. Suspensi tersebut diinkubasi pada suhu 37°C selama 24 jam setelah distandarisasi. Ekstraksi enzim dilakukan dengan menginokulasikan suspensi bakteri 2 % ke dalam Erlenmeyer yang berisi 100 mL media cair BHI. Suspensi tersebut sebelum diinkubasi kembali selama 24 jam pada suhu 37°C, diaduk menggunakan shaker dengan kecepatan 100 rpm hingga larut optimal. Ekstraksi enzim dilakukan untuk memisahkan sel dari supernatannya dengan cara disentrifugasi dengan kecepatan 5000 rpm pada suhu 4°C selama 60 menit. Hasil ekstraksi enzim yang berupa supernatan disimpan selama 24 jam pada suhu 4°C dalam lemari es (Anggraini, 2018).
Presipitasi dengan ammonium sulfat pada dasarnya adalah prinsip dari permurnian enzim menggunakan ammonium sulfat 80 %. Ekstrak enzim kasar secara bertahap ditambah dengan ammonium sulfat yang kemudian diaduk menggunakan magnetic stirrer sampai benar-benar larut. Selama 24 jam campuran disimpan pada suhu 4°C dalam lemari es. Setelah itu, campuran dipisahkan dengan sentrifugasi pada 4°C selama 30 menit dengan kecepatan 5000rpm. Pelet yang telah disentrifugasi ditimbang dan dilarutkan dengan 1:2mL PBS (Phosphate Buffer Saline) 0,05 M pH 7,0. Setelah ditimbang dan disuspensikan kembali, pelet dipecah secara mekanik dengan sonikasi pada suhu 4°C selama lima menit dengan jeda interval setiap satu menit pada amplitudo 50 (Anggrai-ni, 2018).
Prinsip kerja dari metode BCA™ Protein Assay adalah formulasi yang kompatibel dengan deterjen berdasarkan bi-cinchoninic acid (BCA) untuk deteksi kolorimetri dan kuan-
tisasi protein total. Metode ini menggabungkan reduksi Cu+2 ke Cu+1 oleh protein dalam media alkali (reaksi biuret) dengan deteksi kolorimetri yang sangat sensitif dan selektif dari kation tembaga (Cu+1) menggunakan reagen unik yang mengandung bicinchoninic acid. Produk reaksi berwarna ungu dari pengujian ini dibentuk oleh khelasi dua molekul BCA dengan satu ion tembaga. Kompleks yang larut dalam air ini menunjukkan absorbansi yang kuat pada 562 nm yang hampir linier dengan peningkatan konsentrasi protein pada rentang kerja yang luas (20-2.000 µ g/ml). Langkah untuk menentukan kadar protein menggunakan metode BCA disesuaikan dengan yang terdapat pada katalog PierceTM BCA Protein Assay Kit (He F, 2011).
-
2.2.2.6 Uji Aktivitas Enzim Superoksida Dismutase (SOD) pada Bakteri yang Terdapat di Limbah Cair Tahu dengan Metode WST-1
Uji aktivitas SOD dapat dilakukan menggunakan kit SOD (19160) yang dibeli dari Sigma-Aldrich (Chemie GmbH, Jerman). Prosedur kerjanya yaitu dengan mencampurkan pereaksi yang terdiri dari 1 mL larutan kerja (WST) lalu diencerkan dengan 19 mL larutan buffer. Tabung larutan enzim disentrifugasi selama 5 detik. Selanjutnya 15 µL larutan enzim diencerkan dengan 2,5 mL buffer pengenceran SOD. SOD diencerkan dengan buffer pengenceran untuk membuat larutan standar SOD sebagai berikut: 200 U/mL, 100 U/mL, 50 U/mL, 20 U/mL, 10 U/mL, 5 U/ mL, 1 U/mL, 0,1 U/mL, 0,05 U/mL, 0,01 U/mL, dan 0,001 U/mL. Tambahkan sebanyak 20 µL larutan sampel ke setiap sampel dan sumur blanko 2 sedangkan 20 µL ddH2O (air suling ganda) ditambahkan ke masing-masing blangko 1 dan sumur blangko 3. 200 mL WST ditambahkan ke masing-masing sumur kemudain dicampur. Sumuran blanko 2 dan blanko 3, ditambahkan 20 mL buffer pengenceran kemudian 20 µL larutan kerja enzim ditambahkan ke masing-masing sampel dan sumur blanko 1, kemudian diaduk hingga homogen. Langkah berikutnya pelat diinkubasi pada suhu 37 °C selama 20 menit, absorban-si dibaca dengan microplate reader pada panjang gelombang 450 nm (Qwele, 2013).
Dalam microtube 1,5 mL, diisi dengan 200 L suspensi bakteri, 20 µL proteinase K dan 200 µL buffer AL. Vortex microtube selama 15 detik lalu inkubasi pada suhu 56 °C selama 15 menit dan 96 °C selama 10 menit. Sebanyak 200 µL ethanol absolut ditambahkan dalam campuran tersebut lalu vortex selama 15 detik. Kemudian campuran tersebut dipindahkan dalam tube berfilter (mini column) lalu disentri-fugasi selama 3 menit pada kecepatan 8000 rpm. Supernatan dibuang dan bilas filter menggunkan 750 L buffer AW1 (washing buffer). Buffer AW2 digunakan untuk mengulangi proses pembilasan ini dengan cara yang sama. Selanjutnya dilakukan sentrifugasi dengan kecepatan 14000 rpm selama 1 menit. Tahap terakhir melibatkan sentrifugasi filter pada 8000 rpm selama 1 menit setelah membilasnya dengan 200 L buffer AE (elution buffer). Buang filter lalu simpan supernatant pada suhu –20 °C untuk digunakan sebagai template DNA (Sunarno et al., 2014).
Gen 16S rDNA yang sudah diekstraksi kemudian diampli-fikasi dengan PCR menggunakan sepasang primer spesifik, UniBI (5’-GGTTAC(GC)TTGTTACGACTT-3’) dan B act F-I (5’AGAGTTTGATC(AC)TGGCTCAG-3’) (Weisburg et al., 1991). Perangkat Thermal Cycler Veriti Aplied Biosystem diisi dengan tabung PCR. Thermal Cyler dinyalakan dan kondisi PCR sebagai berikut: predenaturasi 95°C selama 2 menit sebanyak 1 siklus, denaturasi 94°C selama 1 menit, annealing 58°C selama 45 detik, extension 72°C selama 45 detik, post extension 72°C selama 5 menit sebanyak 34 siklus. Pita DNA dari proses amplifikasi terlihat setelah hasil PCR dielektroforesis (Sjafaraenan et al., 2018).
Proses visualisasi gen 16S rDNA menggunakan elektroforesis gel agarosa 1 % dengan menimbang 1 gram agarose kemudian dimasukkan dalam Elenmeyer yang sudah berisi TAE (Tris asetat EDTA) buffer 1x sebanyak 100 ml. Panaskan dalam microwave selama 2 menit atau sampai mendidih. Tambahkan 8 µL Ethidium Bromida (EtBr), cairan didiamkan pada suhu kamar hingga dingin. Gunakan sisir gel dengan jumlah sisir 17 sumur lalu tuangkan gel cair ke cetakan elektorforesis gel. Ambil 2 µ L loading dye dan campurkan ke dalam 5 L PCR produk setiap sampel, selanjutnya masukkan ke dalam sumur yang ada pada gel agarose. Ambil 5 µL ladder 100 bp dan masukkan dalam sumur paling ujung dari gel agarose. Setelah menghentikan elektroforesis dan mengeluarkan gel agarosa untuk diperiksa dibawah sinar UV dan dimasukkan ke dalam Gel Documentation untuk melihat pita amplifikasi,sumber listrik dinyalakan dengan tegangan 100 volt selama 50 menit. Jika terdapat pita DNA pada gel yang sejajar dengan marker artinya hasil positif, jika tidak ada maka negatif (Sjafaraenan et al., 2018).
Sekuensing dilakukan dengan metode Sanger menggunakan Automatic DNA Sequencer yang berdasarkan pada metode dye terminator labeling, dimana setiap nukleoida ditunjukkan oleh warna yang berbeda. Nukleotida hijau melambangkan Adenin (A), hitam untuk basa Guanin (G), merah untuk Timin (T) dan Sitosin (C) ditunjukkan dengan warna biru (Sjafaraenan et al., 2018).
Hasil sekuensing dianalisis menggunakan Basic Local Alignment Search Tool (BLAST) dengan server online (www.ncbi.nlm.nih.gov). Analisis ini bertujuan membandingkan informasi sekuens primer biologi misalnya sekuens nukleotida (DNA atau RNA) menggunakan program bioin-formatika yang ada di NCBI.
Sampel diambil langsung menggunakan botol kaca steril dengan kedalaman 5 cm sebanyak 150 mL yang berasal dari salah satu industri tahu. Pengambilan secara langsung ditujukan agar kotoran yang terdapat dipermukaan tidak ikut terbawa.
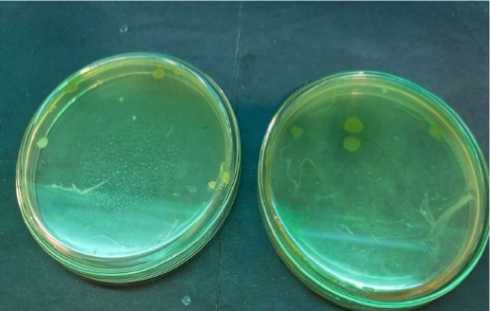
Gambar. 1: Hasil Isolasi sampel pengenceran 10-3
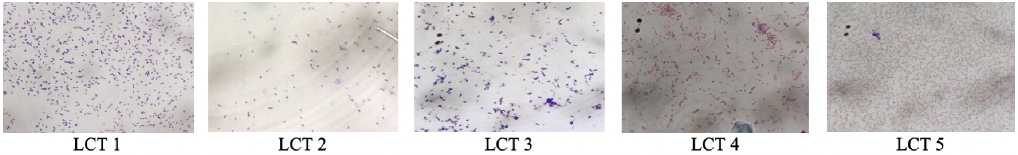
Gambar. 2: Hasil pewarnaan Gram
∣ CPIN985J 733O9I∙733978 BeiktoniiM* KtugiimM PAI I JinMiisunie
-
1 CPI0l695.i.l7U87-Π2374.Be^≡ κnig⅛tt.Mh-∞ICWl
-
1 f I1IOWI 1.1 bM7l-7007JVSaιdι^
CP093965.1.750165-751052 Beiktomun* uugitiM i≡ AICC BAA-CPIOI695J.54OI33I-54O22I8J1S^
-
■ MH018270.1.4-870.PsudoαιoM⅛iefl⅛iwM>ian IISS-6 IfcSjiMMi ON91MΛI ∣6-9O8 PkikIiuniMaj XtugitkM αmn PBU IfcS ribuωaι CP093965.l.5377 I99-5378O86 JtarilHMi, uugik>M anh ATCC BA ,CPIO½93.1.44832234184110. KiktonnMM auugik∣M an» 2O2ICK∙ 'CP093965.l 6I523O2-6153389 KiktoniiMM UiigitkiM an» ATCC. BA 1CHMKM 63lfc707-63l7594.BeuitonaiM.HiigDOi<PAI4.d>MiiMMi 'CPl(M985J.55∙K)25O-55-lll37.^^ PAU Jimiummi
∖p∣OI985J.49562354957 U2JtariMθ^ PAU Jimiumi
Metode gores pada media NA dengan pengenceran 10-3 dilakukan untuk mengisolasi bakteri. Koloni bakteri yang berhasil tumbuh diamati secara makroskopis berdasarkan morfologinya yang meliputi ukuran isolat bakteri limbah cair tahu LCT 1 kecil, LCT 2,3 dan 4 sedang serta LCT 5 besar. Bentuk sirkuler dengan elevasi datar dan tepi entire. Warna isolat LCT 1,2,4 dan 5 hijau sedangkan isolat LCT 3 putih (Tabel 1). Tahap penting dalam proses identifikasi adalah isolasi dan karakterisasi morfologi bakteri dari segi susunan
dan struktur sel. Morfologi yang serupa menunjukkan bahwa isolat tersebut termasuk dalam genus yang sama. Lima koloni yang telah dipilih dari cawan petri kemudian dilakukan isolasi kuadran empat untuk mencari dan memastikan koloni bakteri yang tumbuh adalah koloni tunggal yang selanjutnya koloni-koloni tersebut diinokulasikan selama 24 jam pada suhu 37 °C pada media NA miring (Safrida et al., 2012).
Hasil identifikasi mikroskopis berupa pewarnaan Gram dari kelima isolat menunjukkan bentuk yang sama yaitu basil atau batang namun jenis Gram yang berbeda. Isolat bakteri
Tabel 2: HASIL EKSTRAKSI ENZIM SOD
|
Isolat |
Volume sampel (mL) |
Supernatan (mL) |
Endapan (g) |
|
LCT 1 |
100 |
77 |
2,253 |
|
LCT 2 |
100 |
70 |
2,228 |
|
LCT 3 |
100 |
89 |
1,427 |
|
LCT 4 |
100 |
70 |
1,106 |
|
LCT 5 |
100 |
78 |
3,324 |
Tabel 1: HASIL IDENTIFIKASI MAKROSKOPIS BAKTERI LIMBAH CAIR TAHU
|
Isolat |
Ukuran koloni |
Bentuk koloni |
Warna koloni |
Elevasi |
Tepi koloni |
|
LCT 1 |
Kecil |
Sirkuler |
Hijau |
Datar |
Entire |
|
LCT 2 |
Sedang |
Sirkuler |
Hijau |
Datar |
Entire |
|
LCT 3 |
Sedang |
Sirkuler |
Putih |
Datar |
Entire |
|
LCT 4 |
Sedang |
Sirkuler |
Hijau |
Datar |
Entire |
|
LCT 5 |
Besar |
Sirkuler |
Hijau |
Datar |
Entire |
Tabel 3: HASIL PRESIPITASI ENZIM DENGAN AMMONIUM
Sulfat 80 %
Isolat Endapan (g)
Tabel 4: HASIL PENETAPAN KADAR PROTEIN TOTAL
|
Isolat |
Nilai Konsentrasi protein (mg/mL) |
X±SD |
|
LCT 1 |
2,21 |
2,21 ± 0,0785 |
|
LCT 2 |
1,95 |
1,95 ± 0,0419 |
|
LCT 3 |
1,69 |
1,69 ± 0,0087 |
|
LCT 4 |
2,07 |
2,07 ± 0,0472 |
|
LCT 5 |
2,05 |
2,05 ± 0,0684 |
Tabel 5: HASIL UJI% INHIBISI DENGAN WST-1
|
Isolat |
% inhibisi |
X±SD |
|
LCT 1 |
17,74 |
17,74 ± 0,0014 |
|
LCT 2 |
54,03 |
54,03 ± 0,0007 |
|
LCT 3 |
66,93 |
66,93 ± 0,0007 |
|
LCT 4 |
80,65 |
80,65 ± 0,0 |
|
LCT 5 |
87,90 |
87,90 ± 0,0007 |
LCT 1, LCT 2 dan LCT 3 termasuk Gram positif yang ditandai dengan warna ungu sedangkan LCT 4 dan LCT 5 termasuk Gram negatif yang ditandai dengan warna merah. Bakteri gram positif memiliki dinding peptidoglikan yang lebih tebal daripada Gram negatif sehingga ketika bakteri Gram positif diwarnai, mereka akan tetap menghasilkan warna ungu saat diamati dibawah mikroskop. Meskipun dilunturkan dengan alkohol, pigmen kristal ungu masih dapat diikat oleh pepti-doglikan yang tebal ini. (Hamidah et al., 2019).
Teknik pewarnaan Gram bakteri harus dilakukan secara merata agar bakteri tidak saling bertumpuk dan memudahkan dalam pengamatan mikroskopis. Bakteri difiksasi menggunakan pemanasan api bunsen dengan tujuan untuk membunuh bakteri dan melekatkan bakteri di atas objek glass. Membunuh bakteri tidak menyebabkan perubahan dan struktur bakteri serta mengubah daya ikat zat warna (Ibrahim et al., 2015).
Ekstraksi enzim ini bertujuan untuk mengeluarkan enzim yang ada pada bakteri tersebut dan memisahkan enzim dari zat pengotor. Suspensi bakteri yang diekstraksi enzim di-sentrifugasi dengan kecepatan 5000 rpm selama 60 menit. Sentrifugasi bertujuan untuk memisahkan supernatan yang diduga ekstrak kasar enzim dengan pengotornya. Prinsip pemisahan sentrifugasi adalah sistem pemisahan berdasarkan ukuran dan berat molekul. Partikel mengendap pada kecepatan yang berbeda sesuai dengan bobot, ukuran, dan bentuknya. Proses sentrifugasi dilakukan pada suhu rendah karena enzim bersifat mudah terdenaturasi pada suhu tinggi. Enzim yang diisolasi terdapat di filtrat (supernatan) sedangkan endapan ekstraksi adalah zat pengotor (Masruroh et al., 2018).
Supernatan yang diperoleh kemudian dipretisipasi menggunakan amonium sulfat. Tujuan presipitasi enzim adalah untuk memisahkan enzim yang ada di dalam sel dari senyawa yang tidak diinginkan atau pengotor. Prinsip presipitasi dengan amonium sulfat 80% yaitu penambahan garam amonium sulfat pada konsentrasi tinggi akan menyebabkan semakin berkurangnya kelarutan protein dalam larutan karena garam menyerap air pada sampel (Sinaga et al., 2014). Hasil presipitasi pada tabel 3 diperoleh hasil endapan yang berbeda-beda. Hal ini dapat disebabkan karena faktor hasil pemisahan pelet dan supernatan dari sentrifugasi pertama menghasilkan volume supernatan yang berbeda-beda sehingga berpengaruh pada penambahan amonium sulfat disetiap sampel juga berbeda.
Pelet yang diperoleh diresupensi dengan PBS dengan perbandingan 1:2 (pelet yang didapat: PBS) untuk memper-
Tabel 6: HASIL ANALISIS BLAST ISOLAT LCT 5 DENGAN DATABASE NCBI
Spesies Terdekat Pseudomonas aeruginosa strain HSS-6
Query Coverage 96%
% Maximum Identity 100%
Accession Number
MH038270
tahankan pH supaya tidak mengalami perubahan dan digunakan sebagai media pengencer untuk pelet isolat bakteri. Pelet hasil resuspensi kemudian dilakukan pemecahan melalui metode sonikasi. Metode sonikasi berfungsi untuk memecahkan membran sel, yang melepaskan semua protein ke dalam larutan. Prinsip dasar sonikasi adalah pemisahan dengan getaran berdasarkan frekuensi gelombang ultrasonik 15-25 kHz. Dinding sel bakteri dapat dirusak oleh kavitasi yang disebabkan oleh getaran ultrasonik ini. Supernatan yang mengandung ekstrak enzim intraseluler digunakan untuk penetapan kadar protein disimpan pada suhu 4°C dalam lemari pendingin (Rizky et al., 2022).
Penetapan kadar dalam penelitian ini menggunakan metode BCA Protein Assay Kit dari Thermoscientific. Metode ini dipilih karena dibandingkan dengan metode Bradford, metode ini lebih objektif dan universal (He F, 2011). Baku pembanding yang digunakan yaitu BSA (Bovine Serum Albumin) 2mg/mL. BSA digunakan sebagai baku pembanding dalam penetapan kadar ini karena mampu memberikan tingkat keakuratan yang tinggi (Azhar et al., 2019). Hasil penetapan kadar metode BCA Assay pada tabel 4 terlihat bahwa kadar dari setiap sampel berbeda karena pada sampel mengandung buffer dalam konsentrasi cukup tinggi sehingga dapat mengganggu pembacaan absorbansi di masing-masing sampel. Metode penetapan kadar BCA Protein Assay Kit memiliki kekurangan yaitu rentan terhadap gangguan seperti zat pereduksi, zat pengkhelat logam dan buffer dengan konsentrasi tinggi (He F, 2011).
Berdasarkan protokol uji WST-1 (Sigma-Aldrich), metode ini didasarkan pada pembelahan garam tetrazolium WST-1 menjadi formazan oleh dehydrogenase mitokondria seluler. Semakin besar jumlah sel hidup maka semakin tinggi aktivitasnya dan semakin besar jumlah pewarna formazan yang terbentuk. Uji aktivitas SOD dengan menggunakan kit SOD yang menghasilkan pewarnaan formazan. Produk WST-1, di-dalamnya terdapat tiga blanko yang digunakan untuk uji aktivitas SOD yang terdiri dari blanko 1 (WST, pereaksi enzim dan air), blanko 2 (sampel uji dan WST), serta blanko 3 (air, WST dan buffer). Ketika melakukan pengujian karena superoksida akan segera dilepaskan setelah penambahan larutan kerja enzim ke dalam sumur, maka gunakan pipet multisaluran untuk menghindari jeda waktu reaksi di setiap sumur. Pembacaan absorbansi dilakukan pada panjang gelombang 450nm. Berdasarkan tabel 5, isolat bakteri LCT 5 yang menunjukkan persentase aktivitas SOD tertinggi dengan nilai persen inhibisi mencapai 87,90 %. Nilai persen inhi-bisi menunjukkan besarnya aktivitas enzim SOD yang dihasilkan oleh isolat bakteri untuk menghambat reaksi radikal bebas. Agen penganggu seperti asam askorbat 0,1mM, glu-tation bentuk tereduksi 5mM dan Albumin Serum Bovine
-
5 %b/v dapat mempengaruhi hasil uji metode ini.
-
4 .6 Identifikasi molekuler dengan PCR 16S rDNA dan DNA sekuensing
Identifikasi molekuler 16S rDNA berupa sekuen yang dapat digunakan untuk mengidentifikasi bakteri dari urutan pasangan basanya, sehingga diperoleh hasil yang lebih akurat. Metode molekuler ini didasarkan pada teknik PCR (Polymerase Chain Reaction) yang merupakan teknik penggandaan DNA secara in vitro. Penggunaan metode PCR 16S rDNA banyak digunakan untuk mengetahui filogeni bakteri karena informasi dari sekuen gen 16S rDNA pada data base gene bank cukup lengkap (Pananjung et al., 2016).
Secara prinsip dasar PCR yaitu melibatkan beberapa tahapan secara berurutan. Tahapan dimulai dengan denaturasi atau pemisahan rantai DNA, kemudian annealing atau penempelan pasangan primer pada untai DNA target dan elon-gasi atau pemanjangan primer dimana reaksi polimerisasi ini dikatalis oleh enzim DNA polimerase. Proses PCR diperlukan cetakan atau template berupa DNA kromosom untuk membentuk molekul DNA baru melalui isolasi DNA kromosom. Hasil analisis pensejajaran menggunakan BLAST nu-kleotida gen 16S rDNA dari isolat LCT 5 dengan database genkBank NCBI menunjukkan hasil bahwa isolat LCT 5 memiliki kedekatan dengan bakteri Pseudomonas aeruginosa strain HSS-6 (Tabel 6). Menurut penelitian sebelumnya, bakteri penghasil SOD diklasifikasikan menjadi empat genus yaitu Pantoea, Escherichia, Basil, dan Pectobacterium dan tiga spesies, yaitu Flavobacterium okeanokoites, Escherichia fergunosii dan E. coli. Dua strain termasuk kelompok Pseudomonas sedangkan lima isolat termasuk kelompok Gamma-Proteobakteri (Indrayati et al., 2011).
Hasil parameter query coverage dan persen maximum identity secara berturut-turut yaitu 96 % dan 100 %. Query coverage yaitu parameter yang digunakan untuk menentukan berapa persentase panjang total sampel yang cukup baik untuk disejajarkan dengan urutan nukleotida dalam bank data NCBI, sedangkan maximum identity merupakan parameter yang digunakan untuk menentukan persentase urutan nukleotida sampel yang mirip dengan urutan nukleotida di bank data. Setiap sekuens memiliki accession number yang berbeda-beda. Accession number yaitu nomor identitas sekaligus nomor akses yang khas untuk setiap sekuens. Menurut Noer (2021) kriteria yang digunakan untuk identifikasi suatu spesies dikatakakan mirip jika tingkat kemiripan sekuensnya lebih dari 99 % atau idealnya lebih dari 99,5 %.
Selain analisis yang sudah dijelaskan sebelumnya, juga dilakukan analisis menggunakan pohon filogenik. Analisis ini bertujuan untuk mengetahui hubungan kekerabatan sesuai dengan nenek moyang dan hubungan evolusioner antar organisme. Hasil dari analisis pohon filogenik (Gambar 3) diperoleh dengan menggunakan software MABL (Methodes Algorithmes Bioinformatique Lirmm) pada web (http://www.phylogeny.fr/simple_phylogeny.cgi
Lima isolat bakteri yang diisolasi dari limbah cair tahu memiliki karakteristik yang berbeda-beda dengan kadar protein total tertinggi mencapai 2,21mg/mL dan nilai persen inhibi-si SOD tertinggi yaitu 87,90 %. Nama spesies bakteri yang memiliki aktivitas SOD tertinggi dapat diindentifikasi dengan metode molekuler PCR 16S rDNA adalah Pseudomonas aeruginosa strain HSS-6.
Puji syukur penulis panjatkan kepada Allah SWT atas segala nikmat dan karunia yang dilimpahkan, serta kepada semua pihak yang telah membantu.
Akihary, C. V., Kolondam, B. J. (2020). Pemanfaatan Gen 16s rRNA sebagai Perangkat Identifikasi Bakteri untuk Penelitian-Penelitian di Indonesia. Pharmacon 9(1): 1622.
Amaliah, Z. Z. N., Bahri, S., Amelia, P. (2018). Isolasi dan Karakterisasi Bakteri Asam Laktat dari Limbah Cair Rendaman Kacang Kedelai. Jurnal Fitofarmaka Indonesia 5(1): 253-257.
Anggraini, N. E. D., Setiawan, N. C. E. (2018). Uji Daya Hambat Antibakteri Ekstrak Kasar Enzim Bromelin dari Bonggol Nanas Terhadap Lactobasillus acidhopi-lus (Ananas comosus (L) Merr) (Doctoral dissertation, Akademi Farmasi Putera Indonesia Malang).
Azhar, F. F., Elvinawati, E., Nurhamidah, N. (2019). Perbandingan sensitivitas nanopartikel perak dengan Reduk-tor Albumin dari Telur Ayam dan Bebek untuk Analisis Merkuri. Alotrop, 3(2).
Brahmachari, G., Laskar, S., Barik, P. (2013). Magnetically Separable MnFe 2 O 4 Nano-Material: An Efficient and Reusable Heterogeneous Catalyst for The Synthesis of 2-Substituted Benzimidazoles and The Extended Synthesis of Quinoxalines at Room Temperature Under Aerobic Conditions. RSC Advances 3(34): 1424514253.
Fitriya, R. T., Ibrahim, M., Lisdiana, L. (2015). Keefektifan metode isolasi DNA kit dan CTAB/NaCl yang dimodifikasi pada Staphylococcus aureus dan Shigelladysen-triae. LenteraBio, 4(1), 87-92.
Frka-Petesic, B., Radavidson, H., Jean, B., Heux, L. (2017). Dynamically Controlled Iridescence of Cholesteric Cellulose Nanocrystal Suspensions Using Electric Fields. Advanced Materials 29(11): 1606208.
Hamidah, M. N., Rianingsih, L., Romadhon, R. (2019). Aktivitas antibakteri isolat bakteri asam laktat dari peda dengan jenis ikan berbeda terhadap E. coli dan S. aureus. Jurnal Ilmu dan Teknologi Perikanan, 1(2), 11-21.
He, F. (2011). BCA (bicinchoninic acid) protein assay. Bioprotocol, e44-e44.
Indrayati, A., Yurina, V., Pitayu, L. A., Retnoningrum, D.
-
S. (2011). 16S rDNA-Based Identification of Novel Superoxide Dismutase Producing Bacteria Isolated from Indonesia. Microbiology Indonesia 5(2): 6-6.
Kementerian Kesehatan RI. (2020). Farmakope Indonesia. Edisi VI. Jakarta: Kementerian Kesehatan RI.
Malik, A., Fitri, N., Muna, F., Soebandrio, A., Karuniawati, A., Sunarno, S. (2014). Metode Cepat Ekstraksi DNA
Corynebacterium Diphtheriae untuk Pemeriksaan PCR. Indonesian Bulletin of Health Research 42(2): 20075.
Noer, S. (2021). Identifikasi Bakteri Secara Molekular Menggunakan 16S rRNA. EduBiologia: Biological Science and Education Journal, 1(1), 1-6.
Nofiani, R., Rizky, R., Briliantoro, R., Ardiningsih, P. (2022). Anti-bacteria and toxicity potential of a rare Actinobacterium Pseudonocardia sp. SM1A, isolated from Mangrove Park, West Kalimantan, Indonesia. Biodiver-sitas Journal of Biological Diversity, 23(1).
Pananjung, A. M. S., Ulfa, E. U., Senjarini, K., Arimurti, S. (2016). Karakterisasi isolat bakteri fibrinolitik WU 021055 asal perairan Pantai Papuma, Jember. Jurnal Bioteknologi Biosains Indonesia (JBBI), 2(1), 1-8.
Qwele, K., Hugo, A., Oyedemi, S. O., Moyo, B., Masika, P. J., Muchenje, V. (2013). Chemical Composition, Fatty Acid Content and Antioxidant Potential of Meat from Goats Supplemented with Moringa (Moringa oleifera) Leaves, Sunflower Cake and Grass Hay. Meat Science 93(3): 455-462.
Rachmawati, D., Monika, N. L. G. M., Masruroh, U. (2018). Potensi abu sekam padi untuk meningkatkan ketahanan oksidatif non-enzimatik dan produksi padi merah pada cekaman kekeringan. Jurnal Agronomi Indonesia (Indonesian Journal of Agronomy), 46(1), 24-32.
Safrida, Y. D., Yulvizar, C., Devira, C. N. (2012). Isolasi dan karakterisasi bakteri berpotensi probiotik pada ikan kembung (Rastrelliger sp.). Depik, 1(3).
Sinaga, M., Nugroho, T. T., Dahliaty, A. (2014). Pemekatan Enzim Selulase Penicillium sp. LBKURCC20 dengan Pengendapan Amonium Sulfat 80 % Jenuh (Doctoral dissertation, Riau University).
Sjafaraenan, S., Lolodatu, H., Johannes, E., Agus, R., Sa-bran, A. (2018). Profil DNA Gen Follicle Stimulating Hormone Reseptor (FSHR) pada Wanita Akne dengan Teknik PCR dan Sekuensing DNA. Bioma: Jurnal Biologi Makassar 3(1): 1-11.
Sugiharto. (1994). Dasar-Dasar Pengolahan Air Limbah. Universitas Indonesia. Jakarta.
Weisburg W.G., Barns S.M., Pelletier D.A., Lane DJ. (1991). 16S Ribosomal DNA Amplification for Phylogenetic Study. Journal of Bacteriol 173(2): 697-703.
Wulandari, Lestari. (2019). Penapisan Bakteri Penghasil Enzim Superoksida Dismurase (SOD) dari Tanah Hutan Mangrove Maron Edupark Semarang. Skripsi. Universitas Setia Budi Surakarta.
PANESTI
100
Discussion and feedback