Potensi Terbentuk Interaksi molekuler pada Fitokimia alami sebagai inhibitor Sap 2 dari Candida albicans: Pendekatan In silico
on

JURNAL FARMASI UDAYANA | pISSN: 2301-7716; eISSN: 2622-4607 | VOL. 11, NO. 2, 2023
https://doi.org/10.24843/JFU.2022.v11.i02.p04
Potensi Terbentuk Interaksi Molekuler pada Fitokimia Alami sebagai Inhibitor Sap 2 Candida albicans: Pendekatan In silico
Gusnia Meilin Gholam1 and I Made Artika1
1 Departemen Biokimia, Fakultas Matematika dan Ilmu Pengetahuan Alam, IPB University, Bogor, 16680
Reception date of the manuscript: 2022-03-11
Acceptance date of the manuscript: 2022-01-20
Publication date: 2023-01-31
Abstract—Candida albicans (C. albicans) is a fungus that can live in the digestive tract, mouth, and vagina. C. albicans is the main cause of vulvovaginal candidiasis (VVC), with a percentage of about 80-90 %. In addition, C. albicans also causes systemic candidiasis, one of the most common bloodstream infections in hospitalized patients worldwide and is associated with a 40-70 % mortality rate. This study aimed to investigate natural phytochemicals potential in inhibiting Sap 2 of C. albicans, thereby reducing its virulence factor. The methods used in this study were Sap 2 protein receptor preparation, test ligand preparation, validation and molecular docking of Sap 2, data analysis and visualization, and ADMET prediction using pkCSM. Results of this study showed that Silibinin has binding energy of 8,658 kcal/mol (closest to that of the crystallographic ligands) and Apigenin (7,608 kcal/mol), Catechin (7,469 kcal/mol), Resveratrol (6,329 kcal/mol), Gallic acid (5,245 kcal/mol) respectively. Visualization of receptors-ligands complex showed data on van der Waals interaction and hydrogen bonds, including hydrogen bond distances. Each test ligand binds with to the catalytic residues and the S2 S3 and S4 substrate binding pockets from Sap 2. This study concludes that there are molecular interactions of the phytochemicals to inhibit Sap 2.
Keywords—Candida albicans, Molecular docking, Sap 2, Natural phytochemicals, In silico
Abstrak—Candida albicans (C. albicans) merupakan jamur yang dapat hidup di saluran pencernaan, mulut, dan vagina. C. albicans menjadi penyebab utama kandidiasis vulvovaginalis (KVV) dengan persentase sekitar 80-90 %. Selain itu, C. albicans juga menyebabkan kandidaisis sistemik, yaitu salah satu infeksi aliran darah yang paling umum pada pasien yang dirawat di rumah sakit di seluruh dunia dan dikaitkan dengan 40-70 % tingkat kematian. Tujuan penelitian ini adalah melakukan investigasi potensi fitokimia alami sebagai inhibitor Sap 2 C. albicans, sehingga mengurangi faktor virulensinya. Metode yang digunakan dalam penelitian ini meliputi Preparasi reseptor protein Sap 2, Preparasi ligan uji, validasi dan penambatan molekuler Sap 2, Analisis data dan visualisasi, Prediksi ADMET menggunakan pkCSM. Hasil penelitian ini menunjukkan bahwa Silibinin mempunyai energi ikatan sebesar 8,658 kkal/mol (paling mendekati energy ikatan ligan hasil kristalografi) dan berturut-turut Apigenin (7,608 kkal/mol), Catechin (7,469 kkal/mol), Resveratrol (6,329 kkal/mol), Gallic acid (5,245 kkal/mol). Visualisasi kompleks reseptor-ligan menghasilkan data tentang gaya van der Waals, dan ikatan hidrogen, termasuk jarak ikatan hidrogen. Setiap ligan uji berikatan dengan residu katalitik, kantong pengikat substrat S2, S3, dan S4 dari Sap 2. Kesimpulan dari penelitian ini yaitu terdapat interaksi molekuler dari fitokimia untuk dapat menghambat Sap 2.
Kata Kunci—Candida albicans, Penambatan molekular, Sap 2, Fitokimia alami, In silico
Candida albicans (C. albicans) merupakan jamur yang dapat hidup di saluran pencernaan (Monroy-Pérez et al., 2013), mulut (Li et al., 2014), dan vagina (Monroy-Pérez et al., 2013). C. albicans dapat menyebabkan infeksi mulut dan vagina serta penyakit sistemik. Menurut Nugraheni et al. (2015), C. albicans menjadi penyebab utama kandidiasis vul-vovaginalis (KVV) dengan persentase sekitar 80-90 %. KVV
Penulis koresponden: Gusnia Meilin Gholam,
Email: gusnia_26@apps.ipb.ac.id
adalah salah satu infeksi paling umum yang dapat menimpa wanita di usia reproduksi. Bahkan diperkirakan sekitar 75 % wanita akan mengalami setidaknya sekali selama masa hidup mereka (Monroy-Pérez et al., 2013; Willems et al., 2017). Laporan menunjukkan bahwa diperkirakan sekitar 5-8 % dari seluruh wanita mengalami infeksi berulang (Willems et al., 2017). Selain itu, C. albicans juga menyebabkan kandidia-sis sistemik, yaitu salah satu infeksi aliran darah yang paling umum pada pasien yang dirawat di rumah sakit di seluruh dunia dan dikaitkan dengan 40-70 % dari tingkat kematian (Shukla & Rohatgi, 2020). C. albicans mempunyai beberapa faktor virulensi yang terlibat dalam pembentukan hifa, pergantian fenotip, adhesi sel, dan produksi enzim hidrolitik
ekstraseluler (Calugi et al., 2012; Monroy-Pérez et al., 2013; Shukla & Rohatgi, 2020; Willems et al., 2017). Secreted aspartic proteinase (Sap) adalah enzim yang disekresikan oleh C. albicans dan dikodekan oleh keluarga Sap yang terdiri dari Sap 1 hingga Sap 10. Masing-masing Sap tersebut mempunyai peran penting pada mukosa (Sap1-Sap3) dan infeksi sistemik (Sap4-Sap6) (Monroy-Pérez et al., 2013), sehingga apabila ditemukan suatu senyawa yang dapat menghambat aktvitas Sap, tentu dapat mengurangi faktor virulensi dari jamur ini (Santos et al., 2021).
Pengobatan berbagai penyakit dapat menggunakan tanaman obat sebagai sumber obat alternatif (Shakya, 2019). Studi eksperimental menunjukkan bahwa tanaman obat mempunyai metabolit sekunder yang dikenal sebagai fitokimia. Saat ini, fitokimia sedang banyak diuji dalam aktvitas pengobatan seperti anti-oksidan, anti-inflamasi, anti-mutagenik, anti-alergi, anti-bakteri, antivirus, neuroprotektif dan hepato-protektif (Shakya, 2019). Berdasarkan penjelasan diatas maka tujuan dari penelitian ini adalah melakukan investigasi potensi fitokimia alami sebagai inibitor Sap 2 C. albicans, dalam upaya mengurangi faktor virulensinya melalui pendekatan in silico.
Bahan dan Alat
Alat-alat yang digunakan dalam penelitian ini adalah laptop hp 250 G5 Notebook PC dengan sistem operasi Windows 10 Professional 64-bit, spesifikasi prosessor Intel ® Core TM i3-6006U, dan 4,00 GB RAM. Perangkat lunak (software) yang digunakan untuk melakukan penelitian yaitu YASARA structure (YASARA Biosciences GmbH) dan BIOVIA Discovery Studio. Bahan yang digunakan dalam penelitian ini adalah reseptor Sap 2 dan ligan uji Gallic acid, Catechin, Resveratrol, Apigenin, dan Silibinin (Tabel 1).
Metode
Preparasi reseptor protein Sap 2
Struktur tiga dimensi (3D) Secreted aspartic proteinase (Sap) 2 dari C. albicans diunduh dari database RCSB PDB dengan kode 1EAG dalam format (.pdb). Struktur diprepara-si menggunakan bantuan perangkat lunak YASARA structure. Sap 2 dalam database PDB mempunyai satu Chain yaitu Chain A. Struktur Sap 2 dengan menggunakan Chain A dipreparasi dengan menghapus beberapa bagian yang tidak diperlukan dalam protokol penambatan (Agistia et al., 2013). Penelitian ini menghapus molekul air dan menambahkan atom hidrogen. Penambahan atom hidrogen secara manual disebabkan karena resolusi struktur kristal tidak mampu memprediksi keberadaan hidrogen (Agistia et al., 2013; Etsè et al., 2021). File hasil preparasi disimpan dalam format (.pdb). Ligan hasil kristalografi yang tertambat bersama dengan Sap 2 yaitu A70450, dibiarkan masih melekat dengan reseptor, hal ini dimaksudkan untuk membantu memudahkan proses penentuan Gridbox saat validasi dilakukan.
Preparasi ligan uji
Struktur tiga dimensi (3D) ligan uji dari Gallic acid, Catechin, Resveratrol, Apigenin, dan Silibi-nin diunduh dari database PubChem melalui laman https://pubchem.ncbi.nlm.nih.gov/ dalam format (.sdf). Ligan uji dipreparasi menggunakan bantuan perangkat lunak
YASARA structure dengan melakukan energi minimisasi pada ligan uji (Cojocaru et al., 2019). Ligan ditambahkan hidrogen (Trivedi, Vaidya, Patel, Prajapati, Bhatt, 2020; Gholam, 2022).
Validasi dan penambatan molekuler Sap 2
Secreted aspartic proteinase (Sap) 2 mempunyai struktur kompleks yang telah dikristalkan bersama A70450. Adanya ligan hasil kristalografi tersebut, tentu membantu memudahkan ketika validasi. Validasi dengan menentukan extend untuk Gridbox yang menentukan ruang gerak didalamnya saat penambatan sedang berjalan (Frimayanti et al., 2021). Validasi Gridbox menggunakan YASARA structure. Parameter validasi yang digunakan disebut root mean square deviation (RMSD). Skor RMSD yang ditentukan dan dapat dinyatakan valid apabila memilikir skor <2 Å (kurang dari 2 Å) (Mu-latsari et al., 2019).
Setelah Gridbox ditemukan, maka penambatan dapat dilakukan. Penambatan dengan ligan uji terhadap reseptor target menggunakan YASARA structure dengan menggunakan play macro dock_run.mcr (Ali et al., 2020; Srivastava et al., 2018; Wang et al., 2019). Metode penambatan menggunakan VINA (Wang et al., 2019). Perhitungan penambatan yaitu secara otomatis dilakukan dengan bantuan perangkat lunak YASARA structure dan Force field (Cojocaru & Cli-ma, 2020). Force field yang digunakan dalam penelitian ini adalah Amber14 (Ali et al., 2020). Penambatan diatur dalam runs=25 dan kemudian hasil telah otomatis diurutkan berdasarkan energi ikatan paling tinggi (kkal/mol) Ali et al. (2020); Cojocaru & Clima (2020); Wang et al. (2019).
Analisis data dan visualisasi
Visualisasi semua interaksi baik secara 2D maupun 3D menggunakan Discovery Studio (Kumar et al., 2020). Prediksi ADMET menggunakan pkCSM Prediksi ADMET (absorption, Distribution, Metabolism, excretion, toxicity) dilakukan menggunakan pkCSM yang diakses melalui laman web http://biosig.unimelb.edu.au/pkcsm/ (Ferrari, 2021; Pires et al., 2015).
Preparasi Reseptor dan ligan
Reseptor yang telah dilakukan protokol preparasi, kemudian disimpan dalam format .pdb untuk dilanjutkan tahap penambatan. Ligan uji yang telah dipreparasi, kemudian disimpan dalam format (_ligand.sdf).
Validasi dan penambatan molekuler Sap 2
Hasil validasi dalam penelitian ini yaitu didapatkan RMSD sebesar 0.00 Å pada Gridbox 2.5 Å. Ukuran tersebut digunakan untuk tahap penambatan ligan uji terhadap reseptor target. Penambatan kelima ligan uji menggunakan perangkat lunak YASARA structure dengan dock_run serta beberapa pengaturan yang digunakan yaitu 25 runs dan Am-ber14. Hasil penambatan meliputi energi ikatan, konstanta disosiasi, dan kontak residu asam amino. Hasil dapat dilihat pada Tabel 2.
Analisis data dan visualisasi
Analisis dan visualisasi dilakukan menggunakan bantuan perangkat lunak Discovery Studio. Hasil yang dapat diketahui dari Discovery Studio yaitu berupa 2D, 3D, jarak ika-
Tabel 1: DAFTAR LIGAN UJI DARI FITOKIMIA ALAMI (SHAKYA, 2019)
|
CID Ligan |
Sumber paling kaya |
|
370 Gallic acid 9064 Catechin 445154 Resveratrol 5280443 Apigenin 31553 Silibinin |
Kacang gallnut, Suren merah, dan Terminalia belerica Tanaman teh, Anggur, Bluberi, dan Apel Vitis vinifera dan Anggur merah Articok, Jeruk, dan Seledri Silymarin marimun |
Tabel 2: HASIL PENAMBATAN MOLEKULER DAN ANALISIS KONTAK RESIDU ASAM AMINO
|
Ligan |
Energi ikatan (kkal/mol) |
Konstanta disosiasi [pM] |
Van der waals |
Ikatan hidrogen |
Jarak ikatan hidrogen (Å) |
|
Gallic acid |
5.245 |
143009216 |
Ile30, Ile119, Ile123, Asp32, |
Gly85 |
2.24, 2.86, |
|
Asp218, Gly34, Ser35, Tyr84 |
2.95, 3.08 | ||||
|
Catechin |
7.469 |
3350814.75 |
Asp86, Tyr225, Asn301, Ala303, |
Asp86 |
2.48 |
|
Ile305, Gly85, Thr221, Gly220, Asp32, Ile123, Ile119 |
Asp218 |
2.59 | |||
|
Tyr51, Ser88, Ser118, Tyr84, | |||||
|
Silibinin |
8.658 |
450403.5938 |
Ile305, Thr221, Tyr225, Thr222, Asp32, Ile123, Thr13, Ile30, Asp120 |
Asp218 |
2.65 |
|
Gly220 |
2.85 | ||||
|
6.329 |
22949936 |
Tyr84, Ser35, Ile82, Ala133, |
Asp32 |
2.60 | |
|
Resveratrol |
Asn131 |
2.57 | |||
|
Gly34, Gly85, Ile305 |
Gly220 |
2.49 | |||
|
Thr221 |
2.56 | ||||
|
Asp120, Ile119, Asp86, Gly85, | |||||
|
Apigenin |
7.608 |
2650098.75 |
Tyr84, Asp218, Thr221, Ile123, Thr13, Val12 |
Asp32 |
2.52 |
|
Gly34 |
2.16 | ||||
|
A70450 (ligan hasil kristalografi) |
Ile123, Ala303, Asp32, Gly83, |
Gly85 |
2.41 | ||
|
9.387 |
131594.5156 |
Ser35, Ser36, Glu132, Ala133, |
Asp218 |
2.16 | |
|
Asn131, Leu216, Arg195, Glu193, |
Gly220 |
2.11 | |||
|
Thr13, Gln54, Thr50, Tyr51, Asn301 |
Thr221 |
2.39, 2.92 | |||
|
Thr222 |
2.20, 2.64 |
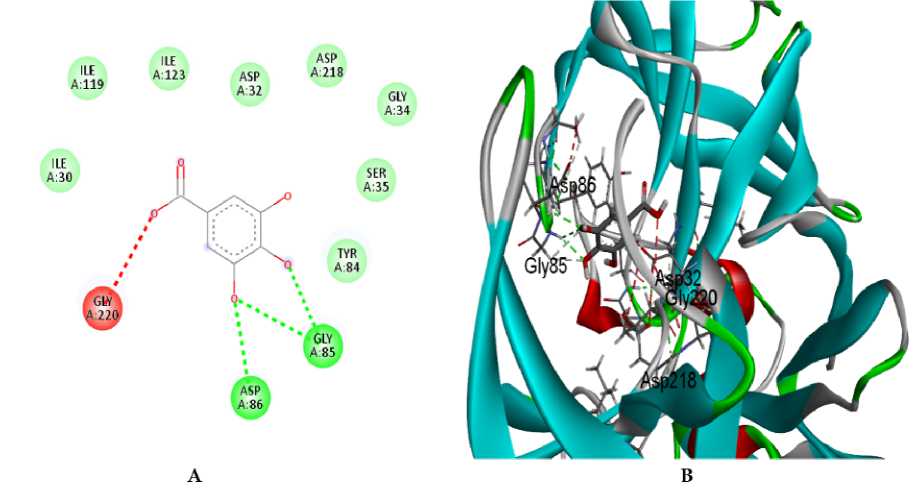
Gambar. 1: A) Visualisasi interaksi 2D ligan uji Gallic acid yang tertambat pada Sap 2. (B) Visualisasi interaksi 3D ligan uji Gallic acid yang mempunyai kontak dengan residu katalitik, kantong pengikat substrat S2 dan S3 pada Sap 2. Visualisasi menggunakan Discovery Studio.
Gambar. 2: (A) Visualisasi interaksi 2D ligan uji Catechin yang tertambat pada Sap 2 . (B) Visualisasi interaksi 3D ligan uji Catechin yang mempunyai kontak dengan residu katalitik, kantong pengikat substrat S2 dan S3 pada Sap 2. Visualisasi menggunakan Discovery Studio.
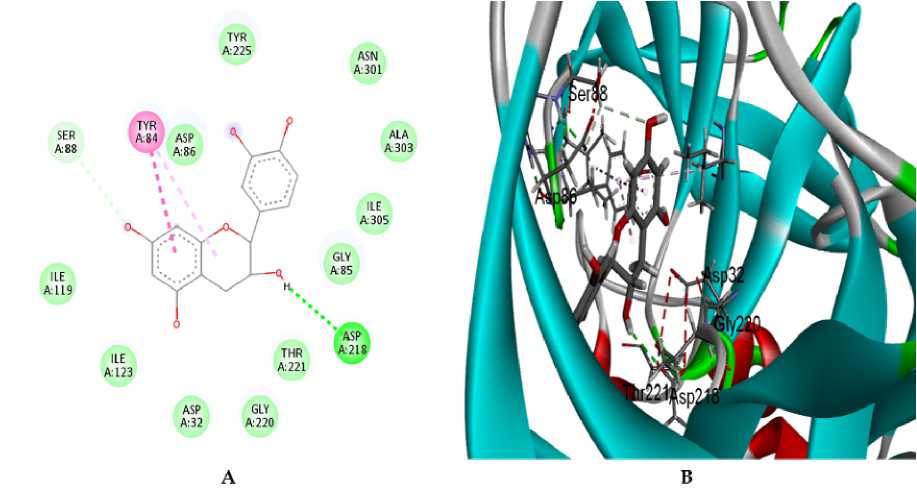
Gambar. 3: (A) Visualisasi interaksi 2D ligan uji Silibinin yang tertambat pada Sap 2. (B) Visualisasi interaksi 3D ligan uji Silibinin yang mempunyai kontak dengan residu katalitik dan kantong pengikat substrat S3 pada Sap 2. Visualisasi menggunakan Discovery Studio.
tan, van der waals, dan ikatan hidrogen. Hasil disajikan pada Tabel 2.
Prediksi ADMET menggunakan pkCSM
Hasil prediksi ADMET (absorption, Distribution, Metabolism, excretion, toxicity) disampaikan pada Tabel 3.
Penambatan molekuler digunakan untuk memprediksi konformasi kompleks reseptor-ligan. Umumnya, reseptor dapat berupa protein atau molekul asam nukleat, sedangkan li-
gan dapat berupa protein lain, molekul lain atau bahkan ion logam (Ali et al., 2020). Selain itu, penambatan molekuler juga merupakan metode komputasi yang digunakan dalam menghitung afinitas ikatan reseptor-ligan dan memperkirakan tempat ikatan senyawa dengan protein target (Herman, 2019). Penelitian ini menggunakan protein target Sap 2 dan ligan uji dari fitokimia alami (Tabel 1). Penggunaan ligan uji ini karena prediksi Lipinski Rule of Five yang telah dilakukan oleh Shakya (2019) menunjukkan bahwa kelima li-gan uji dari fitokimia alami ini lolos dalam prediksi Lipinski. Prediksi Lipinski membantu dalam menentukan suatu sen-
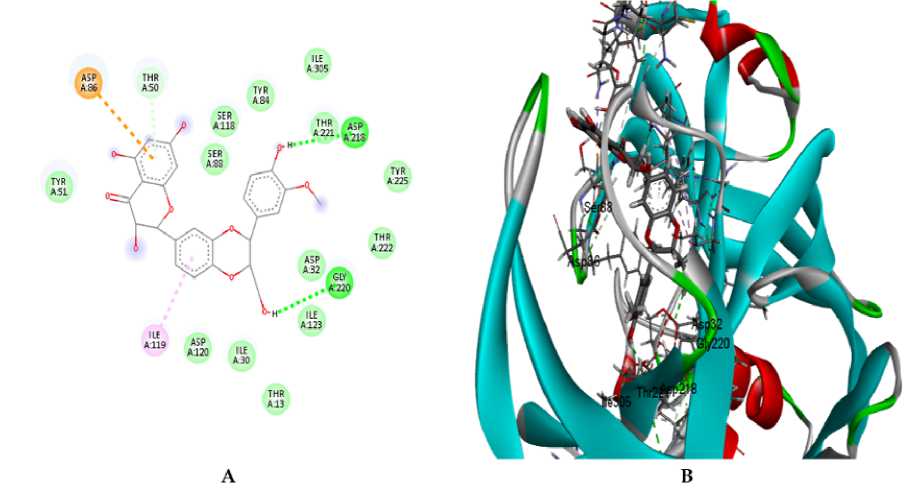
Gambar. 4: A) Visualisasi interaksi 2D ligan uji Resveratrol yang tertambat pada Sap 2 (A). (B) Visualisasi interaksi 3D ligan uji Resveratrol yang mempunyai kontak dengan residu katalitik dan kantong pengikat substrat S2 dan S3 pada Sap 2. Visualisasi menggunakan Discovery Studio.
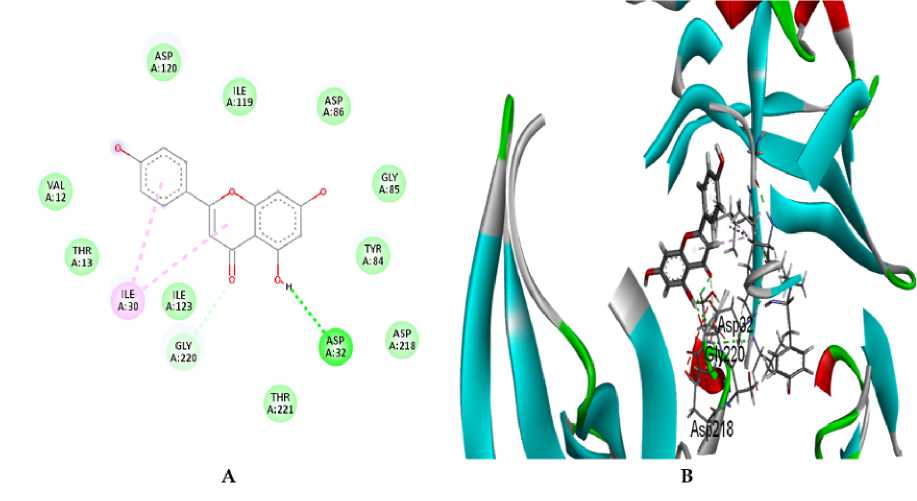
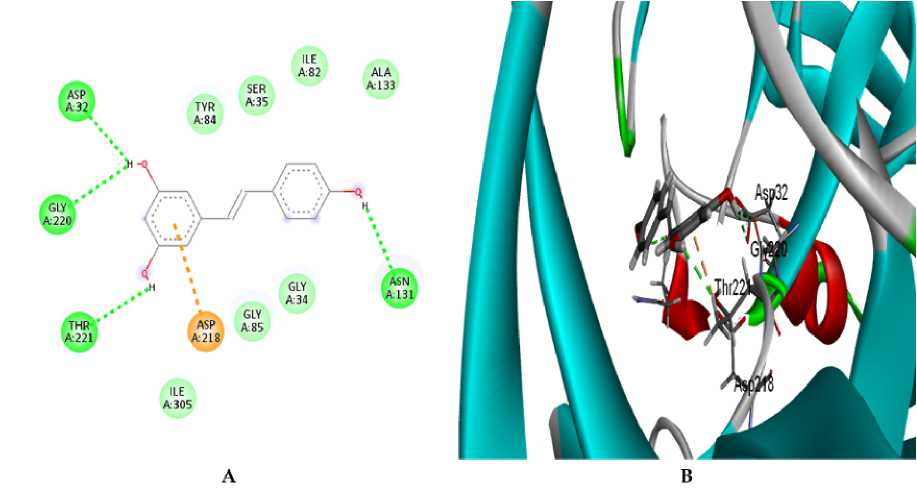
Gambar. 5: (A) Visualisasi interaksi 2D ligan uji Apigenin yang tertambat pada Sap 2. (B) Visualisasi interaksi 3D ligan uji Apigenin yang mempunyai kontak dengan residu katalitik dan kantong pengikat substrat S3 pada Sap 2. Visualisasi menggunakan Discovery Studio.
yawa apakah dapat dijadikan sebagai obat atau tidak dengan mengikuti kelima aturan yang berlaku pada Lipinski Shakya (2019). Kelima ligan uji tersebut yaitu Gallic acid, Catechin, Resveratrol, Apigenin, dan Silibinin. Kelima ligan tersebut bisa didapat dari beberapa sumber yang kaya akan keberadaannya pada beberapa tanaman ataupun buah (dapat dilihat pada Tabel 1).
Validasi penambatan molekuler
Selain ligan uji yang ditambatkan terhadap Sap 2, A70450 juga dilakukan penambatan ulang untuk validasi dan sebagai
perbandingan (patokan) seperti dalam penelitian Santos et al. (2021).
A70450 digunakan untuk validasi dalam penentuan Gridbox. Gridbox berfungsi untuk menentukan ukuran cube pada daerah sisi aktif dari reseptor yang akan ditambatkan, sehingga harapannya ligan dapat mempunyai kontak dengan sisi aktif suatu reseptor (Mulatsari et al., 2019; Sari et al., 2020). Validasi penambatan menggunakan metode penambatan ulang Sari et al. (2020), ligan hasil kristalografi A70450 dengan reseptor Sap 2 yang telah dipreparasi. RMSD dinyatakan valid karena mempunyai skor <2 Å dan ukuran grid-
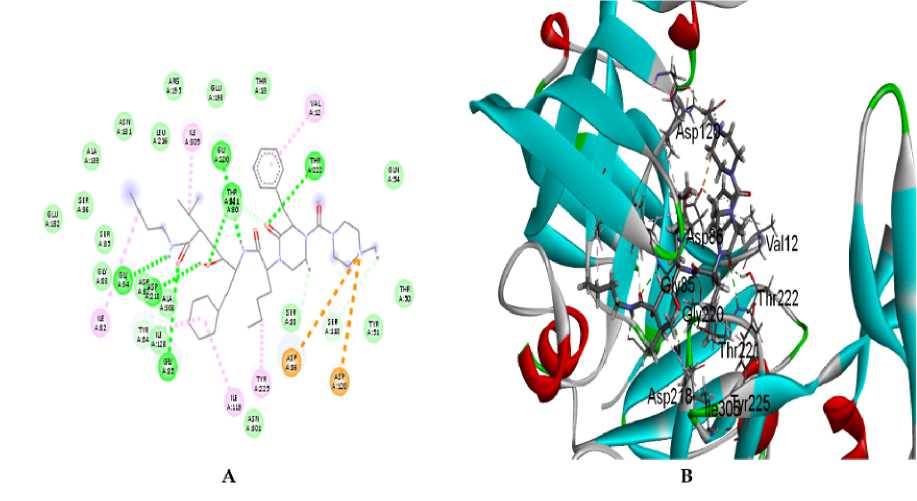
Gambar. 6: (A) Visualisasi interaksi 2D ligan hasil kristalografi A70450 yang tertambat pada Sap 2. (B) Visualisasi interaksi 3D ligan hasil kristalografi A70450 yang mempunyai kontak dengan residu katalitik dan kantong pengikat substrat S2, S3 dan S4 pada Sap 2.
Visualisasi menggunakan Discovery Studio.
Tabel 3: DAFTAR LIGAN UJI DARI FITOKIMIA ALAMI (SHAKYA, 2019)
|
ADMET |
Ligan uji (CID) | ||||
|
Gallic acid |
Catechin |
Silibinin |
Resveratrol |
Apigenin | |
|
Intestinal absorption (human) |
43.374 % |
68.829 % |
61.861 % |
90.935 % |
93.25 % |
|
Skin permeability |
-2.735 log Kp |
-2.735 log Kp |
-2.735 log Kp |
-2.737 log Kp |
-2.735 log Kp |
|
VDss (human) |
-1.855 log L/kg |
1.027 log L/kg |
0.369 log L/kg |
0.296 log L/kg |
0.822 log L/kg |
|
BBB permeability |
-1.102 log BB |
-1.054 log BB |
-1.207 log BB |
-0.048 log BB |
-0.734 log BB |
|
CYP2D6 substrate |
Tidak |
Tidak |
Tidak |
Tidak |
Tidak |
|
CYP2D6 inhibitor |
Tidak |
Tidak |
Tidak |
Tidak |
Tidak |
|
Renal OCT2 substrate |
Tidak |
Tidak |
Tidak |
Tidak |
Tidak |
|
Total clearance |
0.518 log |
0.183 log |
-0.103 log |
0.076 log |
0.566 log |
|
ml/min/kg |
ml/min/kg |
ml/min/kg |
ml/min/kg |
ml/min/kg | |
|
AMES toxicity |
Tidak |
Tidak |
Tidak |
Ya |
Tidak |
box dapat digunakan dalam penambatan dengan ligan uji (Frimayanti et al., 2021; Mulatsari et al., 2019; Sari et al., 2020). Validasi dilakukan menggunakan bantuan perangkat lunak YASARA structure. Kontak residu dari hasil penambatan ulang ligan hasil kristalografi A70450 antara lain Val12, Thr13, Ile30, Asp32, Gly34, Ser35, Ser36, Thr50, Tyr51, Gln54, Ile82, Gly83, Tyr84, Gly85, Asp86, Ser88, Ser118, Ile119, Ile123, Asn131, Glu132 , Ala133, Glu193, Arg195, Leu216, Asp218, Gly220, Thr221, Thr222, Tyr225, Asn301, Ala303, Ile305. Keseluruhan residu asam amino dari A70450 juga dapat dijadikan patokan dalam analisis kontak residu asam amino pada ligan uji (Santos et al., 2021). Dari hasil validasi dalam penelitian ini diketahui bahwa A70450 mempunyai kontak dengan residu katalitik (Asp32, Asp218), S2 (Gly85, Asp86, Thr221, Tyr225, Asn301, Ala303, Ile305), S3 (Val12, Thr13, Asp86, Ser88, Ser118, Gly220, dan S4 (Val12, Thr222, Tyr225).
Interaksi, analisis dan visualisasi penambatan molekuler Sap 2
Penambatan dilakukan menggunakan bantuan perangkat lunak YASARA structure. Hasil penambatan menggunakan YASARA structure dapat dilihat pada file log. Hasil penambatan diurutkan berdasar pada energi ikatan (kkal/mol) dan konstanta disosiasi [pM]. Selain itu, terdapat kontak residu asam amino secara luas yang dihasilkan oleh penambatan li-gan uji (Srivastava et al., 2018).
Secreted aspartic proteinase (Sap 2) dari Candida albicans yang dikomplekskan dengan A70450 yang dapat diakses melalui database PDB mempunyai resolusi 2.10 Å (Cutfield et al., 1995). Sap 2 mempunyai sisi aktif berupa residu katalitik dan beberapa kantong pengikat substrat. Residu katalitik Sap 2 terletak pada residu asam amino Asp32 dan Asp218 (Borelli et al., 2008; Cutfield et al., 1995; Santos et al., 2021). Borelli et al. (2008) dalam penelitiannya membantu membagi kantong pengikat substrat pada susunan S2, S3 dan S4. Residu asam amino pada kantong S2 terdiri dari Gly85, Asp86, Thr221, Tyr225, Asn301, Ala303, Ile305, kantong S3 terdiri dari Val12, Thr13, Asp86, Ser88, Ser118,
Asp120, Gly220, dan kantong S4 terdiri dari Val12, Thr222, Ile223, Tyr225, Gln295, Leu297, Asp299. Dengan demikian, telah diketahui residu-residu penting dari Sap 2.
A70450 sebagai ligan hasil kristalografi mempunyai energi ikatan tertinggi yaitu sebesar 9.387 kkal/mol. Energi ikatan tersebut lebih tinggi daripada ligan uji. Urutan energi ikatan dari ligan uji yang mendekati energi ikatan ligan hasil kristalografi A70450 yaitu Silibinin 8.658 kkal/mol, Api-genin 7.608 kkal/mol, Catechin 7.469 kkal/mol, Resveratrol 6.329 kkal/mol, dan Gallic acid 5.245 kkal/mol. Urutan ini didapat karena berdasar pada pernyataan bahwa Ligan yang mempunyai energi ikatan lebih positif menunjukkan adanya interaksi yang lebih kuat dengan reseptor (Ali et al., 2020; Srivastava et al., 2018). Energi ikatan membantu mempengaruhi efisiensi pengikatan ligan dengan reseptor (Nallal et al., 2021). Energi ikatan dirangkum pada Tabel 2.
Hasil penambatan menunjukkan bahwa seluruh Ligan uji maupun ligan hasil kristalografi melalui visualisasi menggunakan Discovery Studio terlihat membentuk van der waals. Adapun rincian residu asam amino yang membentuk van der waals yaitu Gallic acid mempunyai kontak residu asam amino pada Ile30, Ile119, Ile123, Asp32, Asp218, Gly34, Ser35, Tyr84 (Gambar 1A). Catechin pada residu Asp86, Tyr225, Asn301, Ala303, Ile305, Gly85, Thr221, Gly220, Asp32, Ile123, Ile119 (Gambar 2A). Resveratrol pada residu asam amino Tyr84, Ser35, Ile82, Ala133, Gly34, Gly85, Ile305 (Gambar 4A). Silibinin pada residu asam amino Tyr51, Ser88, Ser118, Tyr84, Ile305, Thr221, Tyr225, Thr222, Asp32, Ile123, Thr13, Ile30, Asp120 (Gambar 3A) dan Apigenin pada residu asam amino Asp120, Ile119, Asp86, Gly85, Tyr84, Asp218, Thr221, Ile123, Thr13, Val12 (Gambar 5A). Sedangkan pada ligan hasil kristalografi terbentuk pada residu asam amino Ile123, Ala303, Asp32, Gly83, Ser35, Ser36, Glu132, Ala133, Asn131, Leu216, Arg195, Glu193, Thr13, Gln54, Thr50, Tyr51, Asn301 (Gambar 6A). Peran van der waals dalam pengikatan reseptor-ligan, biasanya berperan mendukung dan menstabilkan pose (Hariono & Rollando, 2016).
Diketahui bahwa jumlah terbentuk pada ikatan hidrogen dapat menentukan kekuatan ikat dalam kompleks reseptor-ligan (Nallal et al., 2021). Hasil analisis memperlihatkan Gallic acid membentuk ikatan hidrogen pada residu asam amino kantong S2 Gly85 dan Asp86 dengan lima ikatan terbentuk. Catechin pada residu katalitik Asp218 dengan satu ikatan terbentuk. Silibinin membentuk ikatan hidrogen pada residu katalitik Asp218 dan kantong S3 Gly220, masing-masing membentuk satu ikatan. Resveratrol membentuk ikatan hidrogen pada residu katalitik Asp32, kantong S3 Gly220, kantong S2 Thr221 dan residu lain Asn131, masing-masing mempunyai satu ikatan. Apigenin membentuk ikatan hidrogen pada residu katalitik Asp32 dengan satu ikatan. Ligan hasil kristalografi mempunyai jumlah terbanyak pada ikatan hidrogen. Detail residu asam amino yang membentuk ikatan tersebut yaitu katalitik Asp218, kantong S2 Gly85 dan Thr221, kantong S3 Gly220, dan kantong S4 Thr222. Seluruh analisis disajikan pada Tabel 2.
Prediksi ADMET (absorption, Distribution, Metabolism, excretion, toxicity)
Pengembangan obat mempunyai tingkat permasalahan yang cukup tinggi seperti sifat farmakokinetik dan keama-
nan yang kurang baik, menjadi rintangan dalam penemuan obat. Salah satu pendekatan melalui komputasi ini, diperkirakan membantu meminimalkan resiko yang tidak diinginkan. Prediksi melalui pkCSM, diketahui bekerja cukup baik dibandingkan metode farmakokinetik lain yang tersedia secara bebas untuk diakses. Server pkCSM untuk prediksi farmako-kinetik molekul kecil (Ferrari, 2021). Tabel 3 menunjukkan analisis prediksi yang disediakan oleh pkCSM. Setiap ligan uji mempunyai absorpsi diatas 30 % dengan rincian Resveratrol (90.935 %), Apigenin (93.25%), Gallic acid (43.374%), Silibinin (61.861 %), dan Catechin (68.829 %). Pires et al., (2015) menjelaskan bahwa suatu senyawa yang mempunyai absorpsi dibawah 30 % memiliki absoprsi yang sangat rendah.
Menurut (Hardjono, 2017) senyawa yang mempunyai permeabilitas kulit yang relatif rendah bila memiliki nilai log Kp >-2.5. Berdasarkan Tabel 3, hasil prediksi menunjukkan nilai dari setiap senyawa mempunyai kesamaan pada Skin permeability (log Kp) sebesar -2.735 log Kp. Hasil itu menunjukkan bahwa senyawa-senyawa pada penelitian ini diprediksi mendekati permeabilitas kulit yang rendah (Pires et al., 2015).
Klasifikasi nilai log VDss yang tinggi pada 2.81 L/kg (log VDss>0,45) dan nilai log VDss rendah bila Log 0.71 L/kg (log VDss <-0,15) (Fida et al., 2021; Hardjono, 2017; Pires et al., 2015). VDss merupakan volume yang menunjukkan nilai suatu dosis total obat yang didistribusikan secara menyeluruh dan mempunyai konsestrasi seperti plasma darah (Fida et al., 2021; Pires et al., 2015).
Senyawa yang tidak menembus sawar otak yaitu Resveratrol dan Apigenin dengan masing-masing nilai -0.048 log BB dan -0.734 log BB. Selain kedua senyawa tersebut, senyawa lain masih tergolong tidak dapat menembus sawar karena tidak lebih dari nilai log BB 0.3. hal ini dijelaskan oleh Fida et al. (2021) bahwa suatu senyawa dapat menembus sawar otak bila nilai log BB >0.3 dan tidak dapat menembus bila log BB <-1 (Pires et al., 2015). Blood Brain Barier (BBB) diperlukan untuk mengetahui kemampuan suatu obat menembus sawar otak, serta membantu mengurangi efek samping, toksisitas dan bahkan dapat meningkatkan khasiat obat yang mempunyai tujuan pengobatan pada otak (Fida et al., 2021; Pires et al., 2015).
Berdasarkan sajian Tabel 3, seluruh senyawa masuk dalam kategori tidak pada CYP2D6 substrate dan CYP2D6 inhibitor. Dengan demikian, seperti dalam penelitian Hardjono (2017) menjelaskan bahwa hal tersebut tidak mempengaruhi atau menghambat enzim CYP2D6, sehingga dapat diprediksi senyawa ini cenderung dimetabolisme oleh enzim P450. Untuk memprediksi proses ekskresi suatu senyawa dapat dilakukan dengan mengukur tetapan Renal Organic Cation Transporter 2 (OCT2) substrate dan Total clearance (Hardjono, 2017).
Total clearance diukur melalui konstanta proporsional CLtot (metabolisme di hati dan empedu) dan renal clearance (ekskresi melalui ginjal) (Tabel 3) (Pires et al., 2015).
Renal Organic Cation Transporter 2 (OCT2) substrate (Tabel 3), OCT2 merupakan transporter yang terletak pada ginjal yang memegang peran penting dalam disposisi dan clearance obat-obatan dan senyawa endogen (Hardjono, 2017).
Terkait Renal Organic Cation Transporter 2 (OCT2)
substrate, Prediktor akan menguji apakah molekul tersebut mungkin berupa substrat OCT2 (Pires et al., 2015). Substrat OCT2 berpotensi menimbulkan interaksi sampingan bila diberikan secara bersama dengan inhibitor OCT2 (Hardjono, 2017).
Hardjono, (2017) menjelaskan bahwa Total clearance merupakan kombinasi dari hepatic clearance (metabolisme di hati dan empedu) dan renal clearance (ekskresi melalui ginjal). Hal tersebut eratkaitannya dengan bioavailabilitas dan penting untuk menentukan tingkatan dosis dalam mencapai konsestrasi steady-state.
Toksisitas AMES membantu memprediksi apakah suatu senyawa bersifat positif AMES dan juga mutagenik (Tabel 3) (Pires et al., 2015).
Hasil dari peneltian ini adalah ligan uji dari Fitokimia alami mempunyai energi ikatan yang cukup besar dan mendekati ligan hasil kristalografi. Selain itu, setiap ligan uji menghasilkan konstanta disosiasi, van der waals, ikatan hidrogen dan jarak ikatan hidrogen (Tabel 2). Keseluruhan ligan uji mempunyai ikatan hidrogen dengan kontak residu asam amino penting dari Sap 2. Dalam penelitian ini juga membantu melakukan prediski ADMET untuk masing-masing ligan uji.
Agistia, D. D., Purnomo, H., Tegar, M., & Nugroho, A. E.
-
(2013) . Interaksi Senyawa Aktif Dari Aegle marmelos Correa Sebagai Anti Inflamasi Dengan Reseptor COX-1 Dan COX-2. Traditional Medicine, 18(2), 80–87.
Ali, N. S. M., Salleh, A. B., Leow, T. C., Rahman, R. N. Z.
R. A., & Ali, M. S. M. (2020). The Influence of Calcium toward Order/Disorder Conformation of Repeat-in-Toxin (RTX) Structure of Family I.3 Lipase from Pseudomonas fluorescens AMS8. Toxins, 12(579), 1–14.
Borelli, C., Ruge, E., Jung, H. L., Schaller, M., Vogelsang, A., Monod, M., Korting, H. C., Huber, R., & Maskos, K. (2008). X-ray structures of Sap1 and Sap5: Structural comparison of the secreted aspartic proteinases from Candida albicans. Proteins: Structure, Function and Genetics, 72(4), 1308–1319.
Calugi, C., Trabocchi, A., De Bernardis, F., Arancia, S., Navarra, P., Cauda, R., Cassone, A., & Guarna, A. (2012). Bicyclic peptidomimetics targeting secreted aspartic protease 2 (SAP2) from Candida albicans reveal a constrained inhibitory chemotype. Bioorganic and Medicinal Chemistry, 20(24), 7206–7213.
Cojocaru, C., & Clima, L. (2020). Polymer assisted ultrafiltration of AO7 anionic dye from aqueous solutions: Experimental design, multivariate optimization, and molecular docking insights. Journal of Membrane Science, 604(February), 118054.
Cutfield, S. M., Dodson, E. J., Anderson, B. F., Moody, P., Marshall, C. J., Sullivan, P. A., & Cutfield, J. F. (1995). The crystal structure of a major secreted aspartic proteinase from Candida albicans in complexes with two inhibitors. Structure, 3(11), 1261–1271.
Etsè, K. S., Etsè, K. D., Nyssen, P., & Mouithys-Mickalad, A. (2021). Assessment of anti-inflammatory-like, antioxidant activities and molecular docking of three alkynyl-substituted 3-ylidene-dihydrobenzo[d]isothiazole 1,1-dioxide derivatives. Chemico-Biological Interactions, 344(April).
Ferrari, I. V. (2021). Open access in silico tools to predict the ADMET profiling and PASS (Prediction of Activity Spectra for Substances of Bioactive compounds of Garlic (Allium sativum L.). BioRxiv.
Fida, S., Dewi, A. R., & Damayanti, D. S. (2021). Studi In Silico Senyawa Aktif Daun Sirsak (Annona muricata L .) pada Aldose Reductase dan Glutathione Reductase untuk Menghambat Katarak Diabetik. Jurnal Kedokteran Komunitas, 9(2), 1–14.
Frimayanti, N., Djohari, M., & Khusnah, A. N. (2021). Mo-lekular Docking Senyawa Analog Kalkon sebagai Inhibitor untuk Sel Kanker Paru-Paru A549 ( Molecular Docking for Chalcone Analogue Compounds as Inhibitor for Lung Cancer A549 ). Jurnal Ilmu Kefarmasian Indonesia, 19(1), 87–95.
Hardjono, S. (2017). Prediksi Sifat Farmakokinetik, Toksi-sitas dan Aktivitas Sitotoksik Turunan N-Benzoil-N’-(4-fluorofenil)tiourea sebagai Calon Obat Antikanker melalui Pemodelan Molekul. Jurnal Ilmu Kefarmasian Indonesia, 14(2), 246–255.
Hariono, M., & Rollando (2016). MOLECULAR DOCKING OF COMPOUNDS FROM Chaetomium Sp. AGAINST HUMAN ESTROGEN RECEPTOR ALPHA IN SEARCHING ANTI BREAST CANCER. JURNAL FARMASI SAINS DAN KOMUNITAS, 13(1), 35–43.
Herman, R. (2019). Studi in Silico Lima Senyawa Aktif Sebagai Penghambat Protein Virus Dengue. Jurnal Kefarma-sian Indonesia, 9(1), 40–47.
Li, W., Yu, D., Gao, S., Lin, J., Chen, Z., & Zhao, W. (2014). Role of Candida Albicans-secreted aspartyl proteinases (Saps) in severe early childhood caries. International Journal of Molecular Sciences, 15(6), 10766–10779.
Monroy-Pérez, E., Paniagua-Contreras, G., Vaca-Paniagua, F., Negrete-Abascal, E., & Vaca, S. (2013). SAP Expression in Candida albicans Strains Isolated from Mexican Patients with Vaginal Candidosis. International Journal of Clinical Medicine, 04(01), 25–31.
Mulatsari, E., Mumpuni, E., & Ramadhan, I. (2019). Skrining Virtual dan Elusidasi Moda Ikatan Senyawa dalam Bawang Putih (Allium Sativum L.) sebagai Penghambat Reseptor Advanced Glycation end Products. Jurnal Ilmu Kefarmasian Indonesia, 17(2), 210–217.
Nugraheni, D., Agusni, I., & Ervianti, E. (2015). Profil Enzim Secreted Aspartyl Proteinase (SAP) pada Isolat Pasien Kandidiasis Vulvovaginalis (KVV) di RSUD Dr. Soeto-mo Surabaya. Berkala Ilmu Kesehatan Kulit dan Kelamin, 27(1), 17–23.
Pires, D. E., Blundell, T. L., & Ascher, D. B. (2015). pkCSM: Predicting small-molecule pharmacokinetic and toxicity properties using graph-based signatures. Journal of Medicinal Chemistry, 58(9), 4066–4072.
Santos, A. L., Braga-Silva, L. A., Gonçalves, D. S., Ramos, L. S., Oliveira, S. S., Souza, L. O., Oliveira, V. S., Lins, R. D., Pinto, M. R., Muñoz, J. E., Taborda, C. P., & Bran-quinha, M. H. (2021). Repositioning lopinavir, an hiv protease inhibitor, as a promising antifungal drug: Lessons learned from candida albicans—in silico, in vitro and in vivo approaches, vol. 7.
Sari, I. W., Junaidin, J., & Pratiwi, D. (2020). STUDI MOLECULAR DOCKING SENYAWA FLAVONOID HERBA KUMIS KUCING (Orthosiphon stamineus B.) PADA RESEPTOR α-GLUKOSIDASE SEBAGAI ANTIDIABETES TIPE 2. Jurnal Farmagazine, 7(2), 54.
Shakya, A. K. (2019). Natural phytochemicals: Potential anti-HCV targets in silico approach. Journal of Applied Pharmaceutical Science, 9(8), 94–100.
Shukla, M., & Rohatgi, S. (2020). Vaccination with secreted aspartyl proteinase 2 protein from candida parapsilosis can enhance survival of mice during c. tropicalis-mediated systemic candidiasis. Infection and Immunity, 88(10), 1– 21.
Srivastava, S., Shree, P., Pandey, H., & Tripathi, Y. B. (2018). Incretin hormones receptor signaling plays the key role in antidiabetic potential of PTY-2 against STZ-induced pancreatitis. Biomedicine and Pharmacotherapy, 97(October 2017), 330–338.
Wang, W., Gan, N., Sun, Q., Wu, D., Gan, R., Zhang, M., Tang, P., & Li, H. (2019). Study on the interaction of er-tugliflozin with human serum albumin in vitro by multis-pectroscopic methods, molecular docking, and molecular dynamics simulation. Spectrochimica Acta - Part A: Molecular and Biomolecular Spectroscopy, 219, 83–90.
Willems, H. M., Bruner, W. S., Barker, K. S., Liu, J., Palmer, G. E., & Peters, B. M. (2017). Overexpression of Candida albicans secreted aspartyl proteinase 2 or 5 is not sufficient for exacerbation of immunopathology in a murine model of vaginitis. Infection and Immunity, 85(10), 1–12.
GHOLAM DKK.
62
Discussion and feedback