DESAIN PRIMER SECARA IN SILICO UNTUK AMPLIFIKASI FRAGMEN GEN rpoB Mycobacterium tuberculosis DENGAN POLYMERASE CHAIN REACTION (PCR)
on
Desain Primer secara in silico untuk Amplifikasi Fragmen Gen rpoB Mycobacterium tuberculosis
dengan Polymerase Chain Reaction (PCR) (Pradnyaniti, D.G, Wirajana, I.N, Yowani, S.C)
DESAIN PRIMER SECARA IN SILICO UNTUK AMPLIFIKASI FRAGMEN GEN rpoB Mycobacterium tuberculosis DENGAN POLYMERASE CHAIN REACTION (PCR)
Pradnyaniti, D.G1), Wirajana, I.N2), Yowani, S.C1)
1)Jurusan Farmasi - Fakultas Matematika dan Ilmu Pengetahuan Alam - Universitas Udayana 2)Jurusan Kimia - Fakultas Matematika dan Ilmu Pengetahuan Alam - Universitas Udayana
Korespondensi: Desak Gede Pradnyaniti
Jurusan Farmasi - Fakultas Matematika dan Ilmu Pengetahuan Alam - Universitas Udayana
Jalan Kampus Unud-Jimbaran, Jimbaran-Bali, Indonesia 80364 Telp/Fax: 0361-703837
Email: ecak_cansa@yahoo.com
ABSTRAK
Amplifikasi DNA Mycobacterium tuberculosis dari gen rpoB dilakukan dengan metode polymerase chain reaction (PCR). Amplifikasi DNA dengan PCR diperlukan sepasang primer (forward dan reverse) untuk membatasi daerah yang ingin diamplifikasi. Penelitian ini bertujuan untuk mendesain sepasang primer agar dapat mengamplifikasi fragmen 0,5 kb gen rpoB M.tuberculosis. Desain primer dilakukan secara in silico dengan bantuan program clone manager suite 6 (University of Groningen). Template yang digunakan dalam mendesain primer adalah sekuen gen rpoB M. tuberculosis H37RV wild type, yang diperoleh dari database NCBI dengan kode genbank U12205.1.
Penelitian ini telah berhasil memperoleh sekuen sepasang primer (forward dan reverse) dengan panjang masing-masing adalah 22 oligonukleotida. Primer ini dapat mengamplifikasi secara in silico fragmen 0,5 kb gen rpoB M. tuberculosis pada rentang daerah 990-1496 pb dengan panjang fragmen sebesar 507 pb.
Kata kunci: desain primer, PCR, gen rpoB Mycobacterium tuberculosis
-
1. PENDAHULUAN
Desain primer dilakukan untuk memperoleh primer yang dapat digunakan dalam amplifikasi DNA dengan metode polymerase chain reaction (PCR). Keberhasilan amplifikasi DNA tergantung dari ketepatan primer yang digunakan (Diss, 2003). Primer yang digunakan dalam proses PCR harus dapat membatasi daerah yang akan diamplifikasi. Primer berfungsi sebagai pembatas fragmen DNA target yang akan diamplifikasi (Handoyo dan Rudiretna, 2001).
Amplifikasi DNA dengan metode PCR terdiri dari tiga tahap. Tahap awal dari proses amplifikasi yaitu denaturasi untai DNA, selanjutnya dilakukan penempelan primer pada fragmen DNA target (annealing) dan tahap akhir merupakan proses extension yaitu proses
pemanjangan sekuen DNA (Sulistyaningsih, 2007).
Perancangan untuk memperoleh suatu primer yang memenuhi kriteria primer yang baik untuk amplifikasi dilakukan secara in silico, yaitu merancang/mendesain primer dengan bantuan suatu program dalam komputer. Penelitian ini dilakukan desain primer dengan sekuen gen rpoB M. tuberculosis. Gen rpoB (DNA-dependent RNA polymerase sub unit β) merupakan salah satu gen pada Mycobacterium tuberculosis yang berperan dalam sebagian besar resistensi terhadap Rifampisin (Syaifudin dkk., 2007). Rifampisin merupakan obat lini pertama yang umum digunakan dalam pengobatan tuberkulosis (TB). Lima negara yang memiliki insiden TB yang tinggi pada tahun 2010 adalah
India, Cina, Afrika selatan, Indonesia dan Pakistan (WHO, 2011).
Tujuan penelitian ini yaitu memperoleh primer yang dapat digunakan untuk membatasi daerah amplifikasi terhadap gen rpoB M. tuberculosis H37RV yang didesain secara in silico. Sehingga dengan primer yang didesain tersebut, dapat digunakan dalam proses amplifikasi menggunakan PCR.
-
2. BAHAN DAN METODE
-
2.1 Bahan
-
Bahan yang digunakan dalam penelitian ini adalah sebuah softwere yaitu clone manager suite 6 (University of Groningen) untuk mendesain primer secara in silico. Data sekuen gen rpoB M. tuberculosis yang digunakan diperoleh dari database NCBI pada URL http://www.ncbi.nlm.nih.gov dengan kode genbank U12205.1.
-
2.2 Metode
Desain primer dilakukan pada program clone manager suite 6. Pada “Molecule List” dimasukkan sekuen gen rpoB M. tuberculosis. Selanjutnya dimasukkan panjang primer dan daerah target yang diinginkan pada menu “Design Primer” sehingga program memberikan data beberapa pilihan primer. Hasil analisis dilihat menggunakan “Primer Report”.
Rentang daerah primer dan hasil analisis juga dapat dilihat dengan menggunakan “Analyze Mix Wizard”, dengan dimasukkan sekuen masing-masing primer (forward dan reverse) dan sekuen gen rpoB M. tuberculosis. Sedangkan untuk menganalisis masing-masing primer dilakukan dengan dimasukkan nama primer dan sekuen primer yang dianalisis menggunakan “Direct Entry”.
-
3. HASIL
Program clone manager suite 6 memberikan 14 pilihan pasang primer setelah dimasukkan panjang primer yaitu 22 oligonukleotida dengan daerah target 1020-1480 (tabel A.1). Sekuen primer dengan kriteria terbaik yang dapat digunakan dalam proses PCR yaitu primer FrTb 5'-GTCGACGCTGACCGAAGAAGAC-3' dan RrTb 5'-GAGCCGATCAGACCGATGTTGG-3'
(tabel A.2). Sekuen primer telah memenuhi kriteria parameter untuk sebuah primer yang digunakan dalam proses PCR (tabel A.3).
-
4. PEMBAHASAN
Secara umum, primer yang ideal memiliki panjang antara 18 sampai 30 oligonukleotida. Panjang ini diharapkan cukup untuk dapat mengikat template pada suhu annealing dan mendapatkan sekuen yang spesifik (Borah, 2011). Jika primer terlalu pendek maka dapat mengurangi spesifisitas primer sehingga mudah menempel pada template dengan suhu annealing yang tidak diinginkan. Sedangkan jika primer terlalu panjang tidak mempengaruhi spesifisitas secara bermakna (Handoyo dan Rudiretna, 2001).
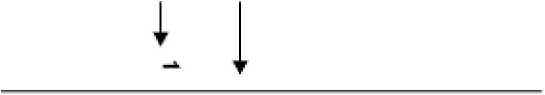
Analisis menggunakan analyze mix wizard, yaitu simulasi primer dan DNA template M. tuberculosis dalam proses PCR menghasilkan fragmen sebesar 507 pb yang berada pada posisi 990-1496 (gambar B.1). Hasil analisis menujukkan bahwa primer berada dalam kondisi terbaik yaitu tidak terdapat dimer pada ujung 3′ maupun di tempat yang lain, primer ini tidak berikatan dengan internal primer ataupun primer pasangannya. Primer yang didesain ini juga tidak menempel pada banyak molekul, dan dapat menempel dengan baik pada daerah yang ditargetkan. Sekuen primer sebaiknya tidak memiliki daerah yang dapat berikatan dengan internal primer tersebut maupun dengan primer pasangannya. Primer sebaiknya menggunakan pasangan basa yang komplemen dan tidak memiliki banyak binding sites dalam genom target (Diss, 2003).
Primer yang baik merupakan primer yang memenuhi kriteria parameter primer. Parameter tersebut antara lain: melting temperature (Tm), persentase jumlah G dan C (%GC), 3′dimer, stabilitas, repeats, dan hairpins.
Melting temperature (Tm) untuk primer forward dan reverse secara umum serupa (dalam rentang 2 sampai 4 oC) dan di atas 60oC untuk menghasilkan produk PCR yang baik (Sulistyaningsih, 2007). Primer dengan Tm yang terlalu tinggi dapat menghasilkan produk PCR yang rendah. Sedangkan Tm yang terlalu rendah memiliki kecenderungan
menempel ditempat lain dan menghasilkan produk yang tidak spesifik. Tm pada primer yang didesain telah memenuhi kriteria yaitu 67oC. Tm dapat dihitung secara manual dengan rumus Tm= 2(A+T) + 4(G+C). Tm dari primer tersebut dapat digunakan untuk menetapkan suhu annealing dalam PCR (Borah, 2011; Handoyo dan Rudiretna, 2001).
Persentase GC merupakan persentase banyaknya guanin dan sitosin dalam suatu primer, sebaiknya % GC berada pada rentang 40-60 % (Borah, 2011). Primer yang didesain memiliki % GC sebesar 59% yang masih berada pada rentang kriteria % CG. Primer dengan % GC yang rendah dapat menurunkan efisiensi proses PCR yang disebabkan karena primer tidak mampu berkompetisi untuk menempel secara efektif pada template (Handoyo dan Rudiretna, 2001).
Dimer pada ujung 3′ primer sebaiknya tidak lebih dari 3 basa karena dapat menurunkan spesifisitas primer (Handoyo dan Rudiretna, 2001). Primer sebaiknya tidak mempunyai 3 atau lebih basa G atau C pada 3′dimer, karena dapat menstabilkan annealing primer non spesifik (Sulistyaningsih, 2007). Dimer pada ujung 3′ masing-masing primer yang dirancang adalah 1 dimer pada primer forward dan 2 dimer pada primer reverse, dimer tersebut tidak melebihi batas dari parameter dimer suatu primer.
Stabilitas suatu primer mempengaruhi penempelan primer pada template. Rentang stabilitas suatu primer adalah 1,2 – 2 kcal. Jika terlalu stabil maka primer akan menempel kuat pada template dan jika tidak stabil primer tidak dapat menempel dengan baik pada template. Pada primer yang telah didesain memiliki stabilitas 1,9 kcal pada primer forward dan 1,6 kcal pada primer reverse, stabilitas tersebut masih berada pada rentang stabilitas primer yang baik.
Repeats merupakan nukleotida yang berulang dalam primer, adanya repeats dapat menyebabkan terjadinya pemempelan primer di tempat yang tidak diinginkan (mispriming). Pada primer yang didesain ini tidak terdapat repeats,
sehingga primer yang dihasilkan berada dalam kondisi yang baik (Borah, 2011).
Haipins merupakan interaksi intramolekuler dalam primer. Haipins dalam primer dapat menganggu proses penempelan primer pada template dalam proses PCR (Borah, 2011). Dalam primer yang didesain tidak terdapat haipins, sehingga primer ini cukup baik dapat digunakan dalam proses PCR.
Primer yang didesain ini dapat memotong daerah target (dapat membatasi daerah amplifikasi dalam proses PCR) dengan tepat sesuai rentang daerah yang dirancang secara in silico. Hal ini menunjukan bahwa primer yang didesain sudah cukup baik untuk dapat digunakan dalam proses PCR dan dapat menghasilkan produk sesuai dengan rentang daerah yang diinginkan.
-
5. KESIMPULAN
Primer dengan panjang sekuen sejumlah 22 oligonukleotida telah berhasil didesain dalam kondisi terbaik dengan fragmen sebesar 507 pb.
UCAPAN TERIMA KASIH
Penulis mengucapkan terimakasih kepada semua pihak yang telah membantu, sehingga penelitian ini dapat terlaksana dengan baik, khususnya kepada Prof. Dr. Bauke W. (Rijk Universiteit Groningen; University of Groningen) atas bantuan softwere yang telah diberikan.
DAFTAR PUSTAKA
Borah, P. 2011. Primer Designing for PCR.
Science Vision 11(3): P. 134 -136.
Diss, T. 2003. The Polymerase Chain Reaction. In Crocker, J. dan Paul, G.M. editors. Molecular Biology in Cellular Pathology. United Kingdom: John Willey and Sons, Ltd. P. 193-210.
Handoyo, D. dan Rudiretna, A. 2000. Prinsip Umum dan Pelaksanaan Polymerase Chain Reaction (PCR) [General Principles and Implementation of Polymerase Chain Reaction]. Unitas, 9(1): P. 17-29.
Sulistyaningsih, E. 2007. Polymerase Chain Reaction (PCR): Era Baru Diagnosis
|
dan Manajemen Penyakit Infeksi. |
Available |
at |
|
Biomedis. 1(1): P. 17-25. | ||
|
WHO, 2011. Global tuberculosis control: WHO |
11/9789241564380_eng.pdf. |
Accesed |
|
report 2011. Geneva, Switzerland: |
on September 18th, 2012. | |
World Health Organization. P. 1-27.
APENDIK A.
Tabel A.1 Hasil pilihan produk primer yang didesain
|
No |
GC (%) |
Tm (oC) |
Dimers Maksimal |
False Priming (oC) | ||||
|
A |
B |
A |
B |
3′ |
Lain |
A |
B | |
|
1 |
59 |
59 |
67 |
67 |
2 |
6 |
- |
- |
|
2 |
59 |
59 |
67 |
67 |
2 |
6 |
- |
5 |
|
3 |
59 |
59 |
68 |
67 |
2 |
6 |
8 |
- |
|
4 |
59 |
59 |
68 |
67 |
2 |
6 |
8 |
5 |
|
5 |
59 |
59 |
67 |
70 |
2 |
6 |
- |
17 |
|
6 |
59 |
59 |
67 |
69 |
2 |
6 |
- |
20 |
|
7 |
59 |
59 |
68 |
70 |
2 |
6 |
8 |
17 |
|
8 |
59 |
59 |
68 |
69 |
2 |
6 |
8 |
20 |
|
9 |
54 |
59 |
66 |
70 |
2 |
6 |
14 |
17 |
|
10 |
59 |
59 |
68 |
69 |
1 |
6 |
8 |
33 |
|
11 |
54 |
59 |
66 |
69 |
2 |
6 |
14 |
20 |
|
12 |
54 |
59 |
64 |
70 |
2 |
6 |
22 |
17 |
|
13 |
54 |
59 |
66 |
69 |
1 |
6 |
14 |
33 |
|
14 |
54 |
59 |
64 |
69 |
2 |
6 |
22 |
33 |
Table A.2 Sekuen primer yang dipilih
|
Nama Primer |
Panjang |
Urutan Sekuen Nukleotida |
|
FrTb |
22 |
5'-GTCGACGCTGACCGAAGAAGAC-3' |
|
RrTb |
22 |
5'-GAGCCGATCAGACCGATGTTGG-3' |
Tabel A.3 Kriteria primer yang didesain
|
Parameter Primer |
Batas Rentang Kriteria Primer |
Primer | |
|
A |
B | ||
|
Panjang |
18-30 basa |
22 |
22 |
|
GC (%) |
50-60 % |
59 |
59 |
|
Tm (OC) |
55-80 OC |
67 |
67 |
|
3′ Dimers |
< 3 pada ujung 3′ |
1 |
2 |
|
Dimers- Any |
< 7 basa |
6 |
4 |
|
Stabilitas (Kcals) |
>= 1,2 kcals |
1,9 |
1,6 |
|
Runs of bases |
< 3 bases Runs |
2 |
2 |
|
Repeats |
< 3 dinuc repeats |
- |
- |
|
Hairpins |
Annealing 55 OC |
- |
- |
|
False Priming (OC) |
- |
- | |
APENDIK B.
993 1496
S07pb
Gambar B.1 Posisi penempelan primer yang didesain pada DNA template.
Keterangan gambar: primer forward berada pada nukleotida ke 990 dan primer reverse berada pada nukleotida ke 1496, menghasilkan fragmen sebesar 507 pb
JURNAL FARMASI UDAYANA
jurusan Farmasi-Fakultas mipa-universitas udayana
bukit Jimbaran - bau
• (0361) 703837 * Email: j »rnalfarmasiudavana@gmail.com
SURAT PERNYATAAN
PERSETUJUAN PEMBIMBING
Yang bertandatangan di bawah ini, menyatakan bahwa :
|
Artikel dengan judul |
:9esa^ ‰'∙Mir stra^ <1 <lrCo unWS C∖Mγ∖i(⅛l^⅞ι H-OiqMfi-X t>Y" Tu^ lY^eofioicW-iu-v. Fvibn-v-u U⅞ is A<ι⅛xqciΛ MyftICroist chou’-s P-Ooichuix CPCi2-") |
|
Disusun oleh |
: Dewf- We VWm√y""*^' |
|
NIM |
: o<⅛oBs^o⅛τ>^fe |
|
Email mahasiswa |
: ecn^-cause’β yc'hι>u cbm |
Telah kami setujui untuk dipublikasi pada “Jurnal Farmasi Udayana”.
Demikian surat pernyataan ini kami buat, agar dapat dipergunakan sebagaimana mestinya.
BukitJimbaran, .⅛3. ..Se.pt^^ 20.15

⅛M.⅛', M
Dr.⅛⅜,⅛Λ⅜ Uh NIP. Iqyic2
Pembimbi gas Akhir
130
Discussion and feedback