DESAIN PRIMER UNTUK AMPLIFIKASI REGIO PROMOTER GEN inhA ISOLAT P016 MULTIDRUG RESISTANCE Mycobacterium tuberculosis DENGAN METODE POLYMERASE CHAIN REACTION
on
Sari dkk.
pISSN: 2301-7716; eISSN: 2622-4607 Jurnal Farmasi Udayana, Vol 7, No.1, Tahun 2018, 34-39
Desain Primer untuk Amplifikasi Regio Promoter Gen inhA Isolat P016 Multidrug Resistance Mycobacterium tuberculosis dengan Metode
Polymerase Chain Reaction
Sari, E.N.1*, R. W. Dewi1, V. R. Dewi1, S. C. Yowani1, P. S. Yustiantara1
1Program Studi Farmasi, Fakultas Matematika dan Ilmu Pengetahuan Alam, Universitas Udayana, Denpasar-Bali, 80361
*Penulis korespondensi:: ekanatasari376@yahoo.co.id
Received July 2, 2018; Accepted July 21, 2018
ABSTRAK
Mutasi pada promoter gen inhA bertanggungjawab terhadap resistensi isoniazid dan resistensi silang terhadap ethionamid. Untuk melakukan identifikasi mutasi dengan polymerase chain reaction (PCR), diperlukan sepasang primer yang mampu mengamplifikasi regio target. Penelitian ini bertujuan menghasilkan sepasang primer terbaik untuk mengamplifikasi regio promoter gen inhA yang didesain dengan menggunakan program Clone Manager Suite 6. Primer didesain menggunakan sekuen promoter gen inhA (Genbank: U66801) yang diperoleh dari situs www.ncbi.nlm.nih.gov. Hasil desain diperoleh sepasang primer terbaik yang diuji secara in vitro menggunakan metode Polymerase Chain Reaction (PCR). Proses amplifikasi PCR dilakukan sebanyak 40 siklus dengan kondisi berikut: predenaturase (95oC selama 15 menit), denaturasi (94oC selama 1 menit), annealing (56oC selama 1 menit 30 detik), elongasi (72oC selama 2 menit), dan elongasi akhir (72oC selama 10 menit). Deteksi produk PCR dilakukan dengan elektroforesis gel agarosa 1,3% b/v dan divisualisasi dengan alat UV transluminator. Hasil penelitian diperoleh primer forward yaitu 5’-GGTCGAAGTGTGCTGAGTC-3‘ dan primer reverse 5‘-TGCTCTTCTACCGCCGTGA-3‘ yang memenuhi kriteria primer yang baik berdasarkan acuan Clone Manager Suite 6. Sepasang primer tersebut telah mampu mengamplifikasi regio promoter gen inhA dengan panjang produk yang dihasilkan sebesar 373 bp.
Kata kunci: Clone Manager Suite 6, promoter gen inhA, primer, Polymerase Chain Reaction
ABSTRACT
Mutations in the inhA promoter region are responsible for isoniazid resistance and crossresistance to ethionamide. To identify mutations with polymerase chain reaction (PCR), a pair of primers that can amplify the target region. This aim of this study was to obtain the best primer pair to amplify the inhA promoter region using the Clone Manager Suite 6 program. The inhA promoter sequence (Genbank: U66801) obtained from the www.ncbi.nlm.nih.gov was used as a template. The design results obtained the best primer pair tested in vitro using Polymerase Chain Reaction (PCR) method. The PCR amplification process was performed for 40 cycles with the following conditions: predenaturase (95oC for 15 minutes), denaturation (94 oC for 1 minute), annealing (56 oC for 1 minute 30 seconds), elongation (72 oC for 2 minutes), and final elongation (72 oC for 10 minutes). Detection of PCR products was performed in agarose gel electrophoresis 1.3% w / v and visualized by UV transluminator tool. The results obtained were forward primers of 5'-GGTCGAAGTGTGCTGAGTC-3 'and reverse primer 5'-
TGCTCTTCTACCGCCGTGA-3' which met the good primary criterion based on Clone Manager Suite 6. The pair of primers has been able to amplify the inhA promoter region by the length of product produced at 373 bp.
Keywords: Clone Manager Suite 6, inhA promoter, primer, Polymerase Chain Reaction
Multidrug resistance tuberculosis (MDR-TB) merupakan tuberkulosis yang disebabkan oleh bakteri yang resisten terhadap setidaknya dua obat anti tuberkulosis lini pertama yaitu rifampisin (RIF) dan isoniazid (INH). Kasus MDR-TB di Indonesia berdasarkan data Kementrian Kesehatan Republik Indonesia hingga triwulan kedua 2015 menyatakan sebanyak 15.380 kasus yang terduga MDR-TB, 1.860 kasus terkonfirmasi, dan 1.566 kasus yang terobati.
MDR-TB secara molekuler terkait erat dengan adanya mutasi pada gen-gen yang spesifik. Bolela et al., (2016) menunjukkan mutasi pada promoter gen inhA bertanggungjawab terhadap terjadinya resistensi isoniazid (INH) dan dapat menyebabkan terjadinya resistensi silang terhadap ethionamid (ETH). Penelitian lainnya yang dilakukan oleh Bang et. al. (2011) menunjukkan pada keseluruhan isolat MDR-TB yang diteliti 45 isolat (23,6%) mengalami mutasi pada promoter gen inhA posisi -15 dengan perubahan basa sitosin menjadi timin (C÷T).
Mutasi pada promoter gen inhA dapat menyebabkan resistensi silang pada Ethionamid (ETH) dengan tingkat yang tinggi. ETH merupakan obat tuberkulosis lini kedua yang sering digunakan pada rejimen terapi MDR-TB, sehingga resistensi ETH yang tidak teridentifikasi dapat menyebabkan rejimen terapi yang diberikan pada pasien MDR-TB menjadi tidak efektif dan dapat mengarah pada terjadinya Extensively Drug Resistant Tuberculosis (XDR-TB).
Identifikasi mutasi dapat dilakukan dengan metode polymerase chain reaction (PCR). Proses amplifikasi dengan PCR selain kondisi yang sesuai, diperlukan pula primer. Primer berfungsi sebagai pembatas fragmen DNA target yang akan diamplifikasi. Setiap fragmen target memerlukan primer yang sesuai sehingga primer harus memenuhi kriteria primer yang baik sehingga dapat mengamplifikasi fragmen gen yang spesifik. Kriteria primer meliputi panjang primer, %GC, Tm (melting temperature), dimer, hairpin, run, repeat, stabilitas, dan false priming. Untuk
mengetahui apakah primer mampu mengamplifikasi fragmen gen target, produk PCR dideteksi dengan metode elektroforesis gel agarosa 1,3% kemudian divisualisasi dengan UV transluminator.
Penelitian ini akan dilakukan desain primer untuk amplifikasi promoter gen inhA dengan Clone Manager Suite 6 isolat P016 MDR-TB. Hasil desain primer kemudian akan diuji secara in vitro dengan metode PCR. Primer yang akan dihasilkan pada penelitian ini belum pernah digunakan pada penelitian lainnya. Primer yang dihasilkan diharapkan nantinya dapat digunakan untuk amplifikasi regio promoter gen inhA pada proses PCR untuk identifikasi mutasi pada regio promoter gen inhA.
Bahan dan Alat
Bahan yang digunakan pada penelitian ini adalah sepasang primer hasil desain untuk amplifikasi regio promoter gen inhA meliputi primer forward 5’-GGTCGAAGTGTGCTGAGTC-3’ dan primer reverse 5’-TGCTCTTCTACCGCCGTGA-3’, High Pure PCR Template Preparation Kit (Roche Germany), isolat P016 MDR-TB yang diperoleh dari Laboratorium Biomedik Terpadu Fakultas Kedokteran Universitas Udayana, TBE (Tris-Borat-EDTA), PCR Mix (Go Taq Green Master Mix), nuclease free water Promega, gel agarosa 1,3% (Top Vision Agarose Thermo Scientific), Marker DNA ladder 100 bp (Thermo Scientific Generuler), DNA loading dye (Blue juiceTM Invitrogen), dan pewarna gel red (Promega)
Alat yang digunakan pada penelitian ini adalah aplikasi Clone Manager Suite 6 (University of Groningen), mesin PCR (Veriti Thermal Cycler), alat elektroforesis (Mupid-Exu Advance), UV-transiluminator Gel Doc (BioRad), microwave, filter tube, Vortex, shaker rotator (tipe H-SR 2000), Sorvall Biofuge Primo R Centrifuge), dan freezer.
Metode
Sekuen gen inhA M. tuberculosis (Genbank: U66801) diperoleh dari www.ncbi.nlm.nih.gov digunakan sebagai template dalam desain primer pada penelitian
ini. Software yang digunakan untuk mendesain primer secara in silico yaitu Clone Manager Suite 6 (University of Groningen). Penelitian ini dilakukan dengan beberapa tahapan yaitu desain primer, Isolasi isolat P016 MDR-TB, Amplifikasi dengan PCR, dan Deteksi dengan elektroforesis gel agarosa 1,3%.
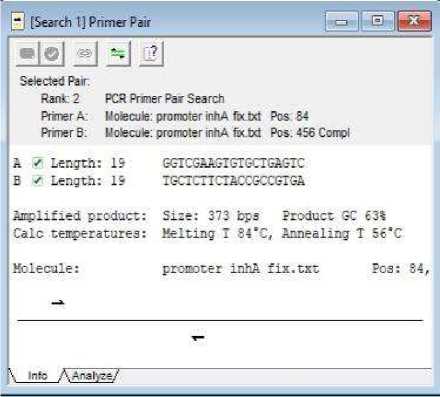
Primer yang digunakan dalam mengamplifikasi fragmen promoter gen inhA dirancang menggunakan program Clone Manager Suite 6 (University of Groningen). Langkah awal yang dilakukan adalah dengan memasukkan data urutan masing-masing nukleotida promoter gen inhA (GenBank : U66801) yang ada pada database URL://www.ncbi.nlm.nih.gov ke dalam program Clone Manager Suite 6. Sepasang primer dirancang dengan cara memilih menu bar primer design. Panjang basa primer yang ingin dibuat dimasukkan dan ditentukan daerah target yang akan diamplifikasi berdasarkan literatur data mutasi. Primerprimer yang dihasilkan dalam barisan rank dilakukan analisis sesuai dengan kriteria primer yang diinginkan. Tahap terakhir adalah memilih sub menubar primer list, dan memilih analyze untuk melakukan analisis produk yang akan terbentuk dari campuran primer dengan template DNA. Primer dipilih sesuai dengan kriteria yang ditetapkan oleh program Clone Manager Suite 6. Hasil desain primer promoter gen gyrA dapat dilihat pada Tabel 1.
Uji primer secara in vitro terdiri dari beberapa tahapan:
Isolasi DNA Isolat P016 MDR-TB
Proses isolasi DNA dilakukan dengan menggunakan High Pure PCR template Preparation Kit (Roche Germany). Sampel bakteri diperoleh dari Laboratorium Biomedik Terpadu Fakultas Kedokteran Universitas Udayana. Tahapan-tahapan isolasi dilakukan sesuai dengan protocol yang terdapat pada kit. Pada akhir tahap isolasi dihasilkan larutan yang mengandung DNA dengan konsentrasi 96 ng/µl yang siap digunakan untuk tahap amplifikasi dengan PCR.
Amplifikasi Regio Promoter gen inhA dengan Teknik PCR
Template yang digunakan pada tahap amplifikasi merupakan hasil isolasi DNA P016. Amplifikasi dilakukan sebanyak 40 siklus yang terdiri dari predenaturasi (95oC selama 15 menit), denaturasi (94oC selama 1 menit), annealing (56oC selama 1 menit 30 detik), elongasi (72oC selama 2 menit), dan elongasi akhir (72oC selama 10 menit).
Deteksi Produk PCR
Produk PCR dideteksi dengan menggunakan elektroforesis gel agarosa dengan konsentrasi 1,3% b/v. Pewarnaan gel menggunakan Gel Red.
Gambar 1. Primer Info pada Clone Manager Suite 6
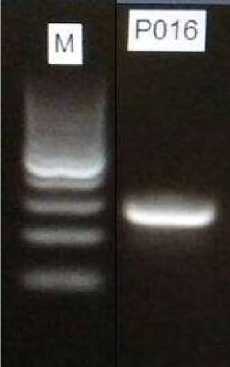
Gambar 2. Elektroforegram amplifikasi fragmen promoter gen inhA isolat P016 pada suhu annealing
56oC, Marker DNA ladder 100 bp (0,1 kb)
|
Tabel 1. Hasil Desain Primer Dengan Clone Manager Suite 6 | |||
|
Kriteria |
Promoter-inhA-F |
Promoter inhA-R |
Acuan Clone Manager Suite 6 |
|
Panjang primer |
19 |
19 |
18-22 |
|
Persen (%) GC |
57 |
57 |
50-60 |
|
Tm (oC) |
62 |
65 |
55-80 |
|
Dimer pada ujung 3’ |
2 |
2 |
<3 |
|
Dimer selain ujung 3’ |
4 |
2 |
<7 |
|
Stabilitas (kcals) |
1,3 |
2,3 |
≥1,2 |
|
Runs (nukleotida) |
2 |
2 |
<3 |
|
Repeat (dinukleotida) |
2 |
2 |
<3 |
|
Hairpin |
Tidak ada |
Tidak ada |
Tidak ada |
|
False Priming |
Tidak ada |
Tidak ada |
Tidak ada |
Primer yang digunakan untuk mengamplifikasi promoter gen inhA didesain menggunakan program Clone Manager Suite 6 dengan data sekuens DNA gen inhA M. tuberculosis (GenBank : U66801.1). Data sekuens diperoleh dari situs URL: www.ncbi.nml.nih.gov. Primer hasil desain akan digunakan untuk mengamplifikasi daerah gen yang mencakup titik mutasi yang sering ditemukan pada promoter gen inhA, yaitu nukleotida ke-209 (posisi -15). Sepasang primer yang dipilih dari hasil desain dapat mengamplifikasi basa nukleotida dari urutan 84 hingga 456 dengan ukuran produk PCR 373 bp. Sepasang primer tersebut meliputi primer forward dengan urutan basa nukleotida 5’-GGTCGAAGTGTGCTGAGTC-3’ dan primer reverse dengan urutan 5’-TGCTCTTCTACCGCCGTGA-3’. Hasil desain dan kriteria primer dapat dilihat pada Tabel 1.
Gambar 1. menunjukkan hasil desain kedua primer promoter gen inhA dengan Clone Manager Suite 6 telah memenuhi kriteria primer yang baik berdasarkan hasil analisis panjang primer, %GC, Tm, 3’dimer, dimer any, stabilitas, run, repeat, hairpin, dan false priming. Panjang primer promoter-inhA-F adalah 19 basa dan primer promoter-inhA-F sebesar 19 basa. Panjang primer yang ideal berdasarkan literatur adalah 18-30 bp (Sasmito dkk., 2014). Primer dengan panjang kurang dari 18 basa memiliki kemungkinan terjadinya mispriming (penempelan primer pada tempat yang tidak diinginkan) tinggi. Hal ini akan menyebabkan
berkurangnya spesifisitas primer dan berakibat pada efektivitas dan efisiensi proses PCR. Penggunaan primer yang terlalu panjang tidak akan meningkatkan spesifisitas primer secara bermakna dan menyebabkan biaya primer yang lebih mahal (Handoyo dan Rudiretna, 2000).
Persentase GC atau kandungan jumlah basa G dan C dapat mempengaruhi Tm yang dimiliki suatu primer. Persen (%) GC primer promoter-inhA-F dan promoter-inhA-R hasil desain masing-masing sebesar 57%. Kandungan basa G dan C pada primer umumnya adalah 35-60% (Henegariu et al., 1997). Hal ini disebabkan karena primer dengan persen GC rendah diperkirakan tidak akan mampu berkompetisi untuk menempel secara efektif pada target yang dituju sehingga akan menurunkan efisiensi proses PCR, sedangkan persen GC yang tinggi akan meningkatkan Tm serta suhu annealing PCR (Handoyo dan Rudiretna, 2000).
Primer promoter-inhA-F dan promoter-inhA-R memiliki Tm (melting temperature) masing-masing 62°C dan 65°C. Kriteria Tm yang ideal adalah 55°-65°C. Primer dengan Tm di atas 65°-70°C akan mudah mengalami mispriming pada temperatur rendah. Primer dengan Tm rendah tidak akan dapat bekerja pada temperatur tinggi. Temperatur ini akan berpengaruh pada suhu annealing dalam proses PCR (Handoyo dan Rudiretna, 2000). Tm merupakan suhu dimana 50% untaian ganda DNA telah terpisah. Sepasang primer yang mengamplifikasi region promoter gen inhA ini memiliki perbedaan Tm 3°C. Perbedaan Tm antar primer yang diperbolehkan tidak lebih
dari 5°C, apabila perbedaan Tm terlalu jauh maka akan menyebabkan penurunan proses amplifikasi, atau bahkan memungkinkan tidak terjadinya proses amplifikasi (Sasmito dkk., 2014).
Pada primer forward dan reverse sebaiknya tidak ada dimer maupun hairpin (Henegariu, et al., 1997). Dimer menunjukkan adanya hibridisasi antara basa primer yang identik akibat adanya urutan yang komplementer pada ujung 3’. Hal ini mempengaruhi efisiensi proses penempelan pada target (Handoyo dan Rudiretna, 2000). Adanya dimer pada ujung 3’ dapat mengganggu proses amplifikasi karena amplifikasi dimulai dari ujung 3’ sehingga dapat mengurangi bahkan tidak membentuk produk PCR. Dimer-any adalah terjadinya dimer pada daerah selain 3’. Dimer-any akan menyebabkan berkurangnya konsentrasi primer yang dibutuhkan untuk proses amplifikasi (Borah, 2011). Pasangan primer promoter gen inhA yang dihasilkan memiliki dimer pada ujung 3’ sebanyak 2 bp pada kedua primer. Dimer pada daerah selain 3’ terjadi 4 bp pada primer forward dan 2 bp pada primer reverse. Kriteria dimer dan dimer any untuk primer promoter gen inhA sudah memenuhi kriteria pada program Clone Manager suite 6 yang memiliki batas toleransi dimer pada ujung 3’ adalah <3 bp dan batas dimer di daerah selain 3’ adalah <7 bp. Hairpin memiliki kesamaan dengan dimer, namun pada hairpin ujung-ujung primer saling berkomplemen (Judelson, 2006). Interaksi hairpin pada primer sama sekali tidak diperbolehkan karena primer yang saling berkomplemen akan membentuk lipatan untai nukleotida sehingga proses amplifikasi tidak dapat berlangsung (Borah, 2011).
Stabilitas kedua pasang primer yang dihasilkan, telah memenuhi syarat dari kriteria pada program Clone Manager Suite 6. Stabilitas yang disarankan adalah lebih dari atau sama dengan 1,2 kcals. Angka ini menunjukkan stabilitas 5 basa (pentamer) dari ujung 5’ lebih besar dibandingkan dengan pada ujung 3’. Proses amplifikasi dimulai dari ujung 3’ dan berakhir pada ujung 5’ sehingga untuk memperoleh produk PCR yang stabil dan sesuai dengan ukuran yang diinginkan,
pentamer pada ujung 5’ harus lebih stabil. Jika primer memiliki ujung 3' lebih stabil, maka ikatan ke situs komplemen akan menggantung di tepi. Hal ini dapat mengakibatkan pita sekunder (PBI, 2009).
Repeats dan runs primer yang diperbolehkan pada program Clone Manager Suite 6 adalah kurang dari 3. Pada primer hasil desain, kedua primer telah memenuhi kriteria repeats maupun runs yang diperbolehkan. Adanya pengulangan tiga basa pada dinukleotida maupun nukleotida akan meningkatkan kemungkinan false priming (PBI, 2009). Adanya false primer akan mengakibatkan kesalahan amplifikasi di luar suhu annealing primer sehingga produk PCR yang diinginkan tidak terbentuk. False priming tidak diperbolehkan ada pada primer dan pada primer hasil desain tidak ada false primer.
Desain primer secara in-silico dapat diuji keberhasilannya secara in vitro melalui proses PCR dengan menentukan terlebih dahulu suhu annealing. Suhu annealing adalah salah satu faktor kritis yang mempengaruhi kemampuan primer untuk dapat mengamplifikasi fragmen target. Suhu annealing merupakan suhu yang diperlukan dimana primer menempel dengan template secara stabil (Abd-Elsalam, 2003; Sasmito, 2014). Penentuan suhu yang akan digunakan pada proses amplifikasi fragmen promoter gen inhA diperoleh melalui analisis in silico dengan menggunakan program Clone Manager Suite 6 yaitu 56°C. Melalui proses PCR, dapat diketahui kemampuan primer untuk amplifikasi regio promoter gen inhA. Proses amplifikasi fragmen promoter gen inhA dimulai dari predenaturasi 95°C selama 15 menit, amplifikasi sebanyak 40 siklus dan elongasi pada suhu72°C selama 2 menit. Proses amplifikasi diakhiri dengan elongasi akhir pada suhu 72°C selama 10 menit. Produk PCR kemudian di deteksi melalui proses elektroforesis menggunakan konsentrasi gel agarose 1,3%. Hasil elektroforesis pada Gambar. 4 menunjukkan bahwa primer promoter gen inhA sudah berhasil mengamplifikasi fragmen promoter gen inhA dengan ukuran produk 373 bp yang sesuai dengan hasil desain Clone Manager Suite 6.
Desain primer dengan Clone Manager Suite 6 menghasilkan sepasang primer yang memenuhi kriteria primer yang baik yaitu primer forward 5’-
GGTCGAAGTGTGCTGAGTC-3’ dan
primer reverse 5’-
TGCTCTTCTACCGCCGTGA-3’. Primer
telah mampu mengamplifikasi regio promoter gen inhA secara in vitro dengan ukuran produk sebesar 373 bp.
Penulis mengucapkan terimakasih kepada semua pihak yang telah membantu baik secara langsung mapun tidak langsung sehingga penelitian ini dapat dilaksanakan.
DAFTAR PUSTAKA
Centers for Disease Control and Prevention.
2017. Tuberculosis (TB) : Drug-Resistant TB. http://www.cdc.gov/tb/topic/drtb/def ault.htm. Tanggal Akses 20 Januari 2018.
Kementerian Kesehatan Republik Indonesia. 2016. Tuberculosis. Kementerian
Kesehatan Republik Indonesia : Pusat Data dan Informasi Kementerian Kesehatan Republik Indonesia.
Bollela, V. R., E. I. Namburete, C. S Feliciano, D. Macheque, L.H. Harrison, J. A. Caminero. 2016. Detection of katG and inhA Mutations to Guide Isoniazid and Ethionamide Use for Drug-resistant Tuberculosis. Int J Tuberc Lung Dis. 20(8) : 1099–1104
Katzung, B. G. 2006. Basic and Clinical Pharmacology LO- Edition. San Fransisco: McGraw Hill. P. 515.
Morlock, G. P., B. Metchock, D. Sikes, J. T. Crawford and R. C. Cooksey. 2003. ethA, inhA, and katG Loci of
Ethionamide-Resistant Clinical
Mycobacterium tuberculosis Isolates. Antimicrobial Agent and Chemotherapy. 47 (12) : 3799 – 3805.
38
Discussion and feedback