ISOLASI DNA METAGENOMIK DARI TANAH HUTAN MANGROVE PANTAI SUWUNG BALI
on
ISSN 1907-9850
ISOLASI DNA METAGENOMIK DARI
TANAH HUTAN MANGROVE PANTAI SUWUNG BALI
I Nengah Wirajana*, Darma Asih Yuliana, dan Ketut Ratnayani
Jurusan Kimia FMIPA Universitas Udayana, Bukit jimbaran
*Corresponding author : wirajana@yahoo.com
ABSTRAK
Isolasi DNA metagenomik dari tanah hutan mangrove pantai Suwung Bali telah dilakukan dalam upaya mengeksploitasi potensi biokatalik dari komunitas mikroba untuk menemukan selulase baru. Isolasi DNA total metagenomik dilakukan dengan modifikasi tahapan preparasi dari Marco (2010) dan tahapan ekstraksi DNA dari Amorim et al. (2008). DNA metagenomik hasil isolasi dianalisis secara spektrofotometri dan dengan elektroforesis gel agarosa. Hasil penelitian yang diperoleh menunjukkan bahwa DNA metagenomik dapat diisolasi dari lokasi tersebut. Hasil penelitian menunjukkan DNA total telah dapat diisolasi dengan sukses, namun masih terkontaminasi protein dan asam humat.
Kata kunci : Selulase, DNA metagenomik, tanah hutan mangrove
ABSTRACT
Metagenomic DNA isolation from mangrove forest soils of Suwung Beach-Bali was conducted to exploit the biocatalytic potential of microbial communities for the discovery of novel cellulase. The whole DNA isolation was conducted by modification of preparation step by Marco (2010) and DNA extraction step by Amorim et al (2008). The results of metagenomic DNA isolation were analyzed by spectrophotometry and agarose gel electrophoresis. The results showed that whole DNA was able be isolated successfully, but protein and humic acid were found as contaminant.
Keywords : Cellulase, metagenomic DNA, mangrove forest soil
PENDAHULUAN
Eksplorasi enzim dapat dilakukan dengan pemanfaatan mikroorganisme sebagai penghasil enzim yang memiliki banyak fungsi dan aktivitas. Enzim selulolitik sangat penting dalam mendegradasi limbah organik menjadi produk yang lebih bermanfaat, seperti bahan bakar (Ohmiya et al, 2003). Namun, sekitar 99% dari biodiversitas bakteria lingkungan tidak
dapat dikulturkan dan informasi genom masih banyak yang belum terungkapkan (Amann et al, 1995). Salah satu cara eksplorasi enzim yang kini sedang dilakukan untuk mengungkap informasi genom baru adalah metagenomik, yaitu: langkah baru dalam analisis genom dengan cara mengisolasi DNA genom secara langsung dari lingkungan dalam rangka membangun pustaka metagenomik dari berbagai macam mikroorganisme yang ada dalam lingkungan
tersebut (Handelsman, 2004). Metagenomik memberikan peluang besar dalam penemuan diversitas enzim yang baru karena kita dapat mengeksplorasi genom mikroba secara langsung dari lingkungan habitatnya (Uchiyama & Mizaki, 2009; Schmeisser et al., 2007). Informasi genom selulase yang ditemukan secara metagenomik dapat dimanfaaatkan dengan mudah dan murah untuk mendegradasi biomassa menjadi produk yang berekonomis tinggi yang digunakan secara luas dalam bidang industri (Li et al, 2009).
Salah satu lokasi yang potensial (biodiversitas tinggi) untuk eksplorasi enzim adalah hutan mangrove. Akar mangrove diketahui mampu menahan limbah organik dan partikel endapan yang terbawa arus (Irwanto, 2006) dan tanahnya kaya akan lignoselulosa yang terdiri atas tiga jenis polimer utama, yaitu: selulosa, hemiselulosa dan lignin (Meryandini et al, 2009). Berkaitan dengan berlimpahnya selulosa dalam tanah hutan mangrove, maka kemungkinan besar akan terdapat aktivitas selulase yang mendegradasi biomassa dalam tanah hutan mangrove. Gilna dan Khaleel (2011) telah berhasil menemukan aktivitas selulase ekstraseluler dari jamur Aspergillus fumigatus yang berasal dari tanah hutan mangrove Kerala-India. Isolasi DNA total dari tanah hutan mangrove dilakukan sebagai pendekatan metagenomik dalam menggali potensi selulase dalam tanah hutan mangrove pantai Suwung Bali. Adanya aktivitas selulase dari tanah hutan mangrove tersebut akan berpotensi ditemukannya gen pengkode selulase dalam DNA total metagenomik yang berhasil diisolasi. Penelitian ini merupakan jenis penelitian deskriptif-eksploratif yang menjadi bagian dari penelitian lain (grup riset) yang bertujuan untuk menemukan gen-gen pengkode enzim baru, khususnya selulase.
MATERI DAN METODE
Bahan
Sampel tanah hutan mangrove, Tris-Cl, Na2EDTA.2H2O, Sodium Dodecyl Sulphate, fenol, kloroform, isoamil alkohol, isopropanol, natrium asetat, etanol 95%, agarosa, etidium
bromida, basa Tris, asam asetat glasial, dan loading buffer.
Peralatan
Botol polietilen steril, neraca analitik, botol semprot, hot plate, stirer magnetik, termometer, labu ukur, gelas beaker, ball filler, pipet volume, mikro pipet, tip mikro, , tabung mikro 1,5 mL, High Speed Refrigeratored Micro Centrifuge TOMY MX-301, UV Transiluminator Bioinstrument, spektrofotometer UVmini-1240 single beam merk Shimadzu, dan seperangkat alat elektroforesis horizontal (Mupid 2Plus).
Cara Kerja
Pengambilan Sampel Tanah
Sampel tanah hutan mangrove diambil di tiga lokasi berbeda, yaitu di lokasi A (8o43’38.20”LS), B (8o43’46,18”LS), dan C (8o43’37,38”LS). Masing-masing lokasi diukur terlebih dahulu pH tanah dan suhunya. Sampel diambil pada kedalaman 0-10 cm dan disimpan dalam botol plastik steril.
Isolasi DNA Total Metagenomik
Tahapan Preparasi
Tahapan preparasi ini diambil dari metode preparasi sampel (Marco, 2010). Tanah mangrove dari masing-masing lokasi diambil sebanyak 16 gram untuk ditambahkan dengan 10 mL buffer TE pH 8, kemudian divorteks dan disentrifugasi berulang kali, lalu supernatannya disentrifugasi 15 menit, (8.100 rpm atau 6.000 x g) dan peletnya siap untuk diekstraksi DNA.
Tahapan Ekstraksi DNA
Tahap ekstraksi DNA ini diambil dari metode Amorim et al (2008). Sebanyak 150 μL buffer TE 50/50 pH 8 ditambahkan dan pelet diresuspensi, lalu ditambah glass beads. Suspensi divorteks dan disentrifugasi 3 menit (5000 x g atau 7.400 rpm) berulang kali, lalu ditambahkan 200 μL SDS dan diinkubasi 20 menit pada suhu kamar. Kemudian disimpan 10 menit (-20oC), lalu dipanaskan 4 menit (100 oC) dan didinginkan kembali (-20oC). Sebanyak 500 μL fenol-kloroform-isoamil alkohol ditambahkan, lalu disentrifugasi 10 menit (5000 g atau 7.400 rpm). Supernatan ditambahkan 150 μL isopropaniol dingin dan 15 μL Na-Asetat, lalu
disuspensi dan disimpan 2 jam pada -20oC, lalu disentrifugasi 10 menit (9.100 x g atau 10.000 rpm). Peletnya dicuci dengan EtOH 70% dan dicuci kembali dengan EtOH 95% lalu peletnya diresuspensi dengan 50 μL buffer TE 10/0,1 pH 8 untuk dianalisa secara spektrofotometri (pada λ 230; 260; dan 280 nm) dan dideteksi dengan elektroforesis gel agarosa.
HASIL DAN PEMBAHASAN
Sampel tanah diambil dari kawasan pusat konservasi hutan mangrove pantai Suwung Kauh, Denpasar-Bali. Sampel tanah diambil dari tiga titik lokasi berbeda, seperti yang terlihat dalam Gambar 1.
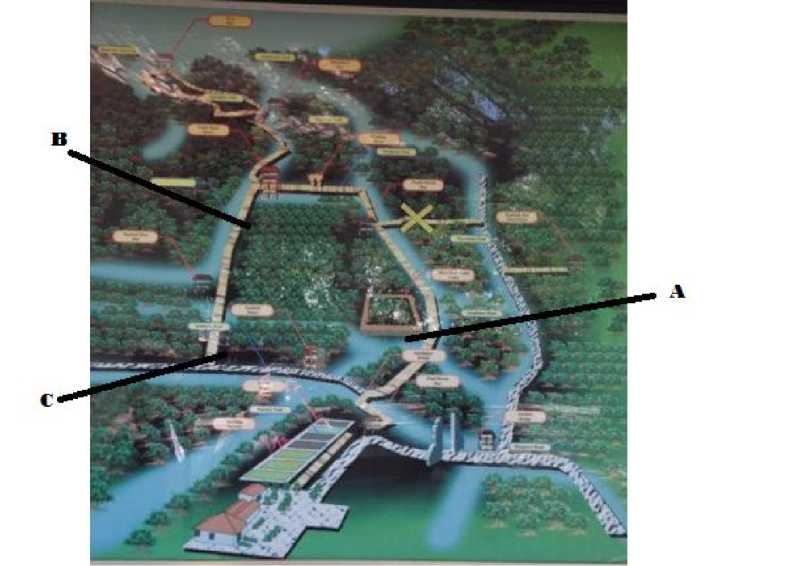
Gambar 1. Peta lokasi pengambilan tanah hutan mangrove di kawasan Pusat Konservasi Mangrove Suwung Kauh Denpasar, Bali. A : letak garis lintang 8o43’38,20” LS; B : letak garis lintang 8o43’46,18” LS; C : letak garis lintang 8o43’37,38” LS
Tabel 1. Kondisi yang diamati pada saat pengambilan tanah hutan mangrove
|
No. Lokasi Waktu Sampling (wita) |
pH Suhu Kondisi tanah yang teramati (oC) |
|
1. A 11.00 |
Cokelat, tidak berbau, air pasang , mengalir. |
|
2. B 11.20 |
Cokelat pekat kehitaman, berbau, air , pasang mengalir. |
|
3. C 11.45 |
Cokelat kehitaman, berbau, air , pasang tergenang/tidak mengalir. |
Pengambilan sampel dilakukan pada tanggal 10 April 2012, pukul 11.00 - 12.00 WITA dengan kondisi air laut pasang pada ketiga lokasi sampling dan cuaca cerah. Kondisi yang diamati saat pengambilan tanah hutan mangrove dapat dilihat pada Tabel 1.
Dari hasil pengamatan saat pengambilan sampel tanah diperoleh data pH tanah A dan B adalah 7, sedangkan pH tanah C adalah 6. Suhu tanah di titik A adalah 30oC, sedangkan untuk titik B dan C adalah 29 oC. Isolasi DNA metagenomik diawali dengan tahapan preparasi. Pada tahapan ini terjadi pelepasan sel-sel dari matriks tanah yang telah dilakukan dengan cara menggetarkan dengan vorteks. Sel-sel yangg terlepas dari matriks tanah berada pada fase supernatan setelah didiamkan sekitar satu jam. Supernatan disentrifugasi sehingga diperoleh endapan coklat yang dianggap sebagai pelet sel. Massa pelet sel dari sampel tanah A, B dan C secara berturut-turut adalah 0,1083 g; 0,2122 g; dan 0,1167 g.
Ekstraksi DNA metagenomik dilakukan dengan cara melisis sel secara mekanik dengan vorteks dan secara kimiawi dengan penambahan
sodium dodesil sulfat (SDS). Pemecahan sel secara mekanik memanfaatkan tumbukan glass beads dengan membran sel ketika divorteks, sedangkan pemecahan sel dengan SDS dilakukan untuk membebaskan DNA dari lemak yang ada pada membran sel. Selanjutnya DNA dapat dimurnikan dari kontaminasi protein dengan cara penambahan fenol-kloroform-isoamil alkohol (24:25:1). Fenol berfungsi melarutkan protein, sedangkan kloroform-isoamil alkohol berperan untuk mendenaturasi protein yang telah terlarutkan oleh fenol tersebut (Sambrook et al, 1989). Na-asetat dapat bereaksi dengan fenol sehingga fenol ikut terendapkan dan mengontaminasi endapan DNA total yang diperoleh. Dari hasil isolasi DNA, diperoleh endapan berwarna coklat. Hal ini dapat disebabkan oleh tingginya kadar asam humat yang terkandung pada endapan asam nukleat yang berhasil diisolasi. DNA diresuspensi dengan buffer TE 10/0,1 pH 8 dan dianalisa secara spektrofotometri dan dideteksi dengan elektroforesis gel agarosa. Hasil pengukuran absorbansi secara spektrofotometri pada λ 230; 260; dan 280 nm ditampilkan pada Tabel 2.
Tabel 2. Absorbansi, rasio A260/A230, dan rasio A260/A280 sampel DNA metagenomik.
|
Sampel |
Absorbansi |
Rasio A260/A230 |
Rasio A260/A280 | ||
|
A230 |
A260 |
A280 | |||
|
A |
0,107 |
0,170 |
0,135 |
1,59 |
1,26 |
|
B |
0,263 |
0,820 |
0,580 |
3,12 |
1,41 |
|
C |
0,064 |
0,520 |
0,355 |
8,12 |
1,46 |
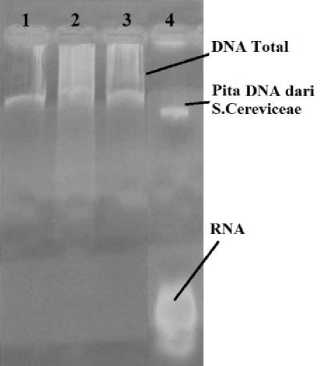
Gambar 2.
Elektroforegram DNA total tanah A, B dan C. (1) DNA total dari tanah A, (2) DNA total dari
tanah B, (3) DNA total dari tanah C, (4) Pita DNA S.cereviceae (kontrol positif)
Molekul DNA murni memiliki rasio A260/A280 yang berkisar pada 1,8 – 2,0 (Sambrook et al, 1989; Boyer, 1993). Kuantitas DNA dapat diukur jika rasio A260/A280 berada di kisaran tersebut. Jika DNA mengalami kontaminasi oleh protein dan senyawa organik seperti asam humat, maka rasio A260/A280 akan menjadi lebih kecil dari 1,8. Dan jika DNA hasil isolasi terkontaminasi oleh fenol rasio A260/A280 lebih besar dari 2,0. Hal ini dapat terjadi karena fenol memiliki serapan maksimal di panjang gelombang 270 nm dan mampu memberi sumbangan kecil serapan di panjang gelombang 260 nm (Linacero et al, 1998). Dari rasio A260/A280 pada Tabel 2 dapat dilihat bahwa ketiga sampel DNA tersebut masih terkontaminasi oleh protein dan asam humat sehingga kadar DNA-nya tidak bisa ditentukan karena rasionya di bawah kisaran 1,8.
Pada hasil elektroforesis gel agarosa, pita DNA dideteksi dengan perendaman gel dalam larutan etidium bromida encer (fluoresens) yang nantinya akan berpendar di bawah sinar UV 254 nm (Sambrook et al, 1989). Konsentrasi terkecil dari DNA yang dapat terbaca berkisar 1-10 ng dengan pengamatan gel di bawah sinar UV (Sharp et al, 1973). Hasil elektroforesis gel agarosa dari DNA total metagenomik yang diisolasi dari masing-masing 16 gram sampel tanah A, B dan C dapat dilihat pada Gambar 2.
Kontrol positif dari DNA S.cereviceae masih terkontaminasi oleh RNA karena dari hasil elektroforesis tersebut masih nampak pendaran (fluoresens) pita di bagian bawah hasil elektroforesis. Dilihat dari hasil elektroforesis sampel DNA lokasi A, B dan C memiliki DNA total yang tidak terkontaminasi oleh RNA. Hal ini dilihat dari bentuk pitanya yang lebar, berekor, dan pita DNA total yang banyak mengumpul di bagian atas, dekat dengan sumur, sedangkan kontaminasi RNA dapat diketahui melalui adanya pendaran di bagian bawah elektroforegram. Secara semi-kuantitatif, pita DNA yang berpendar kuat (tebal) di bawah sinar UV memiliki konsentrasi yang lebih tinggi dibandingkan dengan noda DNA yang tipis (Tenriulo et al, 2001).
SIMPULAN DAN SARAN
Simpulan
DNA metagenomik telah berhasil diisolasi dari tanah hutan mangrove pantai Suwung Bali di tiga lokasi, namun dari hasil analisis speltrofotometri menunjukkan masih mengandung kontaminan asam humat dan protein.
Saran
Pemurnian DNA perlu dilakukan untuk mengurangi kadar asam humat dan protein pada DNA total yang telah berhasil diisolasi. Perlu dilakukan penelitian lebih lanjut tentang pembuatan pustaka metagenom dengan cara kloning DNA total metagenomik tanah hutan mangrove pantai Suwung Bali untuk menemukan gen pengkode selulase yang baru.
UCAPAN TERIMA KASIH
Pada penelitian ini penulis mengucapkan terima kasih kepada Astro Kasih-Yayasan Sampoerna Foundation yang telah mendukung sebagian biaya penelitian ini, serta semua pihak yang telah membantu dalam pelaksanaan penelitian ini.
DAFTAR PUSTAKA
Amann R. I., Ludwig, W., and Schleifer, K. H., 1995, Phylogenetc Identification and In Situ Detection of Individual Microbial Cells Without Cultivation, J.Microbiol. Rev., 59 : 143–69
Amorim, J. H., Macena, T. N. S., Lacerda-Junior, G. V., Rezende, R. P., Dias, J. C. T., Brendel, M., and Cascardo, J. C. M., 2008, An Improved Extraction Protocol for Matagenomic DNA from a Soil of the Brazilian Atlantic Rainforest, Journal Genetic and Moleculer Research, 7 (4): 1226-1232
Ghose, 1987, Measurement Cellulase Activities, J.Pure & Appl.Chem., 59 (2) : 257-268
Gilna, V. V., and Khaleel, K. M., 2011,
Cellulase Enzyme Activity of Aspergillus fumigatus From Mangrove Soil on Lignoscellulosic Substrate, J. Recent Research in Science and Technology, 3 (1) : 132-134
Handelsman J., 2004, Metagenomics:
Application of Genomics to Uncultured Microorganisms, Microbiol. Mol. Biol. Rev., 68 : 669–85
Irwanto, 2006, Keanekaragaman Fauna pada Habitat Mangrove, Yogyakarta
Marco, D. E., 2010, Metagenomics: Theory, Methods and Applications. Academic Press UK, h. 58-59
Meryandini, A., Widosari, W., Maranatha, B., Sunarti T. C, Rahmania, N., dan Satria, H., 2009, Isolasi Bakteri Selulolitik dan Karakterisasi Enzimnya, Makara Sains, 13 (1) : 33-38
Ohmiya, K., Sakka, K., Kimura, T., and Morimoto, K., 2003, Application of
Microbiology Genes to Recalcitrant
Biomass Utilization and Environtmental Conversation, J.Bioscience and
Bioengineering, 95 (6) : 549-561
Otajevwo, F. D., and Aluyi, H. S. A., 2011, Cultural Conditions Necessary for
Optimal Cellulase Yield by Cellulolytic Bacterial Organisms as They Relate to Residual Sugars Released in Broth Medium, J.Modern Applied Science, 3 (5) : 141-151
Sambrook J., Fritsch, E. F., and Maniatis, T. 1989. Molecular Cloning: A Laboratory Manual, 2nd Ed. Could Spring Harbour Laboratory Press, USA
Schmeisser, C., Steele, H., Streit, W. R., 2007, Metagenomics: Biotechnology with
Non-Culturable Microbes, J.Appl Microbiol Biotechnol., 75:955–962
Sharp, P.A., B.Sugden, and J. Sambrook, 1973, Detection of two restriction endonuclease activities in Haemophilus parainfluenzae using analytical agaroseethidium bromide electrophoresis, Biochemistry, 12 : 3055
Uchiyama, T., and Miyazaki, K., 2009,
Functional Metagenomics for Enzyme Discovery: Challenges
Zhang, Percival Y-H., Himmel, E. M., and Mielenz, J. R., 2006, Outlook for
cellulase improvement: Sreening and selection strategies, J.Biotechnology Advances, 24 : 452–481
24
Discussion and feedback