IDENTIFIKASI SPESIES POTYVIRUS PENYEBAB PENYAKIT MOSAIK PADA TANAMAN CABAI (Capsicum frutescens L.) MELALUI SIKUEN NUKLEOTIDA GEN Coat Protein
on
J. Agric. Sci. and Biotechnol.
ISSN: 23020-113
Vol. 6, No. 1, Juli 2017
IDENTIFIKASI SPESIES POTYVIRUS PENYEBAB PENYAKIT MOSAIK PADA TANAMAN CABAI (Capsicum frutescens L.) MELALUI SIKUEN NUKLEOTIDA GEN Coat Protein
I Gede Rian Pramarta1, I Gede Rai Maya Temaja1*), I Dewa Nyoman Nyana1, dan Gede Suastika2
1Program Magister Bioteknologi Pertanian, Program Pascasarjana Universitas Udayana
2Laboratorium Virologi Tumbuhan, Institut Pertanian Bogor Bogor
*) Corresponding author at : Jl. PB. Sudirman Denpasar Bali Indonesia E-mail: tderai@yahoo.com
Abstract
Chili peppers (Capsicum frutescens L.) is one of the important vegetable in the world and one of the leading horticultural commodities in Indonesia. Pepper plants grown in all provinces in Indonesia and got priority for development because it has great potential economic value. These researches was conducted in order to determine the type of infecting Potyvirus on chili and know the proximity nucleotide sequence of Potyvirus infecting chili crop in Kerta village, Payangan, Gianyar, Bali, with the kind of Potyvirus in other areas. This study use RT-PCR technique to detect plant viruses and continued with tracing sequence coat protein gene nucleotide to its proximity to some of the isolates analyzed from GeneBank. The results showed that the infecting Potyvirus chili crop is Chili veinal motle virus species (ChiVMV) with size of approximately 900 bp DNA band, and based on phylogenetic analysis of coat protein gene nucleotide sequence, PayanganIndo isolate had high similarity (93%) and small genetic distance (0.041) with isolate of origin Thailand state.
Keywords: Chili pepper, mosaic, Potyvirus, coat protein.
kadang-kadang pola-pola tersebut menyatu ke tulang daun di dekatnya, leaf cupping dan nekrosis (Sulyo et al., 1995).
Gejala yang disebabkan oleh Potyvirus bervariasi karena gejala yang muncul pada tanaman dipengaruhi oleh inang, strain virus, waktu infeksi dan kondisi lingkungan. Berkaitan dengan hal tersebut, perlu dilakukan penelitian untuk mengidentifikasi penyakit Potyvirus pada tanaman cabai. Identifikasi virus secara molekuler dengan menganalisis sikuen nukleotida serta mencari kedekatan dari sikuen nukleotida virus tersebut dengan sikuen nukleotida homolognya di GeneBank diharapkan dapat digunakan sebagai acuan untuk tindakan pengendalian penyebaran virus selanjutnya. Berkaitan dengan hal tersebut, perlu dilakukan penelitian untuk dapat mengidentifikasi penyakit Potyvirus pada tanaman cabai. Identifikasi virus secara molekuler dengan analisis sikuen nukleotida serta mencari kedekatan dari sikuen nukleotida virus tersebut dengan sikuen nukleotida dari beberapa daerah lain diharapkan dapat digunakan sebagai acuan untuk tindakan pengendalian penyebaran virus.
Tahapan RT-PCR terdiri dari ekstraksi RNA total, sintesis complementary (c) DNA, amplifikasi DNA virus target, dan visualisasi hasil amplifikasi. Total RNA diekstraksi dari jaringan daun tanaman cabai menggunakan Thermo scientific GeneJET Plant RNA Purification Mini Kit. RNA total yang diperoleh selanjutnya ditranskripsikan menjadi DNA komplemen (cDNA) dengan menggunakan teknik reverse transcription-polymerase chain reaction (RT-PCR) pada mesin PCR. Komposisi reagen RT adalah sebagai berikut: 3,7 µl H2O, 2 µl Buffer RT 10x, 0,5µl DTT (dithiothreitol) 50 mM, 0,35 µl dNTP (deoksiribonukleotida triphosphate) 10 mM, 0,35 µl MMuLV (Moloney Murine Leukimia Virus), 0,35 µl RNAse Inhibitor, 0,75 µl Oligo d(T) 10 µM, 0,75 µl Random heksamer, 2 µl template RNA. Reaksi RT dilakukan dalam sebuah Automated Thermal Cycler (Gene Amp PCR System 9700 thermocycler; perkin-Elmercrop., Norwalk, CT) diprogram untuk satu siklus pada suhu 250C selama 5 menit, 420C selama 60 menit, dan 700C selama 15 menit.
Tahapan dilanjutkan dengan amplifikasi cDNA dilakukan dengan teknik PCR dengan pasangan primer yaitu: ChiVMV F Ind (5’- AACCTGAG CGTATAGTTTCA-3’) dan ChiVMV R Ind (5’- TACGCTTCAGCAAG ATTGCT-3’). Kedua primer tersebut merupakan primer yang dapat mengamplifikasi bagian coat protein (CP) virus yang berukuran sekitar 900 bp. Amplifikasi dengan PCR dilakukan sebanyak 35 kali dengan tahapan sebagai berikut : predenaturasi pada suhu 94°C selama 5 menit, denaturasi pada suhu 94°C selama 1 menit, annealing pada suhu 50oC selama 1 menit, sintesis DNA
pada suhu 72°C selama 2 menit, pemanjangan akhir pada suhu 72oC selama 5 menit dan suhu 4 oC untuk suhu penyimpanan (Jan et al., 2000).
-
2.2 Sikuen Nukleotida dan Analisis Filogenetika
Perunutan DNA CP- Potyvirus dari hasil amplifikasi PCR dilakukan di PT. Genetika Sience Indonesia. Sikuen gen coat protein (CP) dianalisis untuk mengetahui tingkat homologi dengan sikuen yang telah dideposit pada GeneBank menggunakan software BLAST (Basic Local Alignment Search Tools) (NCBI 2014). Data sikuen nukeotida yang terpilih kemudian dimodifikasi dan analisis spesifisitas nukleotida dilakukan dengan program multiple alignment, ClustalW dengan software Bioedit V. 7.0.5. Alignment dilakukan dengan membandingkan homologi isolat Potyvirus sampel dengan 11 isolat Potyvirus dari beberapa daerah atau negara dan 1 isolat CMV sebagai pembanding diluar grup (outgroup). Pohon filogenetika dikonstruksi menggunakan program MEGA 5.05 berdasarkan pendekatan Unweighted Pair Group Method with Aritmetic Mean (UPGMA).
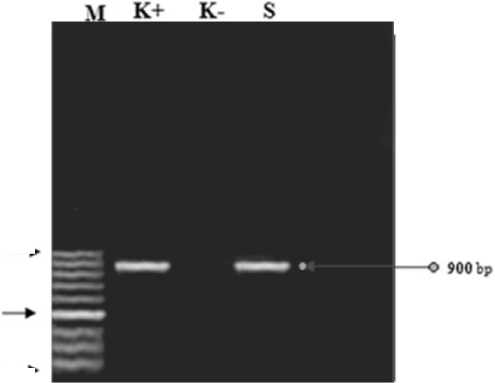
Sampel dengan gejala Potyvirus diambil di kawasan Desa Kerta, Payangan (isolat PayanganIndo), yang kemudian dideteksi dengan RT-PCR menggunakan primer spesifik ChiVMV dengan produk PCR sekitar 900 bp. Pada penelitian ini dengan menggunakan teknik RT-PCR berhasil mengamplifikasi sampel tanaman dari Desa Kerta,Payangan, Gianyar, Bali, dengan produk PCR sekitar 900 bp sesuai dengan analisis primer yang digunakan (Tsai et al., 2008 ; Manzila et al., 2011). (Gambar 1).
IOOO bp →
SOObp
IOObp —►
Gambar 1. Gambar hasil elektroforesis produk RT-PCR isolat PayanganIndo dengan menggunakan primer ChiVMV F Ind dan ChiVMV R Ind. M: 100 bp DNA ladder. K-: control negatif. K+: Kontrol positif (ChiVMV isolate bogor). S: Sampel PayanganIndo.
-
3.2 Analisis Homologi Sikuen Gen Coat Protein Potyvirus
Isolat ChiVMV PayanganIndo kemudian dibandingkan dengan 11 isolat virus ChiVMV yang berasal dari beberapa negara maupun daerah di Indonesia serta satu isolat virus CMV sebagai pembanding outgroup. Untuk tingkat kesamaan antara isolat ChiVMV PayanaganIndo dengan isolat ChiVMV dari daerah maupun negara lain dapat dilihat pada Tabel 1.
Pengujian tingkat homologi antar isolat menunjukan bahwa ChiVMV PayanganIndo mempunyai kesamaan sebesar 99% dengan isolat ChiVMV PatarumanIndo serta mempunyai kesamaan sebesar 93% dengan isolat ChiVMV Thailand. Hasil analisis tersebut membuktikan bahwa isolat-isolat tersebut merupakan kelompok virus yang sama (Frankel et al., 1989). Suatu virus memiliki tingkat kesamaan nukleotida kurang dari 79,4%, virus tersebut termasuk kedalam kelompok Potyvirus lainnya (Tsai et al., 2008).
|
Tabel |
1. Tingkat kesamaan isolat |
ChiVM |
dari beberapa negara. | ||||||||||
|
No |
Isolat/nomor aksesi |
Tingkat Kesamaan (%) | |||||||||||
|
1 |
2 |
3 |
4 |
5 |
6 |
7 |
8 |
9 |
10 |
11 |
12 | ||
|
1 |
PayanganIndo |
- | |||||||||||
|
2 |
PatarumanIndo/ DQ854961.1 |
99 |
- | ||||||||||
|
3 |
CikabayanIndo/ DQ854960.1 |
97 |
97 |
- | |||||||||
|
4 |
BellaryIndia/ EF213685.1 |
94 |
93 |
94 |
- | ||||||||
|
5 |
Thailand/ DQ925442.1 |
93 |
94 |
95 |
93 |
- | |||||||
|
6 |
Vietnam/ DQ925442.1 |
92 |
92 |
93 |
94 |
93 |
- | ||||||
|
7 |
China1/ DQ854950.1 |
89 |
89 |
90 |
92 |
92 |
90 |
- | |||||
|
8 |
China2/ KF738253.1 |
77 |
77 |
77 |
78 |
78 |
77 |
77 |
- | ||||
|
9 |
china3/ HQ317867 |
77 |
77 |
78 |
78 |
79 |
77 |
77 |
99 |
- | |||
|
10 |
China4/ KC693766.1 |
77 |
77 |
78 |
78 |
78 |
77 |
77 |
99 |
99 |
- | ||
|
11 |
Taiwan/ DQ854946.1 |
89 |
89 |
90 |
92 |
92 |
90 |
99 |
77 |
77 |
77 |
- | |
|
12 |
Jepang/ AB012221.1 |
89 |
89 |
90 |
92 |
90 |
90 |
93 |
77 |
78 |
78 |
93 |
- |
Analisis sikuen nukleotida dengan ClustalW menunjukkan bahwa isolat PayanganIndo memiliki homologi yang tinggi dengan isolat-isolat ChiVMV berasal dari negara lain seperti India, Vietnam dan Thailand. Hasil analisis penyejajaran sikuen juga memperkuat hal tersebut, dengan nilai similaritas berkisar 92-94% yang mengindikasikan kemiripan sikuen nukleotida antar isolat yang sangat tinggi (Tabel 1). Hasil analisis similaritas menunjukan bahwa isolat PayanganIndo adalah strain yang sama dengan ChiVMV dari India, Vietnam dan Thailand yang secara geografis berada di Asia Tenggara dan Asia Selatan. Isolat dari Thailand memiliki similaritas paling tinggi dengan isolat yang berasal dari Indonesia.
Jarak genetik merupakan tingkat perbedaan gen diantara populasi atau spesies (NEI, 1987). Jarak genetik antar isolat ChiVMV berkisar antara 0,007-
1,762 (Tabel 2). Jarak genetik menunjukan dekat atau tidaknya hubungan kekerabatan antara sikuen nukleotida yang diamati. Jarak genetik antar isolat ChiVMV yang menginfeksi tanaman cabai dapat dilihat pada Tabel 2. Isolat-isolat tersebut berasal dari beberapa daerah di Indonesia serta berasal dari beberapa negara yang secara geografis berada di benua Asia.
Tabel 2. Matrik jarak genetik isolat ChiVMV yang berasal dari daerah dan negara berbeda berdasarkan runutan gen coat protein.
Jarak Genetik
No. Isolat
1 2 3 4 5 6 7 8 9 10 11 12 13
1. PayanganIndo
2. PatarumanIndo 0.007
3. CikabayanIndo 0.028 0.02
4. BellaryIndia 0.053 0.058 0.059
|
5. Thailand |
0.041 |
0.033 |
0.03 |
0.05 | ||||||||
|
6. Vietnam |
0.069 |
0.069 |
0.064 |
0.058 |
0.055 | |||||||
|
7. China1 |
0.09 |
0.09 |
0.09 |
0.078 |
0.081 |
0.091 | ||||||
|
8. China2 |
0.152 |
0.148 |
0.141 |
0.145 |
0.138 |
0.153 |
0.156 | |||||
|
9. China3 |
0.144 |
0.14 |
0.133 |
0.137 |
0.13 |
0.146 |
0.153 |
0.011 | ||||
|
10. China4 |
0.146 |
0.141 |
0.135 |
0.139 |
0.132 |
0.147 |
0.155 |
0.007 |
0.006 | |||
|
11. Taiwan |
0.093 |
0.093 |
0.092 |
0.077 |
0.081 |
0.088 |
0.012 |
0.16 |
0.156 |
0.158 | ||
|
12. Jepang |
0.099 |
0.099 |
0.097 |
0.08 |
0.092 |
0.101 |
0.056 |
0.149 |
0.147 |
0.146 |
0.054 | |
|
13. IndiaCMV |
1.762 |
1.753 |
1.772 |
1.81 |
1.782 |
1.792 |
1.739 |
1.79 |
1.779 |
1.782 |
1.749 |
1.719 |
Berdasarkan Tabel 2, isolat PayanganIndo mempunyai jarak genetik terkecil (0,007) dengan isolat ChiVMV PatarumanIndo dan mempunyai jarak genetik 0,041 dengan isolat dari Thailand. Analisis filogenetika menunjukkan bahwa hubungan kekerabatan isolat tersebut terbagi menjadi beberapa kelompok (Gambar 2).
98 r Payanganlndo (sampel)
84 Patarumanlndo
96
58
Cikabayanlndo
- Thailand
99
r — Bellarylndia
100 ^—Vietnam
i—Jepang

Chinal
Taiwan
China2
china3
100
China4
IndiaCMV
-I---------------------------------1----------------------------------1---------------------------------1----------------------------------1
0.8 0.6 0.4 0.2 0.0
Gambar 2. Hubungan kekerabatan 13 isolat virus hasil analisis kelompok berdasarkan pola pita DNA dengan metode UPGMA. Skala menunjukkan panjang cabang. Angka pada garpu merupakan persentase tingkat kepercayaan pengelompokan.
Isolat PayanganIndo dan PatarumanIndo berada satu kelompok. Isolat asal Indonesia mempunyai similaritas tinggi dengan isolat yang berasal dari Asia Tenggara dan Asia Selatan yaitu isolat Thailand, BellaryIndia dan Vietnam. Isolat yang berasal dari negara China yaitu isolat China 1, 2, 3 dan 4 mempunyai similaritas tinggi dengan isolat Jepang dan Taiwan. Berdasarkan letak geografis kelompok tersebut termasuk dalam wilayah Asia Timur. Isolat IndiaCMV berada diluar kelompok isolat ChiVMV.
ChiVMV ditularkan melalui vektor secara non persisten dan mekanis, namun melihat hasil analisis filogenetika, dimana similaritas yang tinggi antara isolat PayanganIndo dengan negara lain menunjukan adanya kemungkinan penularan melalui benih karena adanya penyebaran benih lintas negara. Spesies ChiVMV belum ada laporan yang menyatakan penularan melalui benih. Tindak lanjut yang dapat diambil berdasarkan analisis filogenetika tersebut yaitu dengan lebih mewaspadai impor benih pertanian dari negara yang dianggap menjadi asal dari virus tanaman. Beberapa spesies dari golongan Potyvirus dilaporkan dapat ditularkan melalui benih yaitu Zucchini Yellow Mosaic Virus (ZYMV), bean yellow mosaic virus (BYMV), Soybean mosaic virus (SMV), Soybean stunt virus (SSV) dan bean common mosaic virus (BCMV) (Provvidenti and Gonsalves, 1984; Roechan, 1992; Iwaki and Auzay, 1977).
Protein yang terdapat pada CP-ChiVMV berkaitan dengan infektivitas dan penyebaran virus dari sel ke sel serta ke seluruh jaringan tanaman. Kajian yang mendalam tentang peranan CP-ChiVMV perlu dilakukan untuk menentukan strategi pengendalian ChiVMV yang efektif (Manzila et al., 2011).
Berdasarkan hasil penelitian dan pembahasan di atas maka dapat disimpulkan, jenis Potyvirus yang menginfeksi pertanaman cabai di daerah Kerta, Payangan, Gianyar, Bali adalah Chilli veinal mottle virus (ChiVMV). Isolat Chilli veinal mottle virus (ChiVMV) PayanganIndo mempunyai kedekatan dengan isolat ChiVMV yang berasal negara Thailand.
Ucapan Terima kasih
Ucapan terima kasih penulis sampaikan kepada Fitrianingrum Kurniawati, S.P., M.Si dan teman-teman virologi yang sudah membantu mengerjakan teknik PCR selama penelitian di Laboratorium Virologi, Institut Pertanian Bogor.
Daftar Pustaka
Frankel M.J., C.W. Ward, D.D. Shukla. 1989. The use of 3’ Non Coding Nucleotide Sequences in Taxonomy of Potyviruses: Application to Watermelon Mosaic Virus2 and Soybean Mosaic Virus-N. J. Gen. Virol. 70:2775-2783.
Iwaki, M. and H. Auzay. 1977. Virus Diseases of Mungbean in Indonesia. Proc the Ist International Mungbean Symposium. AVRDC. p. 169-172.
Jan, F.J., C. Fagoaga, S.Z. Pang, and D., Gonsalves. 2000. A single chimeric transgene derived from two distinct viruses confers multi-virus resistance in transgenic plants through homology dependent gen silencing. J. Gen. Virol. 81: 2103-2109.
Manzila, I., S.R. Hidayat, I. Mariska, S. Sujiprihati. 2011. Virulensi Empat Isolat Chilli Veinal Mottle Potyvirus Pada Tanaman Cabai (Capsicum Annuum L.) . J. HPT Tropika. 11 (2) : 122-129.
NEI, M. 1987. Molecular Evolutionary Genetic. Columbia University Press. USA.
Opriana, E. 2009. “Metode Deteksi untuk Pengujian Respon Ketahanan Beberapa Genotipe Cabai terhadap Infeksi Chilli Veinal Mottle Potyvirus (ChiVMV)” (tesis). Departemen Proteksi Tanaman IPB.
Provvidenti, R. and Gonsalves, D. 1984. "Occurrence of Zucchini Yellow Mosaic Virus in Cucurbits from Connecticut, New York, Florida, and California". Plant Disease 68 (5): 443–446.
Roechan, M. 1992. Virus-virus pada kedelai (Glycinemax (L) Merr.) di Jawa dan
Lampung: identifikasi, penyebaran, dan kemungkinan pengendaliannya. Disertasi. Universitas Padjadjaran Bandung. 325 p.
Sulandari, S. 2004. Karakterisasi Biologi, Serologi dan Analisis Sidik Jari DNA Virus Penyebab Penyakit Daun Keriting Kuning Cabai. Disertasi SPs IPB. Bogor.
Sulyo, Y., S. Duriat, N. Gunaeni, E. Korlina. 1995. Determination of CMV and CVMV strains in Indonesia. Proceeding of the AVNET II Midterm Workshop Philippines 21-25 Februari 1995. AVRDC.
Tsai, W.S., Y.C. Huang, D.Y. Zhang, K. Reddy, S.H. Hidayat, W. Srithongchai, S.K. Green, F.J. Jan. 2008. Molekular characterization of the CP gene and 3’UTR of Chilli veinal mottle virus from South and Southeast Asia. Plant Pathology 57: 408-416.
https://ojs.unud.ac.id/index.php/JASB
34
Discussion and feedback