DESAIN DNA PRIMER SECARA IN SILICO SEBAGAI PENDETEKSI MUTASI GEN gyrA Mycrobacterium tuberculosis UNTUK METODE POLYMERASE CHAIN REACTION
on
Cakra Kimia (Indonesian E-Journal of Applied Chemistry)
Volume 6, Nomor 1, Mei 2018
DESAIN DNA PRIMER SECARA IN SILICO SEBAGAI PENDETEKSI MUTASI GEN gyrA Mycrobacterium tuberculosis UNTUK METODE POLYMERASE CHAIN REACTION
Luh Vida Sasmitha1, Putu Sanna Yustiantara1,2 dan Sagung Chandra Yowani1,2
-
1Jurusan Farmasi, FMIPA, Universitas Udayana, Bukit Jimbaran, Bali-Indonesia, 80361 sasmithavida@gmail.com
-
2Kelompok Studi MDR-TB & XDR-TB, FMIPA, Universitas Udayana, Bukit Jimbaran, Bali-Indonesia, 80361 putuyustiantara@yahoo.com cyowani@gmail.com
ABSTRAK: Penelitian ini bertujuan untuk mendesain DNA primer yang digunakan untuk mengamplifikasi gen gyrA dengan metode PCR. Gen gyrA bertanggung jawab dalam resistensi fluorokuinolon yang merupakan inti regimen dari MDR-TB. Resistensi fluorokuinolon merupakan penanda terjadinya Extensively Drugs Resistance Tuberculosis (XDR-TB). XDR-TB merupakan tuberkulosis yang terjadi akibat adanya resistensi terhadap isoniazid, rifampisin dan fluorokuinolon serta terhadap salah satu obat anti tuberkulosis lini kedua dalam bentuk injeksi (amikasin, kanamisin dan kapreomisin). Pada penelitian ini desain primer dilakukan secara in silico dengan menggunakan program Clone Manager Suite 6 (University of Groningen). Urutan nukleotida gen gyrA Mycrobacterium tuberculosis yang digunakan sebagai cetakan (template) diakses dari website www.ncbi.nlm.niv.gov (GenBank : AL123456.3). Primer dipilih berdasarkan kriteria dari Clone Manager Suite 6 (University of Groningen), meliputi: panjang primer, %GC, Tm (melting temperature), stabilitas, repeats, runs, interaksi primer (dimers dan hairpins) dan false priming. Hasil penelitian mendapatkan sepasang primer dengan panjang masing-masing 19 basa, yaitu primer forward 5’-GCAGCTACATCGACTATGC-3’ (3-S1) dan primer reverse 5'-GGCTTCGGTGTACCTCATC-3’ (4-S1). Pasangan primer ini mampu mengamplifikasi sekuens gen gyrA sebesar 320 pb (nukeotida 90-380) dengan kondisi PCR pada suhu 95°C selama 15 menit untuk proses predenaturasi, diikuti 40 siklus amplifikasi (denaturasi pada suhu 94°C selama 1 menit, annealing pada 58°C selama 1 menit 20 detik, elongasi pada suhu 72°C selama 2 menit) serta proses elongasi akhir dilakukan pada suhu 72°C selama 10 menit.
Kata kunci: Primer, in silico, gyrA, PCR
ABSTRACT: The aim of this study is to design DNA primer that amplifying gyrA gene using PCR method. The gyrA gene is responsible to fluoroquinolone resistance which is the main drug of MDR-TB regimen. Fluoroquinolone resistance is marker of Extensively Drugs Resistance Tuberculosis (XDR-TB). XDR-TB is a type of tuberculosis (TB) that is resistant to at least isoniazid, rifampicin and fluoroquinolone and/or to second-line anti-tuberculosis drugs (amykacin, kanamycin and capreomycin). In this study, primer design were reconstruct in silico using Clone Manager Suite 6 program (University of Groningen). Nucleotide sequence of gyrA accessed from www.ncbi.nlm.niv.gov (GenBank: AL123456.3). The primer was chosen based on the criteria of Clone Manager Suite 6 (university of Groningen), including primer length, %GC, Tm (melting temperature), stability, repeats, runs, primer interaction (dimers and hairpins) and false priming. The result of this research were a pair of primers which each length is 19 bases, e.g. forward primer 5’-GCAGCTACATCGACTATGC-3’ and reverse primer 5’-GGCTTCGGTGTACCTCATC 3’.
PCR product of these primers is 320 pb (nucleotide 90-380), in predenaturation at 95°C for 15 minutes and followed by 40 cycles of amplificarion (denaturation at 94°C for 1 minutes, annealing at 58°C for 1 minutes 20 seconds, and extension at 72°C for 2 minutes) with final extension at 72°C for 10 minutes.
Keyword: Primer, in silico, gyrA, PCR
MDR-TB (Multidrug Resistance Tuberculosis) adalah tuberkulosis yang disebabkan oleh resistensi yang dialami oleh Mycobacterium tuberkolis terhadap setidaknya dua obat anti tuberkulosis (OAT) lini pertama yang efektif yaitu isonoazid (INH) dan rifampisin (RIF) [1]. Di Indonesia, kasus MDR TB terjadi sebanyak 15.380 kasus pada tahun 2009 hingga triwulan kedua tahun 2015 [2]
Network of Supranasional Reference Laboratories melakukan survey yang menyatakan pada tahun 2000-2004 telah terjadi resistensi terhadap OAT lini kedua yang disebut dengan XDR-TB. Extensively Drugs Resistant Tuberculosis (XDR-TB) didefiniskan sebagai MDR-TB yang mengalami resistensi terhadap lebih dari 3 regimen dari total 6 regimen OAT [1]. XDR-TB merupakan tuberkulosis yang disebabkan oleh bakteri yang resisten terhadap INH dan RIF (MDR-TB) serta ditambah resistensi terhadap salah satu dari OAT lini kedua dalam bentuk injeksi (amikasin, kanamisin dan kapreomisin) dan fluorokuinolon [3]. WHO, 2016 menyebutkan bahwa telah terjadi sebanyak 7579 kasus Extensivelydrug resistant Tuberculosis (XDR-TB) pada 74 negara di tahun 2016. Pengobatan yang diberikan untuk pasien resistensi tuberkulosis khususnya strain XDR-TB sangat terbatas, selain itu sulitnya mendiagnosa pasien dengan XDR-TB juga menyebabkan terjadinya peningkatan kasus dari XDR-TB [4]
Fluorokuinolon merupakan obat yang paling penting diberikan kepada pasien yang telah mengalami MDR-TB, hal ini karena fluorokuinolon memberikan hasil terapi yang lebih baik dan memililiki toksisitas yang rendah dibandingkan
dengan kelompok obat anti tuberkulosis lini kedua lainnya [5]. Fluorokuinolon merupakan golongan antimikroba sintetik yang menghambat gyrase DNA dan topoisomerase IV disebagian besar spesies bakteri dan akan menyebabkan kematian sel bakteri (Mdluli and Ma, 2007). Mutasi pada daerah gyrA merupakan penentu terjadinya resistensi fluorokuinolon di M. tuberculosis. Mutasi pada gyrA terdeteksi sebanyak 25,1 % dari strain multidrug tuberculosis dari 81,5% isolat yang mengalami resistensi fluorkuinolon [6]
Daerah mutasi yang menyebabkan resistensi fluorokuinolon disebut daerah QRDR (Quinolone resistance determining region) yang terletak antara asam amino posisi 67 dan 106 [7]. GyrA merupakan sub-unit I dari gyrase DNA (DNA topoisomerase II). Mutasi pada daerah QRDR gyrA terjadi pada kodon 90, 91, 94 yang menyebabkan terjadinya resistensi terhadap fluorokuinolon [8]. Li, et al., 2014 menyebutkan pada kodon 90 terjadi perubahan nukleotida GCG (Alanin) menjadi GTG (Valin) dan mutasi yang terjadi pada kodon 94 adalah GAC (Asam aspartat) menjadi GGC (Glisin) dan Asam aspartat (GAC) menjadi AAC (Asparagin).
Pada penelitian sebelumnya telah dilakukan deteksi mutasi pada gen inhA dengan metode Polymerase Chain Reaction (PCR) [9]. PCR memiliki keuntungan dapat menghasilkan jumlah copy DNA template yang sangat banyak dengan waktu yang singkat. Pada proses PCR diperlukan beberapa bahan yaitu template DNA, primer, dNTP, buffer, dan enzim polymerase. Keberhasilan dalam prose PCR tergantung dari primer yang digunakan, primer berfungsi sebagai pembatas fragmen DNA yang akan diamplifikasi dan sekaligus menyediakan
gugus hidroksi (-OH) pada ujung 3’ yang diperlukan untuk proses eksistensi DNA[10]. Dalam merancang dan menentukan primer terbaik diperlukan beberapa kriteria yang harus dipenuhi. Perancangan primer merupakan suatu langkah awal untuk memperoleh primer terbaik yang dapat digunakan dalam proses PCR. Perancangan primer ini menggunakan metode in silico berdasarkan urutan DNA yang telah diketahui ataupun dengan urutan protein yang dituju. Data urutan DNA atau protein bisa didapatkan dari database GenBank [10].
-
2. PERCOBAAN
-
2.1 Bahan dan Peralatan
-
Gen gyrA M. tuberculosis
Desain primer DNA dilakukan dengan memasukkan data sekuen wild-type sensitif gen gyrA M. tuberculosis yang diunduh dari database URL:// www.ncbi.nlm.nih.gov dengan kode Genbank: AL123456.3
Software
Peralatan yang digunakan dalam penelitian ini antara lain laptop dengan spesifikasi Windows 10 64 bit dan software Clone Manager Suite 6 untuk mendesain serta menganalisis hasil rancangan primer DNA.
Berikut adalah tahapan yang dilakukan pada penelitian ini : 3.
-
1. Menginstal program Clone Manager
Suite 6 pada prangkat komputer yang dimiliki
-
2. Mengunduh sekuen gen gyrA dari situs www.ncbi.nlm.nih.gov dan
menyimpannya dalam bentuk file
notepad.
-
3. Membuka program Clone Manager Suite 6, kemudian pilih menu bar primer dan pilih design
-
4. Muncul kotak dialog untuk memasukkan sekuen yang telah diunduh sebelumnya. Masukkan sekuen gen gyrA yang telah
diunduh hingga muncul kotak dialog design primer
-
5. Kemudian pada kotak dialog design primer dimasukkan panjang primer forward dan reverse (19 basa); target region dilengkapi dengan posisi basa yang diinginkan untuk posisi awal dan akhir dari region taget (90-360) lalu pilih ok.
-
6. Lalu akan muncul kotak dialog rank yang menunjukkan peringkat
rekomendasi primer yang terbaik dari program Clone Manager Suite 6.
-
7. Langkah 3 – 6 dapat diulangi sehingga nantinya diperoleh primer yang terbaik sesuai dengan kriteria.
Amplifikasi Fragmen gen gyrA dengan teknik PCR
Template DNA yang digunakan adalah isolat klinis P154 dan P123 MDR-TB. PCR dikondisikan pada suhu pada suhu 95°C selama 15 menit untuk proses predenaturasi, diikuti 40 siklus amplifikasi dengan denaturasi pada suhu 94°C selama 1 menit, suhu annealing yaitu 58°C selama 1 menit 20 detik, elongasi pada suhu 72°C selama 2 menit dan proses elongasi akhir dilakukan pada suhu 72°C selama 10 menit.
Deteksi Produk PCR
Produk PCR dideteksi dengan menggunakan elektroforesis gel agarosa 1,5% b/v, pada tegangan 50 volt selama 70 menit.
HASIL DAN PEMBAHASAN
Pada penelitian ini dilakukan desain primer secara in silico menggunakan program Clone Manager Suite 6 (University of Goreningen). Primer ini merupakan primer yang akan digunakan pada proses amplifikasi pada gen gyrA isolat MDR-TB yang ada di Bali. Desain primer dilakukan dengan data sekuen DNA gen gyrA M. tuberculosis GenBank : AL123456.3 yang diperoleh melalui situs URL: www.ncbi.nml.nih.gov. Primer
didesain pada region 90 hingga 380 pb. Pemilihan region amplifikasi ini dilakukan
dengan tujuan untuk mencakup daerah yang telah mengalami mutasi pada penelitian sebelumnya yaitu pada kodon 90, 91 dan 94 [8]. Selanjutkan dimasukkan panjang primer dimulai dari 18-30 basa didasarkan pada pertimbangan kombinasi acak yang mungkin ditemukan pada satu urutan genom [10]. Jika primer yang dihasilkan memiliki panjang kurang dari 18 basa, primer akan mudah menyebkan terjadinya mispriming. Mispriming adalah penempelan primer pada tempat yang tidak diinginkan [9]. Panjang primer lebih dari 30 basa tidak akan menunjukkan spesifitas yang lebih tinggi dan akan mengakibatkan terjadi proses hibridasi dengan primer lain sehingga akan menghambat terbentuknya polimerasi DNA [10]. Pada desain primer yang dilakukan panjang primer yang dipilih sepanjang 19 basa. Program Clone Manager Suite 6 memberikan beberapa rekomendasi primer, kemudian dilakukan analisis pada masing-masing primer sehingga diperoleh primer terbaik untuk proses amplifikasi gyrA. Rekomendasi primer dari Program Clone Manager Suite 6 ditunjukkan pada Gambar 1.
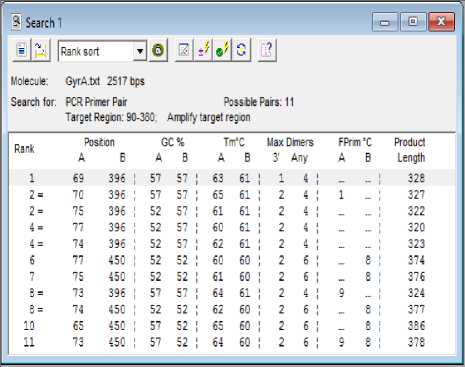
Gambar 1. Rekomendasi Primer dari
Program Clone Manager Suite 6
Hasil yang diperoleh meliputi sepasang primer yang terdiri dari primer forward 5’-GCAGCTACATCGACTATGC-3’ dan primer reverse 5'-GGCTTCGGTGTACCTCATC 3'.
Pasangan primer ini dapat mengamplifikasi sekuens gen gyrA sepanjang ±320 pb. Berikut merupakan analisis hasil desain primer pada program Clone Manager Suite 6 (Tabel 1). Dapat dilihat pada Gambar 2 bahwa primer yang di desain telah menghasilkan hasil yang sesuai dengan kriteria primer yang baik.
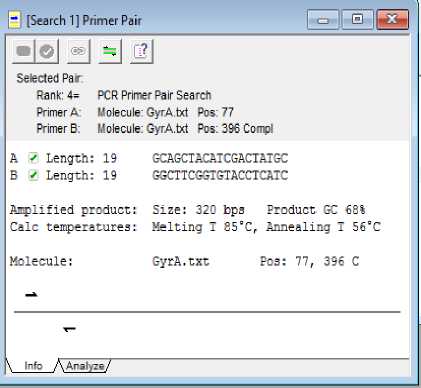
Gambar 2. Hasil design primer
Persentase GC yang dimiliki oleh primer forward sebesar 52% dan primer reverse sebesar 57 %. Persentase GC yang baik berkisar antara 40-60%. Persentase GC adalah kandungan jumlah basa G (guanin) dan C (sitosin) yang dapat mempengaruhi Tm suatu primer, selain itu persentase GC juga mempengaruhi ikatan antar untai pada DNA. Kandungan GC tinggi akan mempersulit pemisahan rantai untai ganda pada primer dan template [9]. Suatu primer yang memiliki kandungan GC rendah tidak akan mampu untuk menempel secara efektif pada target sehingga akan terjadi penurunan efisiensi proses PCR.
Tm adalah suhu dimana 50 % untaian ganda DNA telah terpisah, nilai Tm yang direkomedasikan berkisar antara 5065°C [6]. Tm pada sepasang primer ini adalah 60°C pada primer forward dan 61°C pada primer reverse dengan selisih suhu 1°C. Selisih Tm yang disarankan tidak lebih dari 5°C, apabila lebih tinggi akan
menyebabkan penurunan proses amplifikasi [10].
Tabel 4.1 Hasil Desain Primer untuk Amplifikasi Gen GyrA
|
Kriteria |
3-S1 |
4-S1 |
Acuan pada Clone Manager Suite 6 |
|
Panjang |
19 |
19 |
18-22 |
|
Persen (%) GC |
52 |
57 |
50-60 |
|
Tm (°C) |
60 |
61 |
55-80 |
|
Dimer pada |
2 |
1 |
<3 |
|
ujung 3’ | |||
|
Dimer selain |
4 |
4 |
<7 |
|
ujung 3’ | |||
|
Stabilitas (kcals) |
1.9 |
1.8 |
≥1.2 kcals |
|
Runs |
1 |
2 |
<3 |
|
Repeats |
Tidak ada |
2 |
<3 |
|
Hairpains |
Tidak ada |
Tidak ada |
Tidak ada |
|
False priming |
Tidak ada |
Tidak ada |
Tidak ada |
Keterangan:
Primer forward (3-S1):
5’-GCAGCTACATCGACTATGC-3’
Primer reverse (4-S1):
5'-GGCTTCGGTGTACCTCATC-3'
Hasil desain primer yang dilakukan memiliki dimer pada ujung 3’ sebanyak 2 pb pada primer forward dan 1 pb pada primer reverse. Pasangan primer sebaiknya tidak terdapat dimer. Dimer ini akan mempengaruhi efisiensi proses penempelan pada target [9]. Pada Clone Manager suite 6 batas toleransi dimer pada ujung 3’ adalah <3 pb. Pasangan primer ini memiliki dimers any sebanyak 4 pasang basa pada primer forward dan reverse. Dimers-any adalah terjadinya dimer pada daerah selain 3’. Dimers any ini akan menyebabkan berkurangnya konsentrasi
primer yang dibutuhkan untuk proses amplifikasi [12]. Batas dimer pada daerah selain pada ujung 3 adalah <7 pb.
Stabilitas ujung 5’ harus lebih besar dibandingkan dengan stabilitas ujung 3’. Hal ini akan mempengaruhi efisiensi dari proses penempelan dan pemanjangan primer. Stabilitas pada ujung 3’ lebih tinggi dari 5’ akan menyebabkan terjadinya false priming [13,14]. Batas stabilitas yang dipersyaratkan oleh Clone Manager Suite 6 adalah ≥ 1,2 kcals. Primer yang dihasilkan memiliki stabilitas sebesar 1,9 dan 1,8 kcals sehingga telah memenuhi kriteria
Primer yang dihasilkan memiliki runs sebanyak 1 pb pada primer forward dan 2 pb pada primer reverse. Runs dan repeats maksimal terjadi pada 4 pasang basa [2]. Runs dan repeats yang dimiliki primer akan menyebabkan terjadinya false priming, sehingga pengulangan basa ini harus dihindari. False priming atau kesalahan penempelan primer di luar suhu annealing dapat mengakibatkan terjadinya kesalahan pembentukan produk pada suhu tertentu sehingga produk yang diperoleh tidak sesuai dengan hasil yang diinginkan [10]. Hal lain yang harus dihindari adalah kondisi hairpin. Hairpins adalah kondisi dimana ujung-ujung primer saling berkomplemen. Interaksi hairpins pada primer tidak diperbolehkan sama sekali. Pada pasangan primer yang dihasilkan tidak terdapat kondisi hairpin maupun false priming.
Selanjutnya dilakukan amplifikasi terhadap pasangan primer yang dihasilkan melalui proses PCR dan deteksi produk PCR dengan teknik elektroforesis. PCR dikondisikan pada suhu 95°C selama 15 menit untuk proses predenaturasi, diikuti 40 siklus amplifikasi (denaturasi pada suhu 94°C selama 1 menit, suhu annealing yaitu 58°C selama 1 menit 20 detik, elongasi pada suhu 72°C selama 2 menit) dan proses elongasi akhir dilakukan pada suhu 72°C selama 10 menit. DNA templat yang digunakan adalah P154 dan P123 M.
Multidrug Resistance Tuberculosis. Kemudian dilakukan proses deteksi produk PCR dengan menggunakan elektroforesis gel agarosa 1,5% dengan tegangan 50 volt selama 70 menit. Hasil yang diperoleh berupa elektroforegram yang ditunjukkan pada Gambar 3.
Hasil dari elektroforegram menunjukkan bahwa primer 3-S1 dan 4-S1 telah berhasil mengamplifikasi gen gyrA pada isolat MDR-TB dengan ukuran produk ±320 pb sesuai dengan hasil analisis secara in silico.
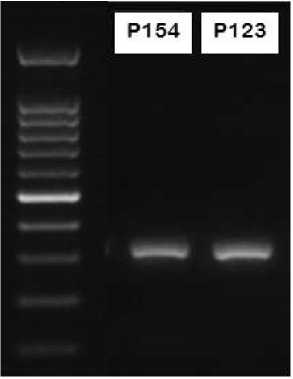
Gambar 3. Hasil elektroforesis isolat P154 dan P123
-
1. Desain primer yang dibuat secara in silico menghasilkan primer terbaik yaitu primer forward 5’-
GCAGCTACATCGACTATGC-3’ (3-S1) dan primer reverse 5'-
GGCTTCGGTGTACCTCATC 3' (4-S1)
-
2. Pasangan primer berhasil mengamplifikasi daerah gen gyrA dengan ukuran ±320 pb sesuai dengan hasil analisi secara in silico yang dihasilkan pada program Clone Manager Suite 6
-
[1] Shah, S., et al. 2007. Worldwide Emergence of Extensively Drugresistant Tuberculosis. Emerging
Infectious Diseases. 13 (3): 380-387
-
[2] Kementerian Kesehatan Republik Indonesia. Infodatin: Tuberculosis Temukan Obati Sampai Sembuh. Jakarta: Pusat Data Dan Informasi Kementerian Kesehatan RI
-
[3] WHO, 2015. Multidrug Resitant Tuberculosis (MDR-TB). World Health Organization. Tanggal Akses 4 Oktober 2016. Tanggal Terbit November 2015.
-
[4] Extensively Drug-Resistant
Tuberculosis As A Cause Of Death In Patients Co-Infected With
Tuberculosis And HIV In A Rural Area Of South Africa. Available At: www.Thelancet.com
-
[5] Caminero, J. A., G. Sotgiu., A. Zumla., G. B. Miglori. 2010. Best drug treatment for multidrug-resistant and extensively drug-resistant tuberculosis. Available At: www.Thelancet.com
-
[6] WHO. 2012. Incidences. Global Tuberculosis Report 2012. Available At: Http://Www.Who.Int/Tuberculosis. (Cited October 3, 2016).
-
[7] Hooper, D. C. 2003. Mechanisms Of Quinolone Resistance. Quinolone Antimicrobial Agents, 3rd
Ed.Washington, D.C: ASM Press
-
[8] Devasia, R., A. Blackman, S. Eden, H. Li, F. Maruri, A. Shintani, C. Alexander, A. Kaiga, C. W. Stratton, J. Warkentin, Y. Tang, And T. R. Sterling. 2011.High Proportion Of Fluoroquinolone-Resistant
Mycobacterium Tuberculosis Isolates With Novel Gyrase Polymorphisms And A Gyra Region Associated With Fluoroquinolone Susceptibility.
Journal Of Clinical Microbiology. P. 1390–1396
-
[9] Maitri, L. K. B., I. N. Wirajana., S. C., Yowani, 2016. Desain Primer Untuk Amplifikasi Fragmen Gen Inha Isolat 134 Multidrug Resistance
Tuberculosis (Mdr-Tb) Dengan Metode Polymerase Chain Reaction. Cakra Kimia. Vol 3 No 2: 89-95
-
[10] Handoyo, D. Dan A. Rudiretna. 2000. Prinsip Umum Dan Pelaksanaan Polymerase Chain Reaction (PCR) (General Principles And
Implementation Of Polymerase Chain Reaction). Unitas. Vol. 9 (1) : 17-29.
-
[11] Sasmito, D. E.K., Rahadian, K., Izzati, Muhimmah. 2014. Karakteristik Primer pada Polymerase Chain Reaction (PCR) untuk Sekuensing DNA: Mini Review. Seminar Nasional Informatika Medis (SNIMed) V: Yogyakarta
-
[12] Borah, P. 2011. Primer Designing For PCR. Science Vision. 11 (3): 134-136
-
[13] Bartlett, J. M. S. And D. Stiring. 2003. Methods In Molecular Biology. Vol. 226: PCR Protocols 2nd Editions. Totowa, NJ: Human Press Inc. P. 81604
-
[14] Maitri, L. K. B., I. N. Wirajana., S. C., Yowani, 2016. Desain Primer Untuk Amplifikasi Fragmen Gen Inha Isolat 134 Multidrug Resistance Tuberculosis (MDR-Tb) Dengan Metode
Polymerase Chain Reaction. Cakra Kimia. Vol 3 No 2: 89-95
-
[15] Li, J., Xu. G., Tao, L., Jie, W., Gang, S., Qinyun, L., Yuan,J., Yangyi, Z., Jian, M., Qian, G. 2014. Association Of Gyra/B Mutations And Resistance Levels To Fluoroquinolones In Clinical Isolates Of Mycobacterium
Tuberculosis. Emerging Microbes And Infections. 3 (19):1-5
69
Discussion and feedback