AMPLIFIKASI DAN IDENTIFIKASI MUTASI REGIO PROMOTER inhA PADA ISOLAT Mycobacterium tuberculosis MULTIDRUG RESISTANCE DENGAN TEKNIK POLYMERASE CHAIN REACTION
on
Cakra Kimia (Indonesian E-Journal of Applied Chemistry)
Volume 2, Nomor 2, Oktober 2014
AMPLIFIKASI DAN IDENTIFIKASI MUTASI REGIO PROMOTER inhA PADA ISOLAT Mycobacterium tuberculosis MULTIDRUG RESISTANCE DENGAN TEKNIK POLYMERASE CHAIN REACTION
Devita Kusdianingrum1, Sanna Yustiantara1,2, Sagung Chandra Yowani1,2* 1Jurusan Farmasi, Fakultas MIPA, Universitas Udayana, Bali-Indonesia 2Kelompok Studi MDR dan XDR-TB, Fakultas MIPA, Universitas Udayana, Bali-Indonesia *cyowani@yahoo.com
ABSTRAK: Sekitar 8-20% isolate M. tuberculosis yang resisten terhadap isoniazid diketahui telah mengalami mutasi pada posisi regio promoter inhA [1]. Untuk memperoleh titik mutasi pada regio promoter, maka amplifikasi fragmen target perlu untuk dilakukan. Tujuan dilakukannya penelitian ini adalah untuk mengamplifikasi regio promoter inhA, mengetahui ada tidaknya mutasi dan jenis mutasi pada isolat 134 MDR-TB. Tahap isolasi DNA dilakukan menggunakan metode Boom yang telah dimodifikasi. Fragmen target diamplifikasi dengan teknik PCR menggunakan sepasang primer (forward primer 5’ ACATACCTGCTGCGCAAT 3’ dan reverse primer 5’ CTCCGGTAACCAGGACT GAA 3’). Amplikon disekuensing secara satu arah menggunakan forward primer. Analisis homologi dilakukan menggunakan program online BLASTn, sementara identifikasi mutasi dilakukan menggunakan software MEGA4. Hasil penelitian menunjukkan bahwa analisis homologi isolate 134 terhadap M. tuberculosis H37Rv adalah sebesar 99%. Tahap analisis mutasi menemukan terjadinya perubahan sitosin menjadi timin (C÷T) pada posisi -15 isolat 134 MDR-TB.
Kata kunci: M. tuberculosis, regio promoter inhA, PCR, homologi, mutasi
ABSTRACT: Approximately 8-20% M. tuberculosis isolates that are resistant to isoniazid habe been known to have a mutation in inhA promoter region [1]. To find the mutation in inhA promoter region, it is necessary to carry out the amplification of the target fragment. The purpose of this research were to amplify the inhA promoter region and to find out if there is a mutation and type of mutation at MDR-TB isolate. DNA isolation was done by a modified Boom method. Target fragment was amplified by a pair primer (forward primer 5’ ACATACCTGCTGCGCAAT 3’ and reverse primer 5’ CTCCGGTAACCAGGACT GAA 3’ using Polymerase Chain Reaction (PCR) technique. Amplicon was sequenced in one forward direction. Homology analysis was conducted by online BLASTn program, while the mutation was identified by MEGA4. The result of this research showed that homology analysis of 134 was homolog by 99% of M. tuberculosis H37Rv. The mutation of cytosine to thymine (C÷T) was found occurring at position -15 of isolate 134 MDR-TB.
Keywords: M. tuberculosis, inhA promoter region, PCR, homology, mutation
Penyakit tuberkulosis (TB) merupakan masalah kesehatan di dunia. Penyakit ini menempati peringkat kedua penyebab kematian karena infeksi setelah HIV [2]. Di Provinsi Bali, khususnya pada tahun 2011
ditemukan sebanyak 1450 kasus TB paru dan 513 (35%) diantaranya ditemukan di Kota Denpasar [3]. Penanganan TB menghadapi tantangan baru dengan munculnya Multidrug resistance TB (MDR-TB). MDR-TB
didefinisikan sebagai penyakit TB yang disebabkan oleh M. tuberculosis yang resisten merupakan obat lini pertama TB [2].
Isoniazid (INH) merupakan obat lini pertama TB yang telah digunakan sejak lama. Resistensi terhadap INH dapat muncul akibat adanya mutasi pada inhA. Studi mutasi terhadap inhA menemukan bahwa dari seluruh isolat M. tuberculosis yang mengalami resistensi terhadap INH (INHr) mengalami mutasi pada regio promoter inhA sebesar 820% dan pada daerah Open Reading Frame (ORF) sebesar 0-5% [1]. Sekitar 66% isolate mengalami mutasi pada posisi -15 regio promoter inhA [4]. Persentase terjadinya mutasi pada regio promoter yang lebih tinggi dibandingkan ORF menyebabkan penelitian mengenai identifikasi mutasi pada regio promoter inhA menjadi penting untuk dilakukan.
Pada penelitian ini dipilih teknik Polymerase Chain Reaction (PCR). Teknik ini mampu mengamplifikasi segmen DNA sehingga dapat menyediakan DNA dalam jumlah yang cukup untuk dideteksi menggunakan sekuensing.
Di Indonesia khususnya Provinsi Bali, penelitian mengenai amplifikasi dan identifikasi mutasi pada regio promoter inhA M. tuberculosis belum dilakukan. Pada penelitian ini akan dilakukan amplifikasi, identifikasi mutasi serta perubahan basa yang terjadi pada isolate 134 MDR-TB. Data yang diperoleh dari penelitian ini akan digunakan dalam usaha melengkapi database mutasi regio promoter inhA isolat M. tuberculosis MDR-TB di Indonesia khususnya Bali.
Bahan yang digunakan antara lain isolat M. tuberculosis MDR, larutan pelisis L6 (GuSCN, Tris-HCl, EDTA, dan Triton-X), washing buffer L2 (GuSCN dan Tris HCl), suspensi diatom, etanol 70%, aseton, aquadest, sepasang primer (forward dan
setidaknya terhadap isoniazid dan rifampisin yang
reverse), buffer TE, H2O, PCR mix, gel agarosa 1,5%, marker 100 bp DNA ladder, TBE, dan EtBr. Peralatan yang digunakan yaitu microwave, vortex (Maxi Mix II®), inkubator (BINDER®), biomedical freezer, shaker rotator (HEALTH® tipe H-SR-200), sentrifuse (Biofuge Primo R®), biological safety cabinet class II type A2 (ESCO®), micro tube, tabung eppendorf 1,5 mL, thermal cycler (Applied Biosystem Veriti 96®), alat elektroforesis (chamber elektroforesis, comb, tray), UV transiluminator Gel Doc XR System (BioRad®) serta sequencer.
Tahap isolasi dilakukan menggunakan metode Boom yang telah dimodifikasi. Sel dilisiskan menggunakan larutan pelisis L6 yang ditambahkan suspensi diatom. Dihomogenkan 100 rpm selama 15 menit. Disentrifugasi 12.000 rpm selama 1 menit. Pelet dicuci dengan washing buffer L2 sebanyak dua kali. Pelet selanjutnya dicuci dengan etanol 70% dan aseton. Pelet dikeringkan pada suhu 56°C selama 15 menit kemudian ditambahkan aquadest. Diinkubasi suhu 56°C selama 10 menit. Disentrifugasi 12.000 rpm selama 5 menit. Supernatan digunakan sebagai templat PCR.
Proses PCR dilakukan dalam alat thermalcycler. Tahap amplifikasi dilakukan menggunakan forward primer (mabA-inhA-promoter-FS) dengan urutan 5’ ACATACC TGCTGCGCAAT 3’ dan reverse primer (mabA-inhA-promoter-R) dengan urutan 5’ CTCCGGTAACCAGGACTGAA 3’. Kondisi PCR yang digunakan meliputi predenaturasi pada suhu 95°C selama 15 menit, amplifikasi sebanyak 45 siklus dan setiap siklus meliputi tahap denaturasi (94°C, 1 menit), annealing
(54°C, 1 menit 20 detik), dan tahap ekstensi (72°C, 1 menit 10 detik), serta diakhiri
Tahap elektroforesis menggunakan gel agarosa 1,5% dalam TBE 1x dan selanjutnya divisualisasi menggunakan alat UV transiluminator (Gel Doc XR System). Sekuensing dilakukan secara satu arah menggunakan forward primer.
Analisis homologi dilakukan dengan membandingkan sekuen isolat 134 MDR-TB dengan sekuen nukleotida M. tuberculosis H37Rv (GenBank U66801.1) menggunakan program online BLASTn. Tahap analisis mutasi dilakukan dengan mengidentifikasi terjadinya perubahan basa pada sekuen menggunakan software MEGA4.
Isolasi DNA menggunakan metode Boom yang telah dimodifikasi berhasil dilakukan, sehingga diperoleh supernatan yang mengandung DNA kromosomal. Selanjutnya dilakukan tahap amplifikasi regio promoter inhA dalam alat thermalcycler menggunakan forward primer dengan urutan 5’ ACATACCTGCTGC GCAAT 3’ dan reverse primer dengan urutan 5’ CTCCGGTAACCAGGAC 3’ [5]. Proses
pemisahan fragmen target dilakukan pada gel agarosa 1,5% dengan penambahan EtBr, kemudian divisualisasi menggunakan UV transiluminator Gel Doc XR System. Dalam proses elektroforesis, TBE maupun tris-asetat EDTA (TAE) merupakan buffer standar yang biasa digunakan [6]. Namun dalam penilitian ini dipilih TBE jarena TBE memiliki kapasitas buffer lebih besar dibandingkan TAE, sehingga dapat memberikan resolusi pemisahan yang lebih baik. Selain itu fragmen DNA yang berukuran kecil (<300
dengan ekstensi akhir pada suhu 72°C selama 10 menit.
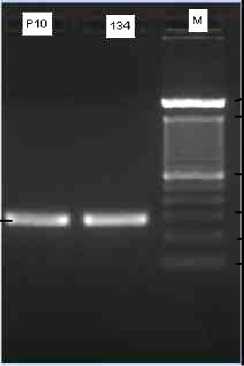
bp) akan bermigrasi lebih cepat dalam TBE dibandingkan TAE [7]. Elektroforegram hasil amplifikasi fragmen target menghasilkan pita amplifikasi tunggal berukuran ±284 bp (Gambar 1). Pita amplifikasi yang tampak jelas pada elektroforegram menandakan bahwa primer dapat mengamplifikasi fragmen target dengan baik.
±284 bp
600 bp
2072 bp
1500 bp
300 bp
200 bp
100 bp
Gambar 1. Elektroforegram hasil amplifikasi M. tuberculosis pada suhu annealing 54°C. M: Marker 100 bp DNA ladder, 134: Isolat 134 MDR-TB
Hasil amplifikasi PCR yang diperoleh hanya mampu memberikan informasi mengenai ukuran fragmen target, tetapi belum mampu memberikan informasi mengenai perbedaan dan persamaan pada tingkat nukleotida. Oleh sebab itu diperlukan teknik sekuensing.
Adanya mutasi akan menyebabkan terjadinya perubahan sekuen basa. Untuk mengetahui terjadinya perubahan tersebut, maka diperlukan teknik sekuensing [8]. Produk PCR isolat 134 MDR-TB disekuensing secara satu arah menggunakan forward primer Produk PCR isolat 134
MDR-TB disekuensing secara satu arah menggunakan forward primer (mabA-inhA-promoter-FS). Tahap sekuensing diproses oleh PT Genetika Science Indonesia. Hasil
Analisis homologi sekuen isolat dilakukan menggunakan BLASTn yang dapat diakses secara online melalui situs URL:// www.ncbi.nlm.nih.gov. Program BLASTn merupakan program yang digunakan untuk membandingkan sekuen nukleotida terhadap database dan menghitung signifikansi statistik sekuen tersebut. Hasil analisis BLASTn akan memberikan informasi mengenai spesies yang memiliki kesamaan dengan sekuen DNA sampel sehingga dapat digunakan untuk identifikasi. Data hasil analisis homologi antara isolat 134 MDR-TB dan sekuen M.tuberculosis H37Rv (U66801.1) memiliki tingkat kesamaan (ident) sebesar 99%. Nilai ident yang tinggi mengindikasikan bahwa isolat 134 MDR-TB dan M.tuberculosis H37Rv (GenBank U66801.1) dianggap sebagai spesies yang sama.
Tahap analisis produk PCR dilanjutkan dengan analisis mutasi. Analisis ini dilakukan dengan cara mengidentifikasi adanya perubahan sekuen basa yang terjadi. Tahap analisis mutasi menggunakan software MEGA4 pada isolat 134 MDR-TB menemukan terjadinya 1 mutasi yang diketahui merupakan substitusi transisi. Mutasi tersebut terjadi pada posisi -15 yaitu perubahan basa sitosin menjadi timin (C÷T). Mutasi pada posisi -15 regio promoter inhA merupakan mutasi yang paling sering ditemukan, dengan angka kejadian bervariasi tergantung wilayah geografis dilakukannya penelitian. Mutasi pada regio promoter inhA diketahui dapat menyebabkan meningkatnya ekspresi protein InhA yang berakibat pada tetap berlangsungnya biosintesis asam mikolat yang merupakan penyusun dinding sel M tuberculosis. Pembentukan dinding sel yang semakin tebal menyebabkan terjadinya peningkatan nilai MIC INH di atas normal [9,10].
sekuensing isolat 134 MDR-TB terbaca sebanyak 256 basa. Sekuen DNA selanjutnya dianalisis homologinya terhadap sekuen M.tuberculosis H37Rv (U66801.1).
Adanya mutasi pada regio promoter inhA biasanya juga diasosiasikan dengan resistensi INH tingkat rendah. Namun, isolat yang resisten terhadap INH (INHr) dan memiliki mutasi pada regio promoter inhA juga dapat memiliki mutasi tambahan pada gen katG yang berakibat pada terjadinya resistensi INH tingkat tinggi [11]. Berdasarkan penelitian sebelumnya pada isolat 134 MDR-TB, ditemukan 2 mutasi yaitu kodon 191 yang mengkode triptofan menjadi arginin (TGG÷CGG) dan kodon 234 yang mengkode alanin menjadi glisin (GCG ÷ GGG) [12]. Terjadinya mutasi pada regio promoter inhA dan gen katG menyebabkan isolat 134 MDR-TB tergolong ke dalam isolat resistensi INH tingkat tinggi.
Tahap amplifikasi regio promoter inhA isolat 134 MDR-TB telah berhasil dilakukan menggunakan sepasang primer, yaitu forward primer (mabA-inhA-promoter-FS) dan reverse primer (mabA-inhA-promoter-R). Jenis mutasi yang terjadi pada regio promoter inhA isolat 134 MDR-TB adalah substitusi transisi dengan perubahan basa sitosin menjadi timin (C÷T) pada posisi -15.
Terima kasih kami ucapkan kepada Kepala Laboratorium Mikrobiologi RS Sanglah yang telah menyediakan isolat MDR-TB dan kepada seluruh staf Laboratorium Biomolekular Fakultas Kedokteran
Universitas Udayana yang telah banyak membantu dalam berlangsungnya penelitian ini.
-
[1] Hazbon, M. H., Brimacombe M., del Vall
M. B., Cavatore M., Guerrero M. I., Basil
M. V., Jacobe H. B., Lavender C, Fyfe ., J., Garcia L. G., Leon C. I., Bose M., dan Alland D. Population Genetics Study of Isoniazid Resistance Mutations and Evolution of Multidrug-Resistant Mycobacterium tuberculosis.
Antimicrobial Agents and Chemotherapy, 2006, 50(8), 2640-2649.
-
[2] World Health Organization. Global Tuberculosis Report 2012. World Health Organization, 2012.
-
[3] Agung, A. A. G., Sawitri A. A. S., dan Wirawan D. N. Rendahnya Proporsi Kontak yang Melakukan Deteksi Dini Tuberkulosis Paru di Puskesmas I Denpasar Selatan Tahun 2012. Public Health and Preventive Medicine
Archive. 2013, 1(1), 55-62.
-
[4] Khadka, D. K., Eampokalap B.,
Panitchakorn J., Ramasoota P, dan Khusmith S. Multiple Mutations in katG and inhA Identified in Thai IsoniazidResistant Mycobacterium tuberculosis Isolates. Southeast Asian Journal Tropical Medicine Public Health, 2007, 38(2), 376-382.
-
[5] Chen, X., Kong F., Wang Q., Li C., Zhang J, dan Gilbert G. L. 2011. Rapid Detection of Isoniazid, Rifampin, and Ofloxacin Resistance in Mycobacterium tuberculosis Clinical Isolates Using High-Resolution Melting Analysis. Journal of Clinical Microbiology, 2011, 49, 34503457.
-
[6] Trigiano, R.N., Ownley B.H., Trigiano A. N., Coley J., Gwinn K. D., dan Moulton J. K. Two Simple and Inexpensive Laboratory Exercises for Teaching Agarose Gel Electrophoresis and DNA Fingerprinting. HorlTechnology, 2008, 18(1), 177-188.
Chaves. F, Murray F., Eisenach K. D., Osornio J. S., Cave M. D., de Leon A. P, [7] Miura, Y., Wake H, dan Kato T. TBE, or Not TBE; That Is The Question:
Beneficial Usage of Tris-Borate For
Obtaining A Higher Resolution of Small DNA Fragments By Agarose Gel
Electrophoresis. Nagoya Medical Journal, 1999, 43(1), 1-6.
-
[8] Purnami, S., Syaifudin M, dan Giyatmi. Penandaan DNA dengan 32P Untuk Deteksi Resistensi Mycobacterium tuberculosis Terhadap Isoniazid. Jurnal Forum Nuklir, 2009, 3(1), 11-20.
-
[9] Ramaswamy, S dan Musser J. M. Molecular Genetic Basis of
Antimicrobial Agent Resistance in Mycobacterium tuberculosis: 1998
Update. Tubercle and Lung Disease 1998, 79(1), 3-29.
-
[10] Cole, S. T., Eisenach K. D., McMurray D. N, dan Jacobs W. R. Tuberculosis and the Tubercle Bacillus. ASM Press, 2005
-
[11] Tekwu, E. M., Sidze L. K., Assam J. P., Tedom J. C., Tchatchoung S., Makafe G. G., Watewale A. L. T., Kuaban C., Eyangoh S., Ntoumi F., Beng V. N. P., and Frank M. Sequence Analysis for Detection of Drug Resistance in Mycobacterium tuberculosis Complex Isolates from the Central Region of Cameroon. BioMed Central
Microbiology, 2014, 14(113), 1-10.
-
[12] Dwiputri, A. W. Identifikasi Mutasi Gen rpoB Pada Isolat P16 dan 134 Mycobacterium tuberculosis Multidrug Resistant (MDR) di Bali Dengan Teknik Polymerase Chain Reaction (PCR.) Skripsi, Universitas Udayana, 2013.
41
Discussion and feedback