DESAIN PRIMER SECARA IN SILICO UNTUK AMPLIFIKASI GEN COA PADA STAPHYLOCOCCUS AUREUS DENGAN POLYMERASE CHAIN REACTION
on
Cakra Kimia (Indonesian E-Journal of Applied Chemistry)
Volume 11, Nomor 1, Mei 2023
DESAIN PRIMER SECARA IN SILICO UNTUK AMPLIFIKASI GEN COA PADA STAPHYLOCOCCUS AUREUS DENGAN POLYMERASE CHAIN REACTION
*
Rhadevka Agnur Saputra dan Didik Wahyudi
Program Studi Sarjana Terapan Teknologi Laboratorium Medis, Sekolah Tinggi Ilmu Kesehatan Nasional, Jl. Solo Baki Kwarasan, Sukoharjo, Jawa tengah, Indonesia
*e-mail korespodensi : didik.wahyudi@stikesnas.ac.id
ABSTRAK: Staphylococcus aureus merupakan bakteri penyebab berbagai infeksi kulit dan jaringan lunak serta penyebab penyakit yang lebih serius dan lebih invasif. Gen coa adalah gen penyandi enzim koagulase yang dimiliki Staphylococcus aureus. Gen coa dapat dideteksi dengan teknik Polymerase Chain Reaction (PCR). Metode PCR memerlukan sepasang primer. Primer dapat didesain secara in silico. Penelitian ini bertujuan untuk mengetahui apakah primer yang digunakan dapat memgamplifikasi gen coa pada Staphylococcus aureus. Primer didesain menggunakan software NCBI kemudian dilakukan uji kualitas primer menggunakan software oligoevaluator dan NetPrimers. Didapatkan 2 kandidat primer yaitu primer pertama forward 5’ CAAAGCAGATGCGATAGTGG 3’ reverse 5’ GTTCGCTTTATTTTCCGGCT 3’ dan primer kedua dengan forward 5’ AGCCGGAAAATAAAGCGAAC 3’ reverse 5’ ACCTTGAATTGTGCCTTGTG 3’. Hasil primer yang didesain diujikan dengan metode PCR. Hasil visualisasi kedua primer terbentuk pita DNA yang tebal dan jelas, tidak ada smear, namun terdapat primer dimer dan letak produk tidak sesuai dengan hasil analisis secara in silico. Primer yang didesain dapat digunakan untuk amplifikasi gen coa pada Staphylococcus aureus. Terdapat perbedaan spesifisitas antar primer yang didesain dimana primer 2 lebih baik dari primer 1.
Kata Kunci : Desain primer; Gen coa; Staphylococcus aureus; PCR
ABSTRACT: Staphylococcus aureus is bacteria that causes of a variety of skin and soft tissue infections and causes more serious and invasive disease. The coa gene is a coagulase enzyme gene that belongs to Staphylococcus aureus. The coa gene can be detected by Polymerase Chain Reaction (PCR) technique. The PCR method requires primers. Primer can be designed in silico. This study aims to determine whether the primer used can amplify the coa gene in Staphylococcus aureus. Primers were designed using NCBI software and then tested for primary quality using oligoevaluator and NetPrimers software. Two primary candidates were obtained, namely the first primer forward 5' CAAAGCAGATGCGATAGTGG 3' reverse 5' GTTCGCTTTATTTTCCGGCT 3' and the second primer forward 5' AGCCGGAAAATAAAGCGAAC 3' reverse 5' ACCTTGAATTGTGCCTTGTGTG 3'. The designed primary results were tested by the PCR method. The results of the visualization of the two primers formed thick and clear DNA bands, no smears, but there were dimer primers and the location of the products did not match the results of in silico analysis. The designed primer can be used for coa gene amplification in Staphylococcus aureus. There are differences in specifications between the designed primers where primer 2 is better than primary 1.
Keywords : Primer design; Gene coa; Staphylococcus aureus; PCR
Staphylococcus aureus merupakan bakteri patogen yang sering dijumpai dan merupakan penyebab berbagai infeksi kulit dan jaringan lunak serta penyebab penyakit yang lebih serius dan lebih invasive [1]. Staphylococcus aureus dapat menyebabkan infeksi karena mampu berkembang biak dan menyebar luas dalam jaringan tubuh karena mampu menghasilkan enzim koagulase [2].
Koagulase merupakan protein yang bekerja terhadap fibrinogen inang. Fungsi utama koagulase yaitu untuk mengubah fibrinogen menjadi benang benang fibrin dengan mengaktivasi protrombin secara non-proteolitik yang akan mengalihkan aktivasi protrombin oleh regulasi tubuh inang [3].
Gen penyandi enzim koagulase (coa) dapat digunakan sebagai penanda adanya bakteri Staphylococcus aureus. Gen coa adalah gen penyandi enzim koagulase, diantara genus Staphylococcus hanya Staphylococcus aureus saja yang memproduksi koagulase sehingga dapat membedakan Staphylococcus aureus dengan Staphylococcus lainnya [2].
Gen coa dapat dideteksi dengan teknik Polymerase Chain Reaction (PCR), berdasarkan penelitian yang dilakukan oleh Wahyuni dkk. [2] telah berhasil mendeteksi adanya gen coa pada Staphylococcus aureus pada susu sapi murni dengan PCR. Keberhasilan amplifikasi dengan teknik PCR sangat tergantung dengan primer yang digunakan. Primer adalah urutan nukleotida dengan panjang 18-30 bp yang diperlukan pada proses isolasi DNA [4]. Untuk mendapatkan primer dapat dilakukan dengan cara melakukan desain primer. Pembuatan desain primer dilakukan melalui secara in silico. In silico merupakan studi dengan menggunakan bantuan komputer dan software [5].
Penelitian ini bertujuan untuk mengetahui mengetahui spesifisitas primer yang didesain untuk amplifikasi gen coa pada Staphylococcus aureus dengan PCR.
Bahan yang digunakan pada penelitian ini adalah media BHI, media NA miring, media MSA, plasma darah 5%, plasma sitrat, agar base, tissue, larutan cat gram A, B, C, D, emersi oil, isolate bakteri Staphylococcus aureus, lysozyme, Gram + buffer, Proteinase K, GB buffer, Elution buffer, Etanol Absolute, W1 Buffer, aquabidest, aquadest, pelarut TBE 1x agarose, loading dye, gel red, primer forward, primer reverse, master mix, dd.H2O, R-Nase Free Water.
Alat yang digunakan dalam penelitian ini diantaranya Erlenmeyer, mikroskop, autoclave, cawan petri, tabung reaksi, ohse bulat, inkubator, ohse lurus, objek glass, collection tube, kolom GD, centrifuge, 1,5 ml microcentrifuge tube, white tip, yellow tip, blue tip, micropipet, vortex, rak tabung, waterbath, kulkas, spektrofotometer UV – VIS, agar case, magnetic stirrer, chamber elektroforesis, elektroforesis gel agarose, Polymerase Chain Reaction (PCR) Thermal Cyler Machine T100, Biorad UV – Transilluminator Gel Doc.
Tahapan prosedur penelitian ini yaitu pembuatan media, identifikasi Staphylococcus aureus, pembuatan standart kekeruhan MC Farland 0,5, pembuatan suspensi Staphylococcus aureus, isolasi DNA menggunakan Presto™ Mini gDNA Bacteria Kit (Geneaid Biotech, Taiwan), uji kualitatif DNA, uji kuantitatif DNA, pembuatan desain primer, optimasi suhu annealing PCR, dan PCR.
Hasil karakterisasi kultur murni Staphylococcus aureus yaitu pengecatan gram, uji hemolisa, uji katalase, uji pigmentasi, uji fermentasi mannitol, uji koagulase. Bakteri Staphylococcus aureus dilakukan pengecatan gram dan diamati secara mikroskopis ditemukan bakteri
berbentuk bulat, berwarna ungu dengan susunan bergerombol. Uji hemolisa pada media BAP menunjukkan adanya zona hemolisis disekitar koloni. Uji katalase menunjukkan hasil positif yang ditandai dengan terbentuknya gelembung gas. Uji pigmentasi pada media NA miring menunjukkan warna koloni kuning emas. Uji koagulase menunjukkan hasil positif yang ditandai dengan terbentuknya aglutinasi. Berdasarkan hasil karakterisasi Staphylococcus aureus dapat disimpulkan bahwa sampel merupakan benar kultur murni Staphylococcus aureus.
Isolasi DNA Staphylococcus aureus dilakukan menggunakan PrestoTM Mini gDNA Bacteria Kit dengan berbagai tahapan yaitu persiapan sampel, lisis DNA, pengikatan DNA, Pencucian DNA, dan elusi DNA. Hasil DNA yang terekstraksi dapat dilihat melalui uji kualitatif DNA dengan elektroforesis gel agarosa.
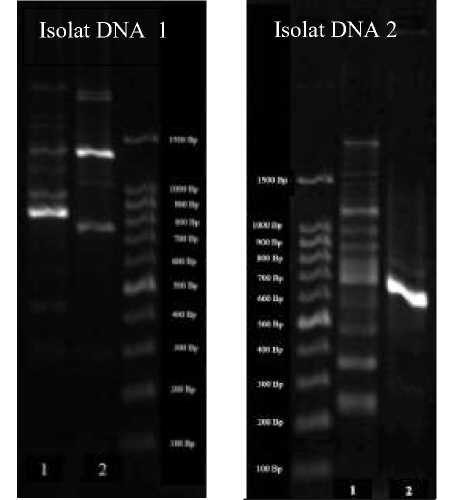
Uji kualitatif DNA dilakukan dengan alat elektroforesis gel agarosa. Bahan yang digunakan pada tahap uji kualitatif DNA meliputi gel red, loading dye, dan isolat DNA. Untuk melihat keberhasilan ekstraksi DNA diamati dengan alat Gel Doc. DNA yang berhasil terekstraksi ditandai dengan terbentuknya pita DNA. Hasil uji kualitatif DNA dapat dilihat pada Gambar 1 dimana DNA dari sampel 1 dan sampel 2 berhasil terekstraksi yang ditandai dengan terbentuknya pita DNA. Hasil uji kualitatif DNA yang baik ditunjukkan dengan terbentuknya pita DNA tidak ada smear [6].
Uji kuantitatif DNA dilakukan dengan menggunakan alat Spektrofotometer UV VIS. Bahan yang digunakan pada tahap uji kuantitatif DNA adalah isolat DNA dan buffer TE 1X. Isolat DNA yang digunakan dilakukan pengenceran sebanyak 150X. Kemudian dilakukan pembacaan pada Spektrofotometer UV VIS dengan absorbansi ƛ260 dan ƛ280. Uji Kuantitatif dilakukan untuk mengetahui kemurnian DNA dan konsentrasi DNA. DNA yang berkualitas baik harus memiliki konsentrai diatas 100 ng/µL dan nilai kemurnian 1,82,0 sehingga dapat disimpulkan bahwa
kedua isolat DNA memiliki konsentrasi yang baik tetapi memiliki nilai kemurnian yang rendah [7]. Hasil uji kuantitatif DNA dapat dilihat pada Tabel 1. Kedua isolat DNA memiliki konsentrasi yang tinggi namun memiliki kemurnian yang rendah karena hanya mempunyai kemurnian berturut-turut sebesar 1,2 dan 1,3 untuk sampel 1 dan sampel 2 yang mengindikasikan adanya kontaminasi oleh protein dan polisakarida [7].
Pita DNA
Sampel 1 Sampel 2
Gambar 1. Hasil uji kualitatif DNA sampel 1 dan 2.
Tabel 1. Hasil uji kuantitatif DNA
|
Sampel |
Abs λ260 |
Abs λ280 |
Kemurnian DNA |
Konsentrasi DNA (ng/µL) |
|
1 |
0.219 |
0.182 |
1,2 |
1642.5 |
|
2 |
0.232 |
0.176 |
1.3 |
1740 |
Pada penelitian ini primer yang didesain merupakan primer dari gen coa pada Staphylococcus aureus yang dapat diakses dari GeneBank dengan ID 66838528. Desain primer dibuat dengan software NCBI, selanjutnya pasangan primer diuji kualitasnya dengan software oligoevaluator, dan NetPrimers. Analisis kandidat primer dilakukan untuk mendapatkan 2 pasang primer terbaik yang memenuhi kriteria. Analisis kandidat primer dilakukan dengan memperhatikan kriteria primer yang baik antara lain primer yang baik berkisar antara 18-30 pasang basa. Primer yang terlalu pendek akan
Tabel 2. Kandidat Primer yang Didesain dengan Software NCBI
|
No |
Primer |
Sequence (5’-3’) |
Length |
Tm |
GC% |
Produk Size |
|
1. |
Forward |
CAAAGCAGATGCGATAGTGG |
20 |
56,97 |
50.00 |
781 |
|
Reverse |
GTTCGCTTTATTTTCCGGCT |
20 |
57,09 |
45.00 | ||
|
2. |
Forward |
ATGCGAGAGAATTAAGGGCA |
20 |
56,99 |
45.00 |
880 |
|
Reverse |
GCGTTTGTCTCACTTGGTTT |
20 |
57,16 |
45.00 | ||
|
3. |
Forward |
GCCGGAAAATAAAGCGAACT |
20 |
57,09 |
45.00 |
618 |
|
Reverse |
TATTCACGGATACCAGTGCC |
20 |
57.10 |
50.00 | ||
|
4. |
Forward |
AGCCGGAAAATAAAGCGAAC |
20 |
57,09 |
45.00 |
257 |
|
Reverse |
ACCTTGAATTGTGCCTTGTG |
20 |
56,82 |
45.00 | ||
|
5. |
Forward |
ATGCGATAGTGGGTAAGGAC |
20 |
56,80 |
50.00 |
774 |
|
Reverse |
AGTTCGCTTTATTTTCCGGC |
20 |
57,09 |
45.00 | ||
|
6. |
Forward |
AAACCAAGTGAGACAAACGC |
20 |
57,16 |
45.00 |
462 |
|
Reverse |
AGTTGCAGTACCATCTGCAT |
20 |
57,20 |
45.00 | ||
|
7. |
Forward |
TGCGAGAGAATTAAGGGCAA |
20 |
57,22 |
45.00 |
452 |
|
Reverse |
CACCTTGAATTGTGCCTTGT |
20 |
56,82 |
45.00 | ||
|
8. |
Forward |
GGGATAACAAAGCAGATGCG |
20 |
57,24 |
50.00 |
984 |
|
Reverse |
CAACTGGAAGTGGTGCTTTT |
20 |
56,82 |
45.00 | ||
|
9. |
Forward |
ACAAGGCACAATTCAAGGTG |
20 |
57.34 |
45.00 |
102 |
|
Reverse |
GTTGGGAAGTCTGGTCCTTT |
20 |
56.60 |
50.00 | ||
|
10. |
Forward |
CACAAGGCACAATTCAAGGT |
20 |
56,82 |
45.00 |
106 |
|
Reverse |
ATTGTTGGGAAGTCTGGTCC |
20 |
57,34 |
50.00 |
Tabel 3. Uji Kualitas Primer dengan Software Oligoevaluator
|
No |
Primer |
Sequence (5’-3’) |
Run |
Secondary Structure |
Primer Dimer |
|
1. |
Forward |
CAAAGCAGATGCGATAGTGG |
3 |
None |
No |
|
Reverse |
GTTCGCTTTATTTTCCGGCT |
4 |
None |
No | |
|
2. |
Forward |
ATGCGAGAGAATTAAGGGCA |
3 |
Weak |
No |
|
Reverse |
GCGTTTGTCTCACTTGGTTT |
3 |
None |
No | |
|
3. |
Forward |
GCCGGAAAATAAAGCGAACT |
4 |
None |
No |
|
Reverse |
TATTCACGGATACCAGTGCC |
2 |
Weak |
No | |
|
4. |
Forward |
AGCCGGAAAATAAAGCGAAC |
4 |
None |
No |
|
Reverse |
ACCTTGAATTGTGCCTTGTG |
2 |
None |
No | |
|
5. |
Forward |
ATGCGATAGTGGGTAAGGAC |
3 |
None |
No |
|
Reverse |
AGTTCGCTTTATTTTCCGGC |
4 |
None |
No | |
|
6. |
Forward |
AAACCAAGTGAGACAAACGC |
3 |
None |
No |
|
Reverse |
AGTTGCAGTACCATCTGCAT |
2 |
Strong |
No | |
|
7. |
Forward |
TGCGAGAGAATTAAGGGCAA |
3 |
Weak |
No |
|
Reverse |
CACCTTGAATTGTGCCTTGT |
2 |
Weak |
No | |
|
8. |
Forward |
GGGATAACAAAGCAGATGCG |
3 |
None |
No |
|
Reverse |
CAACTGGAAGTGGTGCTTTT |
4 |
Very Weak |
No | |
|
9. |
Forward |
ACAAGGCACAATTCAAGGTG |
2 |
Weak |
No |
|
Reverse |
GTTGGGAAGTCTGGTCCTTT |
3 |
Weak |
No | |
|
10. |
Forward |
CACAAGGCACAATTCAAGGT |
2 |
None |
No |
|
Reverse |
ATTGTTGGGAAGTCTGGTCC |
3 |
Weak |
No |
cenderung mengalami mispriming (kesalahan penempelan). Primer yang memiliki panjang lebih dari 30 pasang basa akan menyebabkan terjadinya hibridasi sehingga akan menghambat proses polimerisasi DNA [8].
Primer yang baik mempunyai persentase G dan C sektar 40-60%. Tingginya kandungan GC akan mempersulit pemutusan rantai utas ganda pada primer. Sedangkan kandungan GC yang rendah pada primer menyebabkan primer tidak
mampu menempel dan akan berdampak pada penurunan efisiensi PCR [9].
Temperature melting (Tm) yang baik adalah 50 – 650C. Apabila kurang akan menyebabkan primer menempel ditempat lain sehingga menghasilkan produk yang tidak spesifik. Apabila lebih akan menyebabkan proses amplifikasi tidak berjalan dengan baik [5].
Repeat merupakan pengulangan dua basa yang sama, misal (GAGAGA) yang terdapat pada primer. Repeat pada urutan primer dapat menyebabkan mispriming pada proses PCR, jumlah repeat yang ditoleransi maksimal berjumlah empat [10].
Hasil desain primer dengan software NCBI dapat dilihat pada Tabel 2 yang menunjukkan bahwa didapatkan 10 pasang kandidat primer. Kandidat primer hasil desain oleh software NCBI kemudian dianalisis. Kandidat primer yang memenuhi kriteria dianalisa lanjut dengan menguji kualitas primer dengan software oligoevaluator yang hasilnya dapat dilihat pada Tabel 3.
Berdasarkan hasil uji kualitas primer dengan software oligoevaluator tersebut kemudian dilakukan analisa berdasarkan kriteria primer yang baik dimana pasangan primer yang memenuhi kriteria dilanjutkan analisa dengan software Netprimers yang dapat dilihat pada Tabel 4 dan didapatkan 2 pasangan primer terbaik yang memenuhi kriteria untuk amplifikasi gen coa. Hasil Blast NCBI dari kedua pasangan primer tersebut dapat dilihat pada Gambar 2.
Optimasi suhu annealing dilakukan untuk mendapatkan suhu optimum yang digunakan untuk PCR. Suhu annealing didapatkan dari perhitungan rata – rata primer forward dan reverse kemudian hasilnya dikurangi 5oC, hal ini dikarenakan suhu annealing biasanya dibawah 5oC dari Tm sesungguhnya [11].
Hasil optimasi suhu annealing primer 1 dan 2 dapat dilihat pada Gambar 3. yaitu pada keseluruhan suhu dapat mengamplifikasikan gen coa, namun terbentuk primer dimer dan letak pita DNA
tidak sesuai dengan analisis secara in silico Hal ini dikarenakan perancangan primer tidak spesifik. Primer yang tidak spesifik dapat menyebabkan teramplifikasinya daerah lain dalam genom yang tidak dijadikan sasaran [12]. Suhu optimum yang dapat digunakan untuk amplifikasi gen coa pada primer 1 adalah suhu 46oC, sedangkan suhu optimum pada primer 2 adalah suhu 49oC dimana pada suhu tersebut terbentuk pita DNA yang tebal dan jelas meskipun terdapat primer dimer.
Amplifikasi DNA dilakukan dengan menggunakan suhu annealing optimum yang sudah diketahui melalui optimasi suhu annealing. Instrument PCR Thermal Cycler T100 disetting dengan suhu pra denaturasi 95 oC selama 3 menit, suhu denaturasi 95 oC selama 30 detik, suhu annealing sesuai suhu optimum yaitu untuk primer 1 adalah 46 oC dan untuk primer 2 adalah 49 oC, suhu ekstensi 72 oC selama 1 menit dan final ekstensi 72 oC selama 5 menit. Hasil amplifikasi isolat DNA 1 dan 2 dapat dilihat pada Gambar 4. yaitu kedua primer dapat mengampilifikasikan gen coa pada Staphylococcus aureus yang ditandai dengan terbentuknya pita DNA yang tebal dan jelas, tidak ada smear, namun terdapat primer dimer dan letak produk tidak sesuai dengan hasil analisis secara in silico.
Hasil amplifikasi yang menunjukkan adanya dimer serta letak produk tidak sesuai dengan analisis secara in silico ini disebabkan karena primer tidak mengidentifikasi spesies secara spesifik. Dari hasil Primer Blast terhadap 2 pasang primer yang terpilih seperti terlihat pada Gambar 2 didapatkan bahwa primer mengamplifikasi gen coa pada Staphylococcus aureus, selain itu primer juga dapat mengamplifikasi pada Staphylococcus argenteus sehingga kedua primer yang didesain merupakan primer yang tidak spesifik. Hal ini selaras dengan penelitian yang dilakukan oleh Sihotang dkk [13] dimana primer yang didesain ketika dianalisis dengan Primer Blast menunjukkan bahwa primer dapat mengamplifikasi 2 spesies yang berbeda
Tabel 4. Uji Kualitas Primer dengan Software Netprimers
|
No |
Primer |
Sequence (5’-3’) |
Repeat |
Run |
Rating |
|
1. |
Forward |
CAAAGCAGATGCGATAGTGG |
- |
3 |
82 |
|
Reverse |
GTTCGCTTTATTTTCCGGCT |
- |
4 |
90 | |
|
4. |
Forward |
AGCCGGAAAATAAAGCGAAC |
- |
4 |
100 |
|
Reverse |
ACCTTGAATTGTGCCTTGTG |
- |
- |
80 | |
|
5. |
Forward |
ATGCGATAGTGGGTAAGGAC |
- |
3 |
100 |
|
Reverse |
AGTTCGCTTTATTTTCCGGC |
- |
4 |
80 |
Primer pair 1
|
Sequence (5*-*3,) |
Length |
Tm |
GC% |
Self complementarity |
Self 3 complementarity | |
|
Forward primer |
Caaagcagatgcgatagtgg |
20 |
56.97 |
50.00 |
4.OQ |
0.00 |
|
Reverse primer |
Gl ICGCI IIATIT TCCGGC1 |
20 |
57.09 |
45 OO |
4 OU |
OOU |
Products on target templates
>FRH21777.2 StophyloCGCCus argeπteυs MSHR1132 complete genome sequence
product length - 781 forward primer 1 CAAAGCAGATGCGATAGTGG 20
Template 236Θ79 .................... 236098
Reverse primer 1 GTTCGCTTTATΓTTCCGGCT 20
Template 236859 .................... 236840
>aha3<√>K8 i Staphytpcoccus aureus coo genes Tor hypothetical proteins, Staphyiocoogulase. .IcetyHioA acethyhransfcrasc Homntogue. complete cds1 strain JCsCl469
product length - 781
Forward primer 1 CAAAfeCAGAKKGATAGTGG 20
Template 2476 .................... 2495
Reverse primer 1 GTTCGCTTTATTrTCCGGCT 20
Template 3256 .................... 3237
Gambar 2. Hasil Blast NCBI pasangan primer
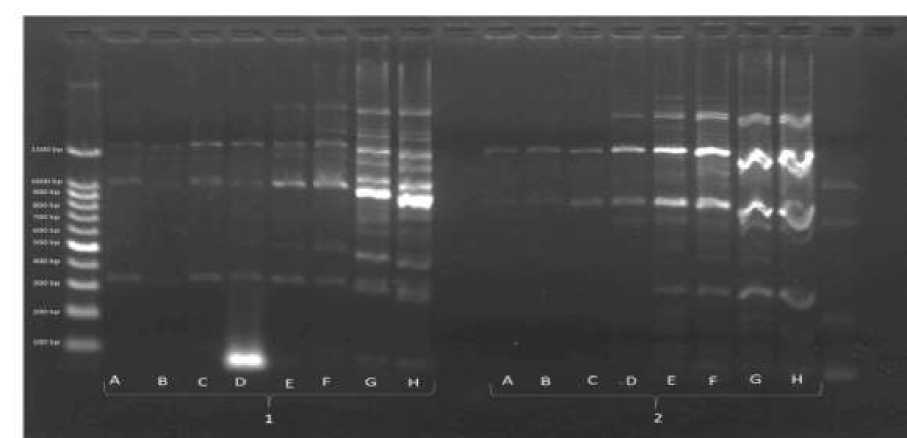
Gambar 3. Elektroforesis hasil optimasi suhu annealing primer 1 suhu 53ºC (A), 52,4ºC (B), 51,6ºC (C), 50,4ºC (D), 48,7ºC (E), 47,3ºC (F), 46,6ºC (G), 46,0ºC (H) dan primer 2 suhu 53ºC (A), 52,4ºC (B), 51,6ºC (C), 50,4ºC (D), 48,7ºC (E), 47,3ºC (F), 46,6ºC (G), 46,0ºC (H)
sehingga rancangan primer menghasilkan pita yang berbeda dari analisis secara In Silico. Rancangan primer yang kurang baik menyebabkan reaksi PCR tidak bekerja dengan baik sehingga menyebabkan produk PCR yang tidak spesifik dan atau terbentuknya primer dimer [14]. .
Terdapat perbedaan spesifisitas diantara kedua primer dimana primer 2 memiliki spesifisitas yang lebih baik daripada primer 1 disebabkan karena pada hasil PCR primer 2 menghasilkan pita DNA yang tebal dengan dimer yang lebih sedikit daripada primer 1. Hal ini sesuai dengan analisis secara in silico pada
software Netprimers bahwa primer 2 memiliki rating yang lebih besar dari pada primer 1.
Gambar 4. Amplifikasi DNA dengan PCR
Berdasarkan hasil penelitian terhadap desain primer secara in silico untuk amplifikasi gen coa pada Staphylococcus aureus dengan PCR yang telah dilakukan maka dapat disimpulkan bahwa kedua pasang primer yang didesain dengan NCBI, oligoevaluator, dan Netprimers dapat digunakan untuk amplifikasi gen coa pada Staphylococcus aureus dengan PCR dan terdapat perbedaan spesifitas antar primer yang didesain, dimana primer 2 memiliki spesifisitas yang lebih baik daripada primer 1 dikarenakan hasil amplifikasi menghasilkan pita DNA yang jelas dengan dimer yang lebih sedikit daripada primer 1.
Peneliti mengucapkan terima kasih kepada Sekolah Tinggi Ilmu Kesehatan Nasional yang telah memfasilitasi penelitian ini serta pihak – pihak yang membantu pelaksanaan penelitin ini.
-
[1] Delost, D. M. (2015). Mikrobiologi Diagnostik. Penerbit Buku
Kedokteran EGC.
-
[2] Wahyuni, R. A., Darmawati, S., &
Prastiyanto, M. E. (2017). Deteksi Gen Isolat DNA 1aphylococcus aureus Yang Diisolasi Dari Susu Sapi Murni. Jurnal Prosiding Nasional Publikasi Hasil-Hasil Penelitian Dan Pengabdian Masyarakat.
-
[3] Kobayashi, S. D., Malachowa, N., & Deleo, F. R. (2015). Pathogenesis of Staphylococcus aureus abscesses. In American Journal of Pathology, 185
(6), 1518–1527 https://doi.org/10.1016/j.ajpath.2014. 11.030
-
[4] Syamsidi, A., Aanisah, N., Fiqram, R., & Jultri, I. al. (2021). Primer Design and Analysis for Detection of mecA gene. Journal of Tropical Pharmacy and Chemistry, 5(3), 245–
253.
https://doi.org/10.25026/jtpc.v5i3.297
-
[5] Sasmitha, L. V., Yustiantara, P. S., & Yowani, S. C. (2018). Desain DNA Primer Secara In Silico Sebagai Pendeteksi Mutasi Gen gyrA Mycrobacterium tuberculosis Untuk Metode Polymerase Chain Reaction. Cakra Kimia (Indonesian E-Journal of Applied Chemistry), 6(1), 63–69.
-
[6] Hikmatyar, M., Ida Royani, J. (2015). Bioteknologi & Biosains Indonesia Isolasi Dan Amplifikasi DNA Keladi Tikus (Thyponium flagelliform)
Untuk Identifikasi Keragaman Genetik Isolation and amplification of Keladi tikus (Thyponium flagelliform) DNA for identification of genetic variation. Jurnal Bioteknologi & Biosains Indonesia, 2, 42–48.
-
[7] Farmawati, D. A., Nengah Wirajana, I., & Yowani, S. C. (2015).
Perbandingan Kualitas DNA Dengan
Menggunakan Metode Boom Original Dan Boom Modifikasi Pada Isolat Mycobacterium tuberculosis 151. Jurnal Kimia, 9, 41–46.
-
[8] Anika, M., & Hilda Putri, D. (2019). Primer Design For Identification Of Beta-Carotene Encoding Genes In Cassava. Jurnal Bio Sains, 4(1): 39– 47.
-
[9] Sasmito, D. E. K., Rahadian, K., & Muhimmah, I. (2014). Karakteristik Primer pada Polymerase Chain Reaction (PCR) untuk Sekuensing DNA: Mini Review. Seminar
Nasional Informatika Medis, 5: 94– 102.
-
[10] Praja & Rosalina. (2021). Perancangan Primer Gen IktB pada Fusobacterium necrophorum untuk Analisis PCR. Jurnal Sains dan Teknologi Pertanian, 2(2), 47-55.
-
[11] Yuenleni. (2019). Langkah-Langkah Optimasi PCR. Indonesian Journal Of Laboratory, 1(3), 51-56.
-
[12] Aris, M., Sukenda., Harris E., Sukadi, F.M., & Yuhana. (2013). Identifikasi Molekuler Bakteri Patogen dan Desain Primer PCR. Jurnal
Budidaya Perairan, 1(3), 43-50.
-
[13] Sihotang, D. E. A. M., Erwinda, E.Y., Suwarni, E., Lusianti, E. (2021). Desain Primer dan Analisis in Silico untuk Amplifikasi Gen mt-Co1 pada Tikus got (Rattus norvegicus). Jurnal Eruditio, 1(2), 20-29.
-
[14] Yustinadewi, D. P., Yustiantara, P. S., & Narayani, I. (2018). MDR-1 Gene 1199 Variant Primer Design Techniques In Pediatric Patient Buffy Coat Samples With LLA. Jurnal Metamorfosa of Biological Siences, 5(1), 105–111.
15

Discussion and feedback