PHYLOGENY ANALYSIS OF BALI ISOLATES OF NEWCASTLE DISEASE VIRUS IN 2013 TO 2014 BASED ON CLEAVAGE SITE OF THE FUSION PROTEIN
on
Volume 13 No. 1: 67-74
Pebruari 2021
DOI: 10.24843/bulvet.2021.v13.i01.p11
Buletin Veteriner Udayana
pISSN: 2085-2495; eISSN: 2477-2712
Online pada: http://ojs.unud.ac.id/index.php/buletinvet
Terakreditasi Nasional Peringkat 3, DJPRP Kementerian Ristekdikti No. 21/E/KPT/2018, Tanggal 9 Juli 2018
Analisis Filogeni Virus Newcastle Disease Isolat Bali Tahun 2013 Sampai 2014 Berdasarkan Sekuen Daerah Pemotongan Protein Fusion
(PHYLOGENY ANALYSIS OF BALI ISOLATES OF NEWCASTLE DISEASE VIRUS IN 2013 TO 2014 BASED ON CLEAVAGE SITE OF THE FUSION PROTEIN)
Fedri Rell1*, Anak Agung Ayu Mirah Adi2, I Gusti Ngurah Kade Mahardika3 1Laboratorium Mikrobiologi, Program Studi Kedokteran Hewan Fakultas Kedokteran Universitas Hasanddin, Jl. Perintis Kemerdekaan kampus Tamalanrea Km. 10 Makassar, Indonesia 90245. 2Laboratorium Patologi Veteriner, Fakultas Kedokteran Hewan Universitas
Udayana, Jl. PB. Sudirman Denpasar Bali, Indonesia 80225. 3Laboratorium Virologi
Veteriner, Fakultas Kedokteran Hewan Universitas Udayana, Denpasar, Jl. PB. Sudirman Denpasar, Bali, Indonesia 80225. *Email: fedrirell@unhas.ac.id
Abstrak
Newcastle disease (ND) merupakan penyakit kontagius yang disebabkan virus Avian paramyxovirus serotype 1 yang menginfeksi bangsa unggas. Penelitian ini bertujuan untuk menganalisis pohon filogeni berdasarkan sekuan gen daerah pemotonan protein fusion dari virus ND pada peternakan ayam di provinsi Bali dari tahun 2013 sampai 2014. Sebanyak empat isolat virus dari kasus ayam sakit/mati yang dicurigai terinfeksi oleh virus Newcastle disease. Sekuen potongan gen F disejajarkan dan dianalisis dengan program MEGA5. Analisis sekuen asam amino daerah pemotongan protein F keempat isolat memiliki sekuen 112R-R-Q-K-R-F117 dan dilanjutkan dengan analisis pohom filogeni yang menunjukan bahwa keempat isolat merupakan virus Newcastle disease yang virulen. Penelitian ini menunjukan bahwa keempat isolat lapang Bali tahun 2013 sampai 2014 masuk ke dalam kelompok virus Newcastle disease genotipe VII.
Kata kunci: virus Newcastle disease; genotipe VII; sekuen; filogeni.
Abstract
Newcastle disease is a contagious disease caused by the Avian paramyxovirus serotype 1 virus, which infected the poultry. The aims of this study were conducted to phylogenetic analysis of the fusion protein cleavage site of Newcastle disease virus found in a chicken farm in the province of Bali throughout the year 2013 to 2014. There are four isolates from sick chicken’s cases/death suspected of being infected by the Newcastle disease virus. The sequence of the protein fusion was aligned and edited using MEGA5. The amino acid sequence of the F cleavage site of all isolates was 112R-R-Q-K-R-F117 and analysis of phylogeny tree, which justified that all four are virulent of Newcastle disease virus. It is concluded that four Bali isolates in 2013 to 2014 under this study are Newcastle disease virus of genotype VII.
Keywords: Newcastle disease virus; genotype VII; sequence; phylogeny.
PENDAHULUAN
Newcastle disease (ND) merupakan penyakit pada bangsa unggas yang disebabkan oleh virus yang sangat kontagius. Penyakit ND berdampak secara signifiakan terhadap perekonomian karena menyebabkan penurunan produksi pada peternakan ayam dan kematian yang tinggi (Piacenti et al, 2006; Dortmans, 2011).
Penyakit ini yang dalam bahasa jawa disebut juga dengan penyakit tetelo bersifat endemik di seluruh peternakan Indonesia (Saepulloh dan Darminto, 2005). Vaksinasi merupakan program utama pengendalian penyakit ND. Outbreak penyakit ND tetap mengancam peternakan unggas di Indonesia.
Avian paramyxovirus serotype 1 (APMV-1) merupakan virus penyebab
penyakit ND. Virus ini berasal dari ordo Mononegavirales famili Paramyxoviridae (ICTV, 2013). Virus Avian paramyxovirus serotype 1 memiliki genom RNA berserat tunggal, dengan panjang ± 15.200 nukleotida (nt) dan berpolarisasi negatif. Materi genetik tersebut menyandi protein struktural (Alexander, 1987). Protein F merupakan protein struktural yang menyusun daerah amplop virus.
Avian paramyxovirus serotype 1 menginfeksi sebagian besar bangsa unggas. Berdasarkan derajat tingkat kesakitan dan kematian pada ayam virus ND menjadi lima subtipe yaitu : tipe velogenik viscerotropik, tipe velogenik neurotropik, tipe mesogenik, tipe lentogenik, tipe asimptomatik enterik (Lee et al., 2008). Virulensi virus ND dapat ditentukan berdasarkan analisis secara molekuler terhadap sekuen gen penyandi protein F (Putra dan Adi, 2016).
Virus ND memiliki amplop tersusun atas protein F dan HN yang berperan dalam proses replikasi virus (Rell et al., 2015). Secara khusus protein F adalah protein sebagai penentu penetrasi partikel virus ke dalam sel terinfeksi (Glickman et al., 1988; Morrison, 2003). Protein F memiliki bagian yang disebut daerah pemotongan (cleavage sites). Cleavage sites merupakan daerah yang sangat mempengaruhi proses infeksi virus. Gen penyandi daerah pemotongan tersebut menetukan virulensi virus (Yusoff dan Tan, 2001)
Sekuen gen penyandi daerah pemotongan protein F isolat Bali tahun 2013 sampai 2014 dianalisis untuk mengetahui hubungan kekerabatan virus ND isolat lapang secara molekuler dengan beberapa genotipe virus ND dikaji sebagai dasar dalam pemelihan bibit vaksin terutama dalam pembuatan vaksin isolat lapang.
METODE PENELITIAN
Sampel Penelitian
Sampel berupa ayam sakit atau mati yang diduga berasal dari kasus infeksi virus ND. Keempat virus ND isolat Bali pada
tahun 2013–2014 diberi kode yaitu VND/ayam/lokal/Badung/Bali/2013, VND/ayam/ras/Klungkung/Bali/2014, VND/ayam/ras/Tabanan1/Bali/2014, danVND/ayam/ras/Tabanan2/Bali/2014.
Isolasi dan Propagasi
Keempat sampel virus ND isolat Bali diprogasi pada telur ayam bertunas umur 9 sampai 10 hari melalui ruang alantois. Setelah embrio mati, cairan inokulum diuji dengan uji hemaglutinasi (HA) dan hambatan hemaglutinasi (HI) untuk mengetahui ada tidanya virus.
Isolasi RNA Virus
Cairan alantois embrio diekstrak untuk memperoleh Asam inti ribonucleic acid (RNA) virus ND, dengan metode Trizol. Sebanyak 250 µl caiaran alantois ditambahkan dengan 750 µl Trizol. Setalah divorteks selama beberapa saat kemudian diinkubasi pada suhu kamar selama 5 menit. Campuran kemudian ditambahkan 200 µl kloroform dan divorteks, kemudian didiamkan selama 15 menit. Campuran tersebut disentrifuga selama 15 menit dengan kecepatan 12.000 rpm. aqueus dipindahkan dan ditambahkan isopropil alkohol sebanyak 500 µl. Campuran diinkubasi pada suhu kamar selama 10 menit dan disentrifuse pada 12.000 rcf selama 10 menit dan supernatan dibuang. Pelet dicuci dengan alkohol 70% sebanyak 1000 µl dan disentrifuse dengan kecepatan 7.500 rcf selama 5 menit. Supernatan dibuang, RNA dikeringkan dan disuspensikan dalam akuades yang tidak mengandung dari enzim RNAse (diethyl pyro carbonat treated water).
Amplifikasi
Sepasang primer digunakan untuk mengamplifikasi daerah pemotongan protein F pada posisi asam nukleotida 4661-5016 dengan panjang pita 316 bp pada mesin reverse transcriptase – polymerase chain reaction (RT-PCR) standar dengan sekun primer depan (FNDFP:GCAGCTGCAGGGATTGTGGT) dan primer belakang
(FNDBP:TCTTTGAGCAGGAGGATGTT) (Nathakumar et al., 2000 dalam Adi et al., 2010). Adapun komposisi dan kondisi proses RT-PCR dilakukan dengan cara sebagai berikut : tabung eppendorf steril diisi dengan R-mix (dNTP, MgSO4 dan buffer) sebanyak 5 µl, primer depan dan primer belakang sebanyak 0,6 µl, enzim SuperScripTM III onestep RT-PCR System with Platinum® Tag DNA Polymerase (Invitrogen) 0,25 µl, aquabides 2,55 µl serta RNA virus sebanyak 1 µl. Tabung Eppendorf tersebut kemudian dimasukkan dalam mesin RT-PCR dan diprogram sebagai berikut : pembalikan RNA menjadi cDNA pada suhu 50°C selama 1 jam, pre-denaturasi pada suhu 95°C selama 7 menit dan denaturasi 94°C selama 45 detik. dilanjutkan proses anneling pada suhu 52°C selama 45 detik dan tahap extension pada suhu 72°C selama 1 menit. Setiap tahap diulangi sebanyak 39 kali. Tahap terakhir penyempurnaan kerja enzim pada suhu 72°C selama 5 menit. Produk RT-PCR yang didapat kemudian dicampur dengan dua loading dye sebanyak enam kali, kemudian dielektroforesis pada agarose 2% selama 25 menit dengan running buffer TAE (Tris Asetic EDTA) satu kali,
kemudian divisualisasikan dengan larutan ethidium bromide (0,5 mg/mL) serta disekuensing.
HASIL DAN PEMBAHASAN
Hasil
Sampel berasal dari beberapa kasus yang diduga mati karena terinfeksi penyakit ND dari beberapa tempat di Pulau Bali. Virus ND diisolasi dan dipropagasi pada telur ayam bertunas (TAB) dengan titer pada uji Ha berkisar 25 – 29 (tabel 1 dan gambar 1a) dan telah dikonfirmasi dengan uji HI, positif ND (gambar 1b). Virus yang dapat menghemaglutinasi sel darrah merah ayam adalah virus avian influenza dan newcastle disease (OIE, 2012; Kencana, 2012). Titer Ha dari masing - masing isolat berkisar 25-29 (Gambar 1a). adanya maternal antibodi embrio pada masing-masing pada TAB mempengaruhi titer virus. Kegenasan tipe virus ND mempengaruhi yang diinokulasi pada TAB dapat mempengaruhi waktu kematian embrio pada TAB dan titer virus (Absalon et al., 2012; Wicaksana et al., 2019).
Tabel 1. Hasil uji hemaglutinasi terhadap keempat sampel VND pada sel darah merah ayam.
|
No |
Isolat |
Kode |
Titer HA |
|
1 |
VNDZayam lokal Badung Bali 2013 |
NDl |
25 |
|
2 |
\"ND ayam ras Klungkung B ali 2014 |
ND2 |
27 |
|
3 |
\ND ayam ras Tabanan 1 Bali 2014 |
ND3 |
29 |
|
4 |
\ND ayam ras Tabanan2 Bali. 2014 |
ND4 |
29 |
Ket. Semua embrio TAB mati pada heri kedua pasca inokulasi.
Uji Rapid HI digunakan untuk mengkonfrimasi virus penyebab yang menghemagltuininasi sel darah merah ayam. Terjadi reaksi pengikatan hiperium serum ND terhadap antigenik virus ND (Jacobson, 1998). Antigenik virus ND yang melakukan pengikatan dengan serum ND adalah protein hemaglutinin-neuraminidase
(HN). Protein tersebut berperan dalam pengikatan virus ND dengan asam sialat sehingga proses hemagltunasi sel darah merah ayam dapat terjadi. Protein HN merupakan protein struktural dan bertindak sebagai pengikat reseptor pada sel terget (Rell et al., 2015; Jin et al., 2017).
2.072
1 500
Gambar 1. Identifkasi virus ND dari cairan alantois terinfeksi uji serologis. a: uji hemaglutinasi (HA), gambar diambil dari bawah mikroplat, lajur (+): kontrol positif, lajur SDM : kontrol sel darah merah. b: uji hambatan hemaglutinasi (HI), gambar diambil dari bawah mirkoplat. lajur K (+/-): kontrol positif (atas)/ kontrol negatif (bawah). Baris sAI (1-4): sumuran hiperimun serum AI. Baris sND (1-4) : sumuran hiperimun serum ND.


Gambar 2. Identifkasi virus ND dari cairan alantois terinfeksi (infected allantoic fluids) dengan uji serologis. a: uji hemaglutinasi (HA), gambar diambil dari bawah mikroplat, lajur (+): kontrol positif, lajur SDM : kontrol sel darah merah. b: uji hambatan hemaglutinasi (HI), gambar diambildari bawah mirkoplat.lajurK (+/-): kontrol positif (atas)/ kontrol negatif (bawah). Baris sAI (1-4): sumuran hiperimun serum AI. Baris sND (1-4) : sumuran hiperimun serum ND.
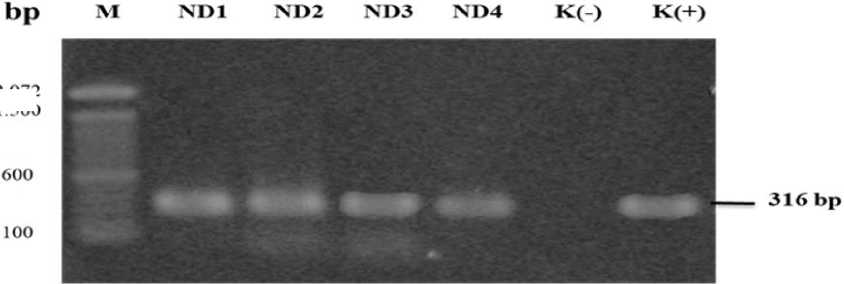
Hasil isolasi RNA yang dilanjutkan dengan identifikasi protein Fusion dengan reaksi RT-PCR, menggunakan sepasang primer. Pasangan primer yang digunakan mengamplifikasi pada posisi nukleotida 4661-5016 yang merupakan daerah pemotongan (cleavage site) dengan produk
16 bp. Produk RT-PCR disekuensing untuk mengetahui sekuen daerah pemotongan. Virulensi VND dapat ditentukan secara molekuler berdasarkan sekuen daerah pemotongan (de Leeuw et al., 2003; 2005; Fernandes et al., 2014).
Hasil Sekuensing produk RT-PCR keempat isolat virus ND isolat diperoleh sekuen nukleotida yang menyandi daerah pemotongan yang menentukan virus ND tersebut virulen atau avirulen yang ditranslasi menggunakan Alignment Explorer-TranslatedProtein Sequences Mega5 (Tamura et al., 2011). Berdasarkan sekuen asam amino keempat isolat virus ND tersebut menunjukkan bahwa semua isolat termasuk virus ND virulen dengan asam amino daerah pemotongan yang multibasik 112R-R-Q-K-R-F117. Sesuai penelitian Otim et al. (2013) meneyatakan sekuen daerah pemotongan 112R/K-R-Q-
K/R-R-F117 merupakan virus ND virulen. Sedangkan virus ND avirulen memiliki sekuen daerah pemotongan 112G/E-K/R-Q-G/E-R-L117 (Collins et al., 1993; de Leeuw et al., 2005). Sekuen daerah pemotongan keempat virus ND isolat lapang tersebut sesuai dengan beberapa laporan penelitian yang menyatakan bahwa sekuen daerah pemotongan 112R-R-Q-K-R-F117 spesifik ditemukan pada virus ND virulen (Adi et al., 2010; Putra et al., 2016). Keempat isolat virus ND tersebut memiliki sekuen daerah pemotongan 112R-R-Q-K-R-F117, sehingga dikelompokan ke dalam virus ND yang virulen.
VNDZayamZrasZTabananI/Bali/2014
VNDZayamZras∕Tabanan2ZBalιZ2014
VNDZayamZlokalZBadungZBalιZ2O13
VNDZayamZras/KlungkungZBali/2014
ChickenZKudusAJI 8Z10
BanjarmasinZOI 0/10 NDVSragen/014/10 NDV Gianyar/013/10
G.7
NDV MM20∕PakistanZ2011
— NDV. TAIWAN
NDV Bah 2007
— Makasar 003/09
----egretZChιna∕GuangxιZ2011
।---chickenZSukorejoZ019Z10
L Bal∣Z020Z10
Malaysia
- NDV Japan NDV, Australia
NDV LaSota
NDV HB1

NDV PHY
V
NDV Ulter strain
0.02
Gambar 3. Pohon filogenetik dari VND berdasarkan sekuen nukleotida gen penyandi protein F pada posisi 4673-4918 (245 bp). Pohon filogenetik dianalisis menggunakan NeighborJoining Tree Method dari Mega5. Skala di bawah pohon menunjukan ukuran jarak antar sekuen. (G.7): cabang isolate yang termasuk ke dalam VND genotip VII. (V) : cabang virus vaksin. Cabang keempat VND isolat lapang ditandai dengan garis tebal.
Daerah pemotongan merupakan daerah protein F yang berperan untuk penetrasi virus ND ke dalam sel selama proses infeksi yang ditentukan oleh enzim mirip protease yang terdapat pada masing-masing sel. Virus ND yang memiliki sekuen daerah pemotongan monobasik (112G/E-K/R-Q-G/E-R-L117) dipotong oleh enzim triptase yang hanya terdapat di daerah pernapasan dan pencernaan. Daerah ini yang menyebabkan virus ND avirulen (misanya: La Sota) hanya menimbulkan pendarahan lokal pada saluran pernapasan dan penyakit yang bersifat kronis pada unggas yang terinfeksi. Sedangkan virus ND yang memiliki sekuen daerah pemotongan multibasik (112R/K-R-Q-K/R-R-F117) dipotong oleh enzim protease kelompok furin (furin-like proteases) yang terdapat pada semua sel. Virus ND virulen yang memiliki daerah pemotongan multibasik dapat menginfeksi secara sistemik dan menimbulkan kematian pada unggas yang terinfeksi dalam waktu yang relatif cepat (Shengqing et al., 2002).
Infeksi VND virulen khususnya tipe velogenik menimbulkan pendarahan pada banyak organ mulai dari pendarahan pada saluran pencernaan (VND velogenik viscerotropik) atau saluran pernapasan (VND velogenik neurotropik) pada awal infeksi (replikasi primer) sampai pendarahan pada organ penting lainnya seperti pada ginjal, pankreas, jantung, hati dan otak (Kim et al., 2011; Putra dan Adi, 2016). Virus ND velogenik dapat menimbulkan morbiditas dan mortalitas yang cukup tinggi pada suatu peternakan unggas walaupun telah divaksin dengan vaksin komersial (Gong dan ZhiZhong, 2011; Absalon et al., 2012).
pohon filogenetik VND berdasarkan sekuen nukleotida protein F mempertegas bahwa keempat VND isolat lapang merupakan virus yang sangat virulen karena termasuk dalam kelompok VND genotipe VII (Gambar 3). Virus ND genotipe VII adalah VND velogenik yang sering outbreak pada peternakan unggas di kawasan Eropa, Afrika dan Asia (Aldous et
al., 2003; Zhang et al., 2011). Hal ini sesuai dengan analisis pohon filognetik yang menunjukkan bahwa semua isolat Indonesia adalah VND genotipe VII. Dalam laporan penelitian Adi et al. (2010) menyatakan bahwa isolat Bali-1/07 yang diisolasi dari outbreak ND di pulau Bali termasuk genotipe VII. Namun, keempat VND isolat lapang berbeda kluster dengan isolat Bali-1/07. Analisis pohon filogenetik juga menunjukkan bahwa keempat VND isolat lapang lebih dekat dengan VND yang berada di luar Bali bahkan lebih dekat dengan isolat NDV MM20/Pakistan/2011. Selain itu, analisis ini memperkuat bahwa keempat VND isolat Bali merupakan virus ND genotipe VII yang sangat virulen.
SIMPULAN DAN SARAN
Simpulan
Berdasarkan analisis filogenetik disimpulkan bahwa keempat virus ND isolat Bali tahun 2013 sampai 2014 adalah virus ND virulen masuk dalam kelompok virus ND genotipe VII.
Saran
Perlu dilakukan pemantauan secara terus meneerus mengingat virus ND ini sangat virulen dan membahayakan.
UCAPAN TERIMAKASIH
Penulis mengucapkan terimakasih kepada Dekan Fakultas Kedokteran Hewan Universitas Udayana atas fasilitas yang diberikan untuk menyeleaikan penelitian ini.
DAFTAR PUSTAKA
Absalón AE, Mariano-Matías A, Vásquez-Márquez A, Morales-Garzón A, Cortés-Espinosa DV, Ortega-García R, Lucio-Decanini E. 2012. Complete genome sequence of a velogenic Newcastle disease virus isolated in Mexico. Virus Genes. 45(2): 304-310.
Adi AAAM, Astawa NM, Putra KSA, Hayashi Y, Matsumoto Y. 2010. Isolation and characterization of a pathogenic newcastle disease virus
from a natural case in Indonesia. J. Vet. Med. Sci. 72(3): 313–319.
Aldous EW, Mynn JK, Banks J, Alexander, DJ. 2003. A molecular epidemiological study of avian paramyxovirus type 1 (Newcastle disease virus) isolates by phylogenetic analysis of a partial nucleotide sequence of the fusion protein gene. Avian Pathol. 32(3): 239257.
Alexander DJ. 1987. Taxonomy and nomenclature of avian
paramyxoviruses. Avian Pathol. 16: 547-552.
Collins MS, Bashiruddin JB, Alexander DJ. 1993. Deduced amino acid sequences at the fusion protein cleavage site of Newcastle disease viruses showing variation in antigenicity and pathogenicity. Archives Virol. (128): 363-370.
de Leeuw OS, Hartog L, Koch G, Peeters BPH. 2003. Effect of fusion protein cleavage site mutations on virulence of Newcastle disease virus: non-virulent cleavage site mutants revert to virulence after one passage in chicken brain. J. General Virol. 84: 475–484.
de Leeuw OS, Koch G, Hartog L, Ravenshort, N, Peeters, BPH. 2005. Virulence of Newcastle disease virus is determined by the cleavage site of the fusion protein and by both the stem region and globular head of the haemagglutinin–neuraminidase protein. J. General Virol. 86: 1759–1769.
Dortmans JCFM. 2011. Virulence Determinants of Newcastle Disease Virus. Dissertation. Universitas Utrecht, Nederlands.
Fernandes CC, Varani, AM., Lemos,
EGM, de Miranda, VFO, Silva KR, Fernando FS, Montassier MFS, Montassier HJ. 2014. Molecular and phylogenetic characterization based on the complete genome of a virulent pathotype of Newcastle disease virus isolated in the 1970s in Brazil. Infect. Genet. Evol. 26: 160–167.
Glickman RL, Syddall RJ, Iorio RM, Sheehan JP, Bratt MA. 1988. Quantitative basic residue requirements in the cleavage-activation site of the fusion glycoprotein as a determinant of virulence for newcastle disease virus. J. Virol. 62(1): 354-356.
Gong YY, ZhiZhong CUI. 2011. Epitope variation in the Newcastle disease virus HN gene under antibody immune selective pressure in cell culture. Sci China Life Sci. 54(5): 474–479.
Jacobson RH. 1998. Validation of serological assays for diagnosis of infectious diseases. Rev. Sci. Tech. Off. Int. Epiz. 17(2): 469-486.
Kencana GAY. 2012. Penyakit Unggas. Udayana Universiati Press. Pp. 34 – 52.
Kencana GAY, Kardena IM, Mahardika, IGN. 2012. Peneguhan diagnosis
penyakit Newcastle disease lapang pada ayam buras di bali menggunakan
teknik RT-PCR J. Kedokteran Hewan. 6(1).
Kim SH, Subbiah M, Samuel AS, Collins PL, Samal SK. 2011. Roles of the fusion and hemagglutinin
neuraminidase proteins in replication, tropism, and pathogenicity of avian paramyxoviruses. J. Virol. 85(17):
8582–8596.
Kusmaedi. 2001. Teknik uji
hemaglutination inhibition Untuk mengukur tingkat kekebalan terhadap Newcastle disease dan egg drop Syndrome’76. Temu Teknis Fungsional Non Peneliti, Balai Penelitian Veteriner.
Lee DH, Kwon JH, Noh JY, Park JK, Yuk SS, Erdene TO, Nahm SS, Kwon YK, Lee SW, Song CS. 2016.
Viscerotropic velogenic Newcastle disease virus replication in feathers of infected chickens. J. Vet. Sci. 17(1): 115–11.
McGinnes LW, Morrison TG. 2003. Inhibition of Receptor Binding Stabilizes Newcastle Disease Virus HN and F Protein-Containing Complexes. J. Virol. 80(6): 2894–2903.
Morrison TG. 2003. Structure and function of a paramyxovirus fusion protein. Biochim. Biophysica Acta. 1614: 73– 84.
OIE. 2012. Newcastle Disease. Version adopted by the World Assembly of Delegates, 2: 3-14.
Otim MO, Christensen H, Jørgensen PH, Handberg KJ, Bisgaard M. 2004. Molecular Characterization and Phylogenetic Study of Newcastle Disease Virus Isolates from Recent Outbreaks in Eastern Uganda. J. Clin. Microbiol. 42(6): 2802–2805.
Putra IGAA, Adi AAAM. 2016. Confirmation of avian paramyxovirus type 1 (APMV-1) infection by
histopathology, serology and molecular method (Indonesian). J. Ked. Hewan. 10(2): 162-165.
Piacenti AM, King DJ, Seal BS, Zhang J, Brown CC. 2006. Pathogenesis of Newcastle disease in commercial and specific pathogenfree Turkeys
experimentally infected with isolates of different virulence. Vet. Pathol. 43: 168-170
Rell F, Adi AAAM, Mahardika IGNK. 2015. Virulensi virus newcastle disease isolat lapang berdasarkan analisis bioinformatika gen protein
hemaglutinin – neuraminidase. J. Ilmu dan Kesehatan Hewan. 3(1): 17-28.
Shengqing Y, Kishida N, Ito H, Kida H, Otsuki K, Kawaoka Y, Ito T. 2002.
Generation of velogenic newcastle disease viruses from a nonpathogenic waterfowl isolate by passaging in chickens. Virology. 301: 206–211.
Tamura K, Peterson D, Peterson N, Stecher G, Nei M, Kumar S. 2011. MEGA5: Molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods. Mol. Biol. Evol. 28(10): 2731–2739.
Wicaksana IGHP, Adi AAAM, Kardena IM. Pathological lesions in chicken embryo caused by newly virulent isolate of newcastle disease virus. J. Vet. 20(3): 337-344.
Yeo SG, Nagy E, Krell PJ. 2003. Indirect method for prediction of
hemagglutination inhibition antibody titers to Newcastle disease virus in chickens by titration of antibodies in egg yolk. J. Vet. Diagn. Invest. 15: 184– 187.
Yusoff K, Tan WS. 2001. Newcastle disease virus: macromolecules and opportunities. Avian Pathol. 30: 439– 455.
Zhang S, Wang X, Zhao C, Liu D, Hu Y. 2011. Phylogenetic and pathotypical analysis of two virulent newcastle disease viruses isolated from domestic ducks in China. PLoS ONE. 6(9):
e2500.
74
Discussion and feedback