Morphologicals and molecular characteristics of regang prawn (Macrobrachium sintangense De Man, 1898) from Special Region of Yogyakarta
on
JURNAL BIOLOGI UDAYANA 25(1): 7-17
P ISSN: 1410-5292 E ISSN: 2599-2856
Karakter morfologis dan molekuler udang regang (Macrobrachium sintangense De Man, 1898) dari Daerah Istimewa Yogyakarta
Morphologicals and molecular characteristics of regang prawn (Macrobrachium sintangense De Man, 1898) from Special Region of Yogyakarta
Rury Eprilurahman, Rini Rahmawati*, Lukman Hakim, Tuty Arisuryanti, Zuliyati Rohmah, Trijoko
Fakultas Biologi, Universitas Gadjah Mada, Indonesia
Jl. Teknika Selatan, Sekip Utara, Sleman, Daerah Istimewa Yogyakarta, Indonesia 55281
*Email: rini.rahmawati@mail.ugm.ac.id
Diterima 18 Oktober 2020 Disetujui 25 Januari 2021
INTISARI
Genus Macrobrachium memiliki anggota spesies yang banyak dan terdistribusi di berbagai negara diantaranya Indonesia, Malaysia, Thailand, Laos dan Vietnam. Salah satu spesies anggota genus Macrobarachium yang dapat ditemukan di Indonesia adalah Macrobrachium sintangense atau lebih dikenal sebagai udang regang. Identifikasi terhadap spesies ini penting dilakukan sebagai upaya untuk memperluas kajian mengenai udang regang di Daerah Istimewa Yogyakarta. Identifikasi terhadap spesies ini dilakukan secara morfologis maupun molekuler dengan gen 16S. Tujuan dari penelitian ini adalah untuk mengetahui karakter morfologis dan molekuler dari M. sintangense. Identifikasi morfologis dilakukan dengan mengidentifikasi karakter morfologi, morfometri, dan meristik sesuai kunci identifikasi. Analisis secara molekuler dilakukan dengan isolasi, amplifikasi, elektroforesis, dan visualisasi DNA dengan UV iluminator, DNA di sekuensing oleh 1st Base. Hasil penelitian ini menunjukkan karakter morfologis berupa rostrum sedikit melengkung ke atas atau seperti pisau, post antennular carapace yang membulat, preanal carina tidak ada, terdapat pubescene pada setengah proksimal hingga dua per tiga chela. Secara morfologis semua sampel teridentifikasi sebagai anggota genus Macrobrachium. Ada delapan (8) spesimen dapat di identifikasi sampai tingkat spesies, sebagai M. sintangense. Hasil identifikasi dua (2) spesimen secara molekuler menunjukkan nilai similaritas tinggi dan hubungan kekerabatan yang dekat terhadap M. sintangense dari sampel GenBank serta hubungan kekerabatan yang dekat dengan M. sintangense dari Tukad Panti, Bali, Indonesia. Kesimpulan pada penelitian ini secara morfologis dan molekuler adalah sampel yang ditemukan pada sungai Winongo, Tambakbayan, Code dan Opak di DIY adalah M. sintangense.
Kata kunci: Identifikasi, Macrobrachium, morfologi, morfologis, sungai DIY
ABSTRACT
Macrobrachium genus has many species members and distributed in various countries including Indonesia. One of the species belonging to the Macrobarachium genus that can be found in Indonesia was Macrobrachium sintangense or known as Sunda River Prawn. Identification of this species is important as an effort to expand studies on Sunda River Prawn in the Region of Yogyakarta. Identification of this species was carried out morphologically and molecularly with the 16S gene. The purpose of this study was to
determine the morphological and molecular characters of M. sintangense. Morphological identification is done by identifying morphological, morphometric, and meristic characters according to the identification key. Molecular analysis was carried out by isolation, amplification, electrophoresis, and DNA visualization with UV illuminators, DNA sequencing by 1st Base. The results of this study indicate the morphological character of the rostrum slightly curved upwards or like a knife, rounded post antennular carapace, absent preanal carina, pubescene in the proximal half to two thirds of the chela. Morphologically, all samples were identified as members of the genus Macrobrachium. There are eight (8) specimens identifiable to the species level, as M. sintangense. The results of molecular identification of two (2) specimens showed a high similarity value and a close relationship with M. sintangense from GenBank samples and also close relationship with M. sintangense from Tukad Panti, Bali, Indonesia. The conclusion of this research is that morphologically and molecularly, the samples found in the Winongo.
Keywords: Identification, Macrobrachium, morphology, morphology, DIY river
PENDAHULUAN
Udang regang atau yang lebih dikenal sebagai Macrobrachium sintangense merupakan salah satu spesies udang dari genus Macrobrachium yang umum ditemukan di Indonesia (Putranto, 2014). Udang ini dapat ditemukan di kolam, waduk, danau, saluran irigasi, sungai dan estuari (Putranto, 2014). Udang regang memiliki kandungan protein yang tinggi yaitu 60% dari berat kering tubuhnya, kadar lemak yaitu 7-12% dan kadar karbohidrat kurang dari 10% (Said et al., 2014). Udang ini biasa digunakan sebagai pakan hidup untuk hewan budidaya seperti ikan dan juga dikonsumsi oleh masyarakat (Putranto, 2014).
M. Sintangense tersebar luas di Thailand, Malaysia, Kalimantan, Sumatera dan Jawa (Sabar, 1979). Berdasarkan penelitian sebelumnya oleh Trijoko et al. (2015) diketahui bahwa udang ini ditemukan di Daerah Istimewa Yogyakarta (DIY) yaitu di aliran sungai Opak. DIY memiliki beberapa aliran sungai diantaranya adalah sungai Opak, Code, Winongo, dan Tambakbayan yang belum dilakukan kajian terhadap M. Sintangense. Sehingga penting dilakukan kajian terhadap spesies ini di berbagai sungai yang ada di DIY sebagai upaya untuk memperluas kajian terhadap udang regang di DIY.
Identifikasi terhadap M. sintangense dapat dilakukan secara morfologis maupun molekuler.
Identifikasi secara morfologis memerlukan karakter identifikasi berupa karakter morfologi, morfometri, dan meristik. Namun, karakter morfologis utama pada udang Macrobrachium bergantung pada keadaan lingkungan, perubahan dalam siklus hidup, jenis kelamin, dan dominansi sosial (Page & Hughes, 2011). Sehingga diperlukan cara identifikasi yang lebih akurat untuk mendukung hasil identifikasi morfologi M. sintangense yaitu melalui identifikasi molekuler. Salah satu metode molekuler yang dikembangkan adalah menggunakan DNA barcoding dari gen mitokondria 16S rRNA (Munasinghe, 2010; Sharma et al., 2014). Gen 16S sering digunakan untuk studi di pertengahan yaitu pada tingkat kategori seperti familia atau genera (Arif & Khan, 2009). Gen pengkode RNA ribosomal 16S adalah gen yang lestari (conserved).
Melalui penelitian ini diharapkan dapat memberikan kontribusi bagi kelengkapan database mengenai karakterisasi genetik pada udang regang (M. sintangense) serta sebagai sumber informasi ilmiah yang dapat digunakan dalam budidaya dan konservasi udang regang. Selain itu, penelitian ini diharapkan dapat melengkapi data keanekaragaman spesies Macrobrachium di Yogyakarta.
MATERI DAN METODE
Tempat dan waktu penelitian
Penelitian dilaksanakan dengan pengambilan sampel udang di empat sungai di Daerah Istimewa
Yogyakarta dengan rentang waktu dari tahun 2012 hingga 2016. Sampel diambil dari sungai Opak, Code, Winongo, serta Tambakbayan. Selanjutnya dilakukan pengambilan data morfologi dilakukan di Laboratorium Sistematika Hewan Fakultas Biologi UGM pada bulan November 2019 dan dilanjutkan pada bulan Februari hingga Agustus 2020. Pengambilan data molekuler dilakukan pada bulan Februari hingga Agustus 2020 di Laboratorium Genetika dan Pemuliaan Fakultas Biologi UGM.
Bahan dan alat
Bahan untuk preservasi udang hasil sampling menggunakan alkohol 96%. Bahan untuk isolasi DNA adalah jaringan dari abdomen udang regang
Metode
Identifikasi morfologis dilakukan dengan cara mengamati bagian tubuh udang secara langsung maupun menggunakan bantuan mikroskop digital. Identifikasi ini dilakukan dengan mengamati karakter morfologi, meristik, dan morfometri sesuai dengan jurnal kunci identifikasi. Pada penelitian ini, kunci identifikasi mengacu pada jurnal Chace & Bruce (1993) dan Wowor et. al, (2004) mengenai udang Macrobrachium sintangense. Pengamatan karakter morfologi berupa ada tidaknya bagian tubuh tertentu pada udang sesuai kunci identifikasi. Pengamatan karakter meristik dilakukan dengan cara menghitung jumlah bagian tubuh tertentu pada udang sesuai kunci identifikasi. Sementara pengamatan terhadap karakter morfometri dilakukan dengan cara mengukur bagian tubuh udang sesuai kunci identifikasi.
Identifikasi molekuler dilakukan dengan melakukan isolasi, amplifikasi, elektroforesis, dan dokumentasi pita DNA hasil elektroforesis. Isolasi DNA lobster dilakukan mengikuti petunjuk QIAGEN DNEasy Blood & Tissue Kits. Amplifikasi DNA menggunakan primer 16 Sar dan 16 Sbr dilakukan berdasarkan Protokol Bioline MyTaq Red Mix. Amplifikasi DNA dilakukan dengan mesin PCR menggunakan lima
sebanyak 50 mg, 180 µl buffer ATL, 20 µl proteinase-K, 200 µl buffer AL, 200 µl ethanol absolut 100%, 500 µl buffer AW-1, 500 µl buffer AW-2, 250 µl buffer AE. Komponen bahan untuk PCR yaitu DNA template (5,5 μl), ddH2O (3 μl), MgCl2 (1 μl ), primer 16S rRNA. Primer yang digunakan adalah primer 16 Sar (5’CCTG TTTANCAAAAACAT-3’) sebagai primer forward dan 16 Sbr (5’AGATAGAAACCAACCTGG-3’) sebagai primer reverse. Bahan untuk elektroforesis yaitu gel agarose 1% dan pewarna Florosafe DNA Stain (2 µl). Alat yang digunakan untuk identifikasi morfologis adalah kamera mikroskop digital, pinset, jangka sorong, dan jurnal kunci identifikasi Macrobrachium sintangense.
Tahapan yaitu predenaturation pada suhu 95ºC, denaturation pada suhu 95ºC, annealing pada suhu 50ºC, extention pada suhu 72ºC, dan final extention pada suhu 72ºC. Hasil PCR kemudian dielektroforesis menggunakan gel agarose 1% tegangan 100 volt dalam buffer TBE (Tris Borat EDTA) dan digunakan DNA ladder sebagai marker. Pita DNA kemudian di dokumentasikan dengan UV illuminator. Apabila pita DNA terlihat jelas dan memenuhi standar yaitu memiliki panjang basepair ±500 bp maka DNA dapat di lanjutkan ke proses sekuensing di PT. Genetika Science Indonesia, Jakarta, Indonesia.
Analisis data
Analisis data meliputi analisis data morfologis dan molekuler. Analisis morfologi dilakukan dengan cara mengkonversi data karakter morfologis yang diperoleh menjadi matriks biner (0-1). Sifat yang tidak dimiliki oleh suatu individu ditulis dengan angka 0. Pada karakter morfometri, nilai yang berada dalam kisaran deviasi ditulis dengan angka 1 dan yang berada di luar kisaran deviasi ditulis dengan angka 0. Matriks data dibuat dengan program Ms. Excel 2007. Analisis clustering menggunakan metode UPGMA (Unweighted Pair Group withArithmetic Average) dengan program MVSP 3.1 (Multi Variate Statistical Package). Analisis data
molekuler dilakukan dengan mengolah hasil sekuensing DNA sampel. Sekuen DNA yang diperoleh diedit menggunakan software Genestudio kemudian di BLAST di NCBI untuk mencocokan sampel dengan sekuens di GenBank. Sekuen yang sudah terverifikasi sebagai Macrobrachium sintangense kemudian dilakukan anlisis jarak genetik, pohon filogenetik, variasi genetik, dan situs polimorfik.
HASIL
Berdasarkan sampling yang telah dilakukan pada sungai Code, Winongo, Opak, dan Tambakbayan maka didapatkan hasil seperti pada Tabel 1. Pada penelitian ini didapatkan 15 individu hasil sampling yang ditemukan baik pada bagian tengah maupun hilir sungai. Hasil sampling kemudian diidentifikasi secara morfologis dan molekuler untuk mengetahui karakter molekuler dan morfologis dari sampel M. sintangense yang didapat.
Tabel 1. Hasil sampling udang di Sungai Opak, Winongo, Code, dan Tambakbayan
|
No |
Sungai |
Bagian Sungai |
Keterangan |
Singkatan Identifikasi Morfologis |
Singkatan Identifikasi Molekuler |
|
1 |
OT1 | ||||
|
2 |
Tengah |
3 individu |
OT2 |
MS1 | |
|
3 |
OT3 | ||||
|
4 |
Opak |
OH1 | |||
|
5 6 |
Hilir |
4 individu |
OH2 OH3 |
MS2 | |
|
7 |
OH4 | ||||
|
8 9 |
Winongo |
Tengah |
2 individu |
WT1 WT2 |
MS3 |
|
10 |
CT1 | ||||
|
11 |
Code |
Tengah |
3 individu |
CT2 |
MS4 |
|
12 |
CT3 | ||||
|
13 |
TH1 | ||||
|
14 |
Tambakbayan |
Hilir |
3 individu |
TH2 |
MS5 |
|
15 |
TH3 |
Identifikasi Morfologis
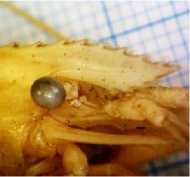
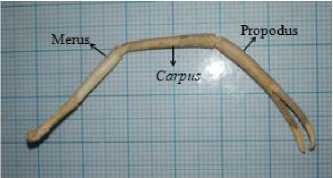
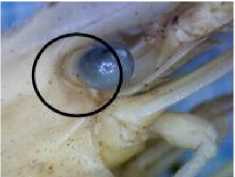
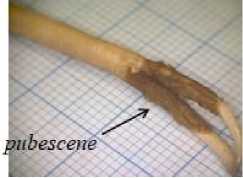
Identifikasi secara morfologis dilakukan dengan melakukan pengamatan karakter morfologi, meristik, dan morfometri. Hasil pengamatan mengacu pada ciri karakter morfologis yang dimiliki oleh Macrobrachium sintangense menurut jurnal Chace & Bruce (1993) dan Wowor et. al, (2004). Berikut ini merupakan karakter M.sintangense yang ditemukan pada sampel yang diteliti (Gambar 1).
Berdasarkan penelitian yang telah dilakukan maka didapatkan hasil seperti pada Tabel 2. Hasil penelitian menununjukan bahwa dalam penelitian didapat 15 sampel yang semuanya teridentifikasi sebagai udang anggota genus Macrobrachium. Hanya sampel OT1, OT2, OH2, WT2, CT1, CT2, CT3 dan TH1 yang dapat teridentifikasi hingga tingkat spesies sebagai Macrobrachium sintangense.
(a)
(b)
(c)
(d)
Gambar 1. Karakter M. sintangense pada sampel yang diteliti
Tabel 2. Hasil analisis morfologis udang dari Sungai Code, Opak, Winongo, dan Tambakbayan
|
No |
Sungai |
Bagian Sungai |
Keterangan |
Singkatan Identifikasi Morfologis |
Hasil Identifikasi Morfologi |
|
1 |
Jantan |
OT1 |
M. sintangense | ||
|
2 |
Tengah |
Jantan |
OT2 |
M. sintangense | |
|
3 |
Juvenile |
OT3 |
Macrobrachium | ||
|
4 |
Opak |
Pleopod kedua hilang |
OH1 |
Macrobrachium | |
|
5 |
Hilir |
Jantan |
OH2 |
M. sintangense | |
|
6 |
Juvenil |
OH3 |
Macrobrachium | ||
|
7 |
Juvenil |
OH4 |
Macrobrachium | ||
|
8 |
Winongo |
Tengah |
Juvenil |
WT1 |
Macrobrachium |
|
9 |
Jantan |
WT2 |
M. sintangense | ||
|
10 |
Jantan |
CT1 |
M. sintangense | ||
|
11 |
Code |
Tengah |
Jantan |
CT2 |
M. sintangense |
|
12 |
Jantan |
CT3 |
M. sintangense | ||
|
13 |
Jantan |
TH1 |
M. sintangense | ||
|
14 |
Tambakbayan |
Hilir |
Juvenil |
TH2 |
Macrobrachium |
|
15 |
Juvenil |
TH3 |
Macrobrachium |
Identifikasi Molekuler
Pada penelitian ini, identifikasi secara molekuler dilakukan dengan gen mitokondrian 16S. DNA yang telah diisolasi, diamplifikasi dan dielektroforesis kemudian dilihat pita DNA nya melalui UV illuminator seperti pada gambar 1. Identifikasi secara molekuler hanya dilakukan terhadap sampel MS1, MS3, MS4, dan MS5. Sampel MS1 dan MS5 tidak dapat diidentifikasi secara molekuler dikarenakan terjadinya kontaminasi dan rusaknya jaringan tubuh. Rusaknya jaringan tubuh ini dapat dikarenakan waktu preservasi yang lama pada suhu rungan. Pada penelitian ini, sampel MS3 dan MS4 memiliki panjang fragmen gen mitokondria 16S sebesar ±500 bp (Gambar 2).
2000 bp
1000 bp
500 bp
250 bp IOObp
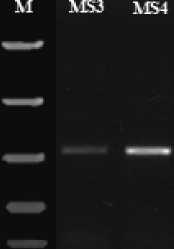
Gambar 2. Pita DNA Individu MS3 dan MS4 hasil amplifikasi PCR dan M merupakan marker DNA.
Sampel yang telah memenuhi standar panjang fragmen DNA kemudian di sekuensing. Sekuen DNA yang diperoleh diedit menggunakan software Genestudio kemudian di BLAST di NCBI untuk mencocokan sampel dengan sekuens di GenBank. Hasil BLAST terlihat pada Tabel 3. Hasil BLAST menunjukan bahwa sampel MS3 memiliki nilai Identity tertinggi dengan sampel JQ362451.1 M. sintangense dan JQ362450.1 M. sintangense dari sampel GenBank. Nilai Query cover tertinggi hasil BLAST terhadap sampel MS3 dan MS4 adalah sebesar 99%.
Setelah dilakukan BLAST, selanjutnya dilakukan analisis jarak genetik menggunakan softwere Mega 7.0.21 dan hasilnya seperti yang terlihat pada Tabel 4. Jarak gentik terkecil sampel MS3 dan MS4 terhadap sampel GenBank adalah sebesar 0,60% dan 1% terhadap sampel U1 Macrobrachium sintangense Tukad Panti dari Bali, Indonesia penelitian sebelumnya. Jarak genetik terkecil kedua adalah antara sampel MS3 dan MS4 terhadap sampel sampel JQ362451.1 M. sintangense dan JQ362450.1 M. sintangense dari GenBank yaitu sebesar 1,40% dan 1,8%.
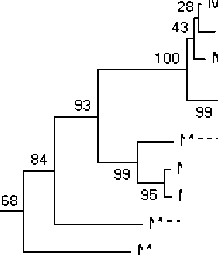
Berdasarkan nilai jarak genetik yang didapat, kemudian direkonstruksi pohon filogenetik sperti yang terlihat pada Gambar 3. Pada pohon
filogenetik yang terbentuk terlihat bahwa sampel MS3 dan MS4 memiliki hubungan kekerabatan yang dekat dengan sampel U1 M. sintangense Tukad Panti, Bali, Indonesia dan juga hubungan kekerabatan yang dekat dengan sampel GenBank M. sintangense JQ362451.1 dan M. sintangense JQ362450.1. Pohon filogenetik kemudian dianalisis lebih lanjut pada variasi genetik yang terjadi antara sampel MS3 dan MS4 terhadap
sampel GenBank M. sintangense JQ362451.1 seperti pada Tabel 5. Berdasarkan data variasi genetik diketahui bahwa panjang fragmen DNA sampel adalah 558 bp, jumlah variable site sebanyak 14 site dengan 0 parsimony site dan 14 polymorphic site. Nucleotide diversity (π) sebesar 0,01691 ± 0,00693 dan haplotipe diversity sebesar 1 ± 0,272.
Tabel 3. Hasil Similaritas dengan BLAST
|
No |
Sampel NCBI |
Spesimen | ||
|
MS3 |
MS4 | |||
|
1 |
JQ362451.1 M. sintangense |
Identity |
97.30% |
97.48% |
|
Query cover |
99% |
99% | ||
|
2 |
JQ362449.1 M. sintangense |
Identity |
93.33% |
93.51% |
|
Query cover |
99% |
99% | ||
|
3 |
JQ362450.1 M. sintangense |
Identity |
97.30% |
97.48% |
|
Query cover |
99% |
99% | ||
|
4 |
FM986641.1 M. sintangense |
Identity |
93.81% |
93.60% |
|
Query cover |
92% |
92% | ||
|
5 |
JQ362452.1 M. tratense |
Identity |
93.33% |
93.51% |
|
Query cover |
99% |
99% | ||
|
6 |
EU493147.1 M. nipponense |
Identity |
92.45% |
92.62% |
|
Query cover |
97% |
97% | ||
Tabel 4. Jarak Genetik (%) Udang Macrobrachium sintangense. Setiap satu sampel model dibandingkan dengan sampel dari GenBank NCBI. Angka dengan warna biru merupakan Standard Error (SE), sedangkan warna hitam adalah jarak genetik.
|
(Z) S |
(Z) S |
S CM K |
σ∖ ⅛ CM K |
O ⅛ CM K sz⅛ |
∞ .R | ||
|
MS4 |
0,30% |
0,60% |
1,10% |
0,60% |
1,20% |
0,40% | |
|
MS3 |
0,40% |
0,50% |
1,10% |
0,50% |
1,10% |
0,30% | |
|
JQ362451.1 M. sintangense |
1,80% |
1,40% |
1,10% |
0% |
1,20% |
0,50% | |
|
JQ362449.1 M. sintangense |
6,40% |
5,90% |
6,20% |
1,10% |
0,70% |
1,10% | |
|
JQ362450.1 M. sintangense |
1,80% |
1,40% |
0% |
6,20% |
1,20% |
0,50% | |
|
FM986641.1 M. sintangense |
6,60% |
6,10% |
6,40% |
2,50% |
6,40% |
1,20% | |
|
U1 Macrobrachium sintangense Tukad Panti |
1% |
0,60% |
1,20% |
6,20% |
1,20% |
6,40% |
28
87
U1 Macrobrachium Sintangense Tukad Panti
Macrobrachium Sintangense JQ362451.1
Macrobrachium sintaπgeπse JQ362450.1
Macrobrachium sintaπgeπse FM986641.1
Macrobrachium Sintangense JQ362449.1
Macrobrachium tratense JQ362452.1
Macrobrachium nipponense EU493147.1
Macrobrachium asperulum AB250452.1 — Macrobrachium idella JF774075.1
5
Macrobrachium gracilirostre EU493142.1
- Macrobrachium jaroense EU493146.1
Macrobrachium Iatimanus DQ194937 1
21
Macrobrachium carcinus HM352450 1
37
Macrobrachium malcolmsonii KR736341.1
Macrobrachium canarae JF774072.1
78
37
Macrobrachium Villosimanus HM751940.1
I— Macrobrachium dacqueti JF792431.1 10□ ∣ Macrobrachium rosenbergii KM610148.1
---------- Caridina pseudodeπticulata DQ194973.1
8,858
Gambar 3. Pohon filogeni Neighbor-Joining dari sampel penelitian, beberapa spesies dari genus Macrobrachium ditambah dengan satu spesimen outgroup dari genus Caridina
Tabel 5. Variasi Genetik Udang Jenis Macrobrachium sintangense. Sampel yang diteliti (2 sekuen) dan GenBank (1 sekuen) berdasarkan gen rRNA mitokondria
|
Jumlah Jumlah pes es p Individu Haplotipe |
Nucleotide Haplotype Parsimony Variable Monomorphicsites Diversity Diversity Sites Sites (π) (Hd) |
|
M. sintangense 517 4 4 |
2 11 501 0,01139 ± 1,00 ± 0,00333 0,177 |
Berdasarkan data variasi genetik, diketahui tedapat 4 haplotipe dari sample yang diteliti yaitu MS3, M. sintangense Tukad Panti, MS4 dan sampel GenBank M. sintangense JQ362451.1
yang berasal dari Thailand (Tabel 6). Berdasarkan hasil situs polimorfik pada Tabel 7 terdapat 9 transisi dan 2 transversi.
Tabel 6. Data Haplotipe Udang Macrobrachium sintangense. Udang dalam penelitian (MS3 dan MS4) dan dari Genbank (JQ362451.1).
|
Haplotipe |
Jumlah GenBank Asal Author Sampel Accession Number |
|
1 |
1 JQ362451.1 Thailand Pinpart et al., 2012- Unpublished |
|
2 |
1 MS4 Yogyakarta, Indonesia Dalam penelitian |
|
3 |
1 MS3 Yogyakarta,Indonesia Dalam penelitian |
|
4 |
1 M. sintangense Tukad Panti Tukad Panti, Bali, Indonesia Unpublished |
tabel 7. situs polimorfik antara sampel yang diteliti (ms3 dan ms4) dengan sampel GenBank.
|
Sampel |
Situs Polimorfik | ||||||||||
|
1 |
6 7 |
8 8 |
1 6 1 |
2 5 8 |
2 8 8 |
2 9 6 |
2 9 7 |
4 5 1 |
4 8 2 |
5 1 7 | |
|
JQ362451.1 |
C |
G |
T |
G |
A |
C |
A |
A |
T |
T |
T |
|
MS4 |
T |
A |
G |
A |
G |
T |
G |
G |
G |
C |
. |
|
MS3 |
T |
A |
. |
. |
G |
T |
G |
G |
G |
C |
. |
|
M. sintangense Tukad Panti |
T |
. |
. |
. |
G |
. |
G |
G |
G |
C |
C |
|
Ctf |
Ctf H |
H |
Ctf H |
Ctf |
Ctf |
Ctf |
∞ Ctf |
∞ |
∞ Ctf |
’S Ctf | |
|
Transisi | |
|
Transversi |
PEMBAHASAN
Hasil sampling berupa 15 sampel udang yang didapat dari Sungai Opak, Code, Winongo, dan Tambakbayan. Pada Tabel 1 diketahui bahwa sampel yang didapat berjumlah 15 sampel yang seluruhnya dianalisis secara morfologis. Sementara analisis secara molekuler hanya dilakukan terhadap sampel MS1, MS3, MS4, dan MS5. Sampel tersebut dianggap telah mewakili hasil sampling secara keseluruhan dari keempat sungai tersebut. Pemilihan sampel ini didasari pada perwakilan sampel pada setiap sungai. Sampel MS1 dan MS5 tidak dapat diidentifikasi secara molekuler dikarenakan terjadinya kontaminasi dan rusaknya jaringan tubuh. Rusaknya jaringan tubuh ini dapat dikarenakan waktu preservasi yang lama pad suhu rungan.Sampel MS3 merupakan sampel yang berasal dari Sungai Code dan sampel MS4 merupakan sampel yang berasal dari Sungai Winongo.
Analisis morfologis dilakukan terhadap semua sampel yang didapat pada saat sampling. Karakter yang digunakan dalam analisis morfologis meliputi tiga karater yaitu karakter morfologi, meristik, dan morfometri. Karakter morfologi berupa ada tidaknya bagian tubuh tertentu. Karakter morfometri berupa ukuran suatu bagian
tubuh. Sementera karakter meristik berupa jumlah dari bagian tubuh yang dapat dihitung. Berdasarkan hasil analisis yang diperoleh pada Tabel 1 diketahui bahwa semua sampel teridentifikasi dalam genus Macrobrachium. Karakteristik dari genus Macrobrachium adalah karapas tidak memiliki branchiostegal spine namun memiliki hepatic spine, Dactylus pada tiga pasang pereiopod terakhir genus Macrobrachium bersifat non bifida atau sederhana. Selain itu, tidak terdapat appendix pada endopod pleopod pertama individu jantan (Chace & Bruce, 1993). Menurut Holthuis (1950) ciri lain yang dapat digunakan untuk mengetahui anggota genus Macrobrachium adalah pleura pada segmen kedua membulat menutupi pleura pada segmen pertama dan ketiga. Dari keseluruhan sampel, hanya beberapa sampel saja yang teridentifikasi sebagai sampel Macrobrachium jantan dewasa sehingga dapat dianalisis lebih lanjut untuk menentukan spesies. Macrobrachium jantan dewasa dapat diketahui dengan adanya spina masculina.
Sampel Macrobrachium jantan dewasa yang teridentifikasi sebagai M. Sintangense adalah sampel OT1, OT2, OH2, WT2, CT1, CT2, CT3 dan TH1. Identifikasi ini dilakukan berdasarkan hasil identifikasi morfologis pada Gambar 1. Ciri morfologi dari M. sintangense diantaranya berupa
rostrum sedikit melengkung ke atas atau seperti pisau, post antennular carapace yang membulat, preanal carina tidak ada, terdapat pubescene pada setengah proksimal hingga dua per tiga capit (Wowor et. al, 2004). Ciri meristik dari M. sintangense menurut Wowor et. al, (2004) dan Chace & Bruce (1993) jumlah gigi pada dorsal rostrum berkisar antara 9 dan 13, sementara jumlah gigi rostrum bagian ventral kurang dari 7 gigi. Sementara ciri morfometri dapat dilihat dari pereiopod kedua dengan carpus lebih panjang daripada. Namun, ukuran carpus yang lebih panjang dari merus tidak dapat menjadi patokan utama, hal tersebut dikarenakan ukuran tubuh udang dapat berubah ukuran sepanjang hidupnya. Pada penelitian ini, sampel OT1, OT2, OH2, WT2, CT1, CT2, CT3 dan TH1 memiliki karakter yang sesuai dengan kunci identifikasi M. sintangense sehingga teridentifikasi sebagai sampel M.sintangense.
Analisis molekuler meliputi isolasi DNA, amplifikasi DNA, elektroforesis, hingga dokumentasi pita DNA. Berdasarkan analisis tersebut diketahui bahwa pita DNA gen mitokondria 16S pada individu MS3 dan MS4 memiliki panjang fragmen ±500 bp (Gambar 2). Gen mitokondria 16S sebaiknya sepanjang 500 bp untuk dapat dianalisis pada BLAST, atau terburuknya adalah sepanjang 400 bp (Yang et al., 2014). Berdasarkan penyataan tersebut, maka panjang gen mitokondria ini telah memenuhi persyaratan untuk dapat dilakukan analisis similaritas dengan menggunakan BLAST. Sehingga sampel MS3 dan MS4 tersebut kemudian di sekuensing.
Hasil sekuensing sampel MS3 dan MS4 kemudian dianalisi menggunakan BLSTN untuk mencocokan dengan sampel yang ada di GenBank. Berdasarkan hasil BLAST pada Tabel 3 diketahui bahwa sampel MS3 memiliki nilai identity tertinggi dengan sampel dengan sampel JQ362451.1 M. sintangense dan JQ362450.1 M. sintangense dari sampel GenBank yaitu sebesar 97,30%. sampel MS3 memiliki nilai identity tertinggi dengan sampel dengan sampel
JQ362451.1 M. sintangense dan JQ362450.1 M. sintangense dari sampel GenBank yaitu sebesar 97,48%. Nilai identity diatas 97% menunjukan bahwa sampel yang diteliti signifikan satu spesies dengan sampel M. sintangense dari GenBank (Hadiprata et al, 2015). Nilai Query cover tertinggi hasil BLAST terhadap sampel MS3 dan MS4 adalah sebesar 99%. Query cover adalah nilai yang menjelaskan seberapa banyak sekuen query yang tertutup oleh sekuen target. Jika sekuen target dalam database membentang seluruh sekuen yang ditarget, lalu nilai query cover adalah 100%. Hal ini menunjukan seberapa kuat hubungan antar sekuen (Fassler et al., 2011).
Setelah dilakukan BLAST kemudian sampel dianalisis jarak genetiknya. Hasil analissi jarak genetik ditunjukan pada Tabel 4. Jarak gentik terkecil sampel MS3 dan MS4 terhadap sampel GenBank adalah sebesar 0,60% dan 1% terhadap sampel U1 Macrobrachium sintangense Tukad Panti dari Bali, Indonesia penelitian sebelumnya. Jarak genetik terkecil kedua adalah antara sampel MS3 dan MS4 terhadap sampel sampel JQ362451.1 M. sintangense dan JQ362450.1 M. sintangense dari GenBank yaitu sebesar 1,40% dan 1,8%. Jarak genetik adalah tingkat perbedaan gen (genom) di antara spesies atau antara populasi dalam satu spesies, berupa proporsi situs nukleotida yang memiliki perbedaan antara dua sekuen yang dibandingkan (Nei, 1987). Suatu individu dikatakan mengalami spesiasi apabila memiliki jarak genetik di atas 3% (Jose et al., 2015). Berdasarkan nilai jarak genetik tersebut maka sampel MS3 maupun MS4 merupakan satu spesies dengan M. sintangense sampel M. sintangense dari Tukad Panti, Bali, Indonesia dan M. sintangense dari Thailand. Nilai jarak genetik didukung dengan pohon filogenetik yang terbentuk seperti pada gambar 2. Pohon filogenetik ini akan menunjukan hubungan kekerabatan yang terjadi.
Berdasarkan Tabel 5 dan 6 diketahui bahwa dari sampel MS3 dan MS4 yang diteliti dibandingkan dengan sampel JQ362451.1 M. sintangense dari GenBank memiliki variasi
genetik. Berdasarkan analisis variasi genetik tersebut menunjukkan bahwa dari 517 bp seluruh sekuens gen mitokondria 16S sampel terdapat 11 variable site dan 2 parsimony. Berdasarkan Tabel 5 diketahui bahwa nilai haplotype diversity (Hd) yang diperoleh sebesar 1,00 ± 0,177. Keragaman haplotype (Haplotype diversity/Hd) menunjukkan banyaknya variasi haplotype yang ada didalam suatu populasi yang menunjukkan dua alel sampel acak yang berbeda (Nei dan Li, 1979). Nilai haplotype diversity sebesar 1 menunjukkan bahwa keseluruhan sampel yang diuji adalah haplotype yang berbeda, seperti yang ditunjukan jumlah haplotype yaitu 4 (Tabel 6). Berdasarkan Tabel 5 diketahui bahwa nilai nucleotide diversity (π) 0,01139 ± 0,00333. Nucleotide diversity adalah rata-rata variasi nukleotida dalam satu bagian sekuen yang sama antar individu (De Jong et al., 2011). Nilai nucleotide diversity tersebut menunjukkan bahwa terdapat perbedaan nukleotida yaitu 11 situs. Perbedaan yang terjadi terdapat pada fragmen DNA ke‐ 1, 67, 88, 161, 258, 288, 296, 297, 451, 482, 517 seperti yang ditunjukan pada tabel situs polimorfik (Tabel 7).
Situs polimorfik menunjukkan letak terjadinya mutasi basa nitrogen pada sekuen sehingga menghasilkan variasi nukleotida. Berdasarkan hasil situs polimorfik pada Tabel 7 terdapat 9 transisi, dan 2 transversi. Indel dapat berupa mutasi gen yaitu insersi atau delesi (Chen et al., 2009). Transisi adalah perubahan basa purin menjadi basa purin lainnya atau pirimidin menjadi pirimidin lainnya. Transisi terjadi pada urutan basepair ke‐ 1, 67, 161, 258, 288, 296. 297, 482, 517 (Tabel 6). Transversi adalah perubahan basa purin menjadi pirimidin atau pirimidin menjadi purin (Lyons dan Lauring, 2017). Transversi terjadi pada urutan basepair ke‐88 dan 451 (Tabel 7).
SIMPULAN
Sampel yang ditemukan pada sungai Winongo, Tambakbayan, Code dan Opak di DIY adalah M. sintangense dengan karakter morfologis berupa rostrum sedikit melengkung ke
atas atau seperti pisau, post antennular carapace yang membulat, preanal carinal tidak ada, terdapat pubescene pada setengah proksimal hingga dua per tiga capit, sementara karakter molekuler berupa nilai similaritas 97.30% dan 97.48% dengan spesies Macrobracium sintangense dari GenBank (JQ362450.1 dan JQ362451.1) saat dilakukan BLAST serta memiliki hubungan kekerabatan yang dekat terhadap M. sintangense dari Tukad Panti Bali, Indonesia yaitu dengan nilai jarak genetik 0,6% dan 1% sehingga sampel diketahui sebagai Macrobracium sintangense.
UCAPAN TERIMA KASIH
Penelitian ini terlaksana atas bantuan dari Fakultas Biologi UGM yang telah memberikan dana penelitian atas nama Rury Eprilurahman, S.Si., M.Sc. melalui program Penelitian Kolaborasi Dosen dan Mahasiswa Tahun 2019 dengan nomor kontrak UGM/BI/1703/M/02/05. Penulis menyampaikan terima kasih kepada: Kepala Laboratorium Genetika & Pemuliaan dan Sistematika Hewan Fakultas Biologi UGM yang telah memberikan ijin menggunakan fasilitas laboratorium; Tim Macrobrachium DIY 2019 dan Bapak Susilo Irwanjasmoro yang telah memberikan waktu dan tenaga selama proses pengerjaan penelitian ini dari awal sampai akhir baik di lapangan maupun di laboratorium.
KEPUSTAKAAN
Arif IA, Khan HA. 2009. Molecular markers for biodiversity analysis of wildlife animals: a brief review. Animal Biodiversity and Conservation 32(1): 9–17.
Chace FA, Bruce AJ. 1993. The Caridean Shrimps (Crustacea: Decapoda) of the Albatross Philippine Expedition, 1907-1910, Part 6: Superfamily Palaemonoidea.
Washington D. C.: Smithsonian Institution Press, pp. 20-22.
Chen J, Wu Y, Yang H, Bergelson J, Kreitman M, Tian D. 2009. Variation in the ratio of nucleotide substitution and indel rates across
genomes in mammals and bacteria. Molecular Biology and Evolution 26(7): 1523–1531
De Jong MA, Wahlberg N, van Eijk M, Brakefield PM, Zwaan BJ. 2011.
Mitochondrial DNA signature for rangewide populations of Bicyclus anynana suggests a rapid expansion from recent refugia. PloSONE 6(6): 1-5.
Fassler J, Cooper P. 2011. Blast Glossary. US.
National Center for Biotechnology Information.P : 8
Hadiprata NLMIYS, Putra IMBAPA, Mahardika IGNK, Wandia IN, Nindhia TS. 2015. Identifikasi Spesies Ikan Kerapu di Pasar Ikan Karangasem dan Kedonganan Bali Menggunakan DNA Mitokondria Gen 16s rRNA. Jurnal Veteriner 16(3): 423-431
Holthuis LB. 1950. Preliminary descriptions of twelve new spesies of palaemonid prawns from american waters (Crustacea Decapoda). Proceedings Koninklijke Nederlandse Akademie van Wetenschappen 53: 93-99.
Jose D, Nidhin B, Kumar KPA, Pradeep PJ, Harikrishnan M. 2015. A molecular approach towards the taxonomy of fresh water prawns Macrobrachium striatum and M. equidens (Decapoda, Palaemonidae) using
mitochondrial markers. Mitochondrial DNA. 1-9. DOI: 10.3109/19401736.2015. 1041114
Lyons DM, Lauring AS. 2017. Evidence for the selective basis of transitionto-transversion substitution bias in two RNA viruses. Molecular Biology and Evolution 34(12): 3205–3215.
Munasinghe DHN, Thushari GGN. 2010.
Analysis of morphological variation of four populations of Macrobrachium rosenbergii (de Man, 1879) (Crustacea: Decapoda) in Sri Lanka. Cey. J. Sci. (Bio. Sci.) 39 (1): 53-60.
Nei M, Li W. 1979. Mathematical Model for Studying Genetic Variation in Terms of Restriction Endonucleases. Proceedings of the National Academy of Sciences of the United States of America 76(10): 5269
5273.
Page TJ, Hughes JM. 2011. Neither molecular nor morphological data have all the answers; with an example from Macrobrachium
(Decapoda: Palaemonidae) from Australia. Zootaxa 2874: 65-68
Putranto TWC, Andriani R, Munawwaroh A, Irawan B, Soegianto A. 2014. Effect of cadmium on survival, osmoregulation and gill structure of the Sunda prawn, Macrobrachium sintangense (de Man), at different salinities. Marine and Freshwater Behaviour and Physiolog. 47(5): 349-360
Sharma C, Krishna G, Kumar AP, Nayak SK. 2014. Phylogeny of Macrobrachium spesies using mitochondrial rRNA ribosomal DNA. Cell Tissue Res. 14(3): 4525- 4529.
Wowor D, Cai Y, Ng PKL. 2004. Crustacea: Decapoda, Caridea. In: Yule, C.M., Sen, Y.H. (Eds.), Freshwater Invertebrates of the Malaysian Region. Academy of Sciences, Malaysia, Kuala Lumpur
Yang L, Tan Z, Wang D, Xue L, Guan M, Huang T, Li R. 2014. Spesies identification through mitochondrial rRNA genetic analysis. Scientific Reports 4: 4089. Doi:
10.1038/srep04089.
17
Discussion and feedback