Morphological and molecular characters of freshwater shrimp of Macrobrachium pilimanus species group in Opak, Winongo and Sempor Rivers, Yogyakarta
on
JURNAL BIOLOGI UDAYANA 25(1): 57-70
P ISSN: 1410-5292 E ISSN: 2599-2856
Karakter morfologis dan molekular udang air tawar anggota Macrobrachium pilimanus species group di Sungai Opak, Winongo dan Sempor, Daerah Istimewa Yogyakarta
Morphological and molecular characters of freshwater shrimp of Macrobrachium pilimanus species group in Opak, Winongo and Sempor Rivers, Yogyakarta
Rury Eprilurahman1, Malya Adzillina Silmi2,*, Lukman Hakim2, Trijoko1
1Laboratorium Sistematika Hewan, Fakultas Biologi, Universitas Gadjah Mada, Indonesia
Jl. Teknika Selatan, Sekip Utara, Sleman, Daerah Istimewa Yogyakarta, Indonesia 55281 2Fakultas Biologi, Universitas Gadjah Mada, Indonesia
Jl. Teknika Selatan, Sekip Utara, Sleman, Daerah Istimewa Yogyakarta, Indonesia 55281
*E-mail: malya.adzillina.silmi@mail.ugm.ac.id
Diterima 15 Oktober 2020 Disetujui 15 Pebruari 2021
INTISARI
Macrobrachium merupakan genus dari subfilum Crustacea dengan keanekaragaman tertinggi. Identifikasi morfologi dari genus Macrobrachium sebagian besar sulit dilakukan karena karakter untuk identifikasi tingkat spesies yang rumit, sedangkan banyak karakter umum untuk semua spesies anggota Macrobrachium. Sulitnya identifikasi morfologi juga disebabkan oleh pengaruh dominansi sosial pada jantan. Diperlukan pendekatan lain untuk mengidentifikasi Macrobrachium secara akurat, salah satunya dengan DNA barcoding menggunakan gen mitokondria 16S. Penelitian ini bertujuan untuk mengetahui karakter morfologi dan molekuler kelompok spesies Macrobrachium pilimanus berdasarkan gen mitokondria 16S dari sungai Opak, Winongo, dan Sempor di Daerah Istimewa Yogyakarta. Sampel udang diidentifikasi berdasarkan karakter morfologi, meristik, dan morfometri. Analisis molekuler menggunakan primer 16Sar sebagai primer forward dan 16Sbr sebagai primer reverse. Berdasarkan hasil identifikasi morfologi, sampel udang yang diperoleh hanya dapat teridentifikasi hingga tingkat genus Macrobrachium. Sampel udang juga dapat dimasukkan ke dalam kelompok spesies Macrobrachium pilimanus berdasarkan karakter umum kelompok spesies tersebut. Sampel udang pada penelitian ini memiliki kemiripan karakter paling tinggi dengan spesies M. pilimanus dan M. empulipke yang tersebar di Jawa. Berdasarkan analisis molekuler, sampel dari Sungai Winongo (WNM1) memiliki similaritas 87,63% dengan M. forcipatum (JQ362454.1) dan 87,02% dengan M. dienbienphuense (JQ390474.1), yang berarti merupakan spesies yang berbeda dari spesies di GenBank. Macrobrachium forcipatum dan M. dienbienphuense merupakan anggota kelompok spesies M. pilimanus. Kesimpulan dari penelitian ini adalah semua sampel merupakan anggota kelompok spesies M. pilimanus.
Kata kunci: 16S, Daerah Istimewa Yogyakarta, karakter morfologi, kelompok spesies Macrobrachium pilimanus
ABSTRACT
Macrobrachium is a genus of the Crustacea subphylum with the highest diversity. Identification of the morphology of the genus Macrobrachium is mostly challenging due to the characters for species level identification are complicated, whereas many characters are common to all Macrobrachium species. In addition, it is difficult to identify morphology because of the influence of social dominance in males.
Therefore, another approach is needed to identify Macrobrachium accurately, one of which is by barcoding DNA using the 16S mitochondrial gene. This study was aimed to determine the morphological and molecular characters of Macrobrachium pilimanus species group based on the 16S mitochondrial gene from the Opak, Winongo, and Sempor rivers, in Daerah Istimewa Yogyakarta. Furthermore, shrimp samples were identified based on morphological, meristic, and morphometric characters. Meanwhile, molecular analysis uses 16Sar as forward primer and 16Sbr as reverse primer. Based on the morphological identification results, the shrimp samples obtained could only be identified up to the level of the Macrobrachium genus. In addition, the shrimp samples can also be included in the Macrobrachium pilimanus species group based on the general characteristics of the species group. The shrimp samples in this study have highest similarities in character with the species M. pilimanus and M. empulipke distributed in Java. Based on molecular analysis, sample from Winongo River (WNM1) has a similarity of 87.63% with Macrobrachium forcipatum (JQ362454.1) and 87,02% with M. dienbienphuense (JQ390474.1), which means that it is a different species from the species in the GenBank. Macrobrachium forcipatum and M. dienbienphuense are members of M. pilimanus species group. The conclusion of this research is that all the samples are member of M. pilimanus species group.
Keywords: Macrobrachium pilimanus species group, Daerah Istimewa Yogyakarta, morphological characters, 16S
PENDAHULUAN
Macrobrachium merupakan genus dari subfilum Crustacea dengan keanekaragaman tertinggi. Terdapat sekitar 240 spesies yang telah terdeskripsi dengan lebih dari 100 spesies dari genus ini ditemukan di Asia Timur dan Tenggara (Wowor et al., 2004; Wowor et al., 2009). Macrobrachium pilimanus species group adalah suatu kelompok spesies yang memiliki beberapa anggota spesies di dalamnya yang didasarkan atas karakter morfologi umum yang sama. Anggota kelompok spesies M. pilimanus dicirikan dengan karakter morfologi berupa rostrum yang tidak melebihi scaphocerite, carpus pereopod kedua berbentuk cangkir pendek, dan adanya pubescence lebat pada chela pereopod kedua (Ng, 1995).
Seiring berjalannya waktu dan banyaknya penelitian baru yang dilakukan, saat ini terdapat 16 spesies yang termasuk ke dalam anggota kelompok spesies M. pilimanus. Spesies-spesies tersebut adalah M. pilimanus (De Man, 1879), M. leptodactylus (De Man, 1892), M. hirsutimanus (Tiwari, 1952), M. dienbienphuense Dang and Nguyen, 1972, M. eriocheirum Dai, 1984, M. ahkowi Chong and Khoo, 1987, M. gua Chong, 1989, M. forcipatum Ng, 1995, M. platycheles Ou and Yeo, 1995, M. poeti Holthuis 1984, M. amplimanus Cai and Dai, 1999, M. sirindhorn
Naiyanetr, 2001, M. pilosum Cai and Dai, 1999, M. kelianense Wowor and Short, 2007, M. empulipke Wowor, 2010, dan M. spelaeus Cai and Vidthayanon, 2016 (Cai & Vidthayanon, 2016).
Identifikasi morfologis untuk genus Macrobrachium tergolong sulit, karena sangat bergantung pada perbedaan karakter morfologis yang unik untuk setiap spesies. Karakter yang digunakan untuk identifikasi berjumlah terbatas, sedangkan banyak karakter umum untuk semua spesies Macrobrachium, maka identifikasi morfologis genus ini cukup sulit dilakukan (Holthuis, 1952). Faktor lain yang menyebabkan sulitnya identifikasi pada genus ini adalah adanya pengaruh dominansi sosial pada morfologi jantan (Short, 2004). Anggota M. pilimanus species group memiliki variabilitas karakter yang luar biasa, sehingga menjadikan taksonomi kelompok spesies ini sangat kompleks. Kemungkinan hanya studi secara ekstensif dari berbagai lokasi di seluruh wilayah jelajahnya dan studi spesimen hidup di habitat aslinya, yang dapat memecahkan masalah ini (Fransen et al., 2010).
Sulitnya karakterisasi genus Macrobrachium secara morfologi, menuntut dilakukannya metode identifikasi lain yang lebih akurat, yaitu secara molekuler. Penggunaan DNA mitokondria dalam analisis filogenetik telah banyak dilakukan. Genom mitokondria diturunkan secara maternal
melalui sitoplasma telur tanpa rekombinasi. Genom ini dianggap memiliki kecepatan evolusioner yang lebih tinggi dari genom nukleus dan dianggap cocok untuk studi sistematika dan genetika molekuler (Chen et al., 2009; Yang et al., 2014). Gen 16S telah menjadi salah satu segmen genom mitokondria yang digunakan dalam analisis filogenetik. Penelitian analisis filogenetik dengan gen 16S pada Crustacea telah banyak dilakukan, salah satunya pada udang air tawar Macrobrachium Bate, 1868 (Murphy & Austin, 2002). Berdasarkan hal tersebut, maka dilakukan penelitian mengenai karakterisasi morfologis dan molekuler dengan gen 16S pada udang air tawar anggota kelompok spesies M. pilimanus di perairan sungai di Yogyakarta.
MATERI DAN METODE
Pengambilan sampel udang Macrobrachium dilakukan di beberapa sungai di Daerah Istimewa Yogyakarta, yaitu Sungai Opak, Winongo, dan Sempor. Pengambilan sampel di Sungai Opak dilaksanakan di dua titik sampling pada tahun 2013. Pengambilan sampel di Sungai Winongo dilaksanakan di satu titik sampling pada tanggal 8-11 Agustus 2015. Pengambilan sampel di Sungai Sempor dilaksanakan di satu titik sampling pula pada tanggal 27 Februari 2020. Data masing-masing lokasi pengambilan sampel ditunjukkan pada Tabel 1. Pengambilan data morfologis dilakukan di Laboratorium Sistematika Hewan, sedangkan data molekuler dilakukan di Laboratorium Genetika dan Pemuliaan, Fakultas Biologi UGM. Penelitian tersebut dilakukan pada bulan Maret-Agustus 2020.
Tempat dan waktu penelitian
|
Tabel 1. Data lokasi sampling | |||
|
Opak |
Winongo |
Sempor | |
|
Titik sampling |
Hulu TS 2 S 07o39’41.51” E 110o27’16.14” Hulu TS 3 S 07o41’20.28” E 110o27’56.33” |
Hulu TS 1 Titik Awal: S 07o39'48.3" E 110o22'51.6" , Titik Akhir: S 07o40'01.1" E 110o22'41.1" |
Hulu TS 1 S 07o38’02”E110o23’15” |
|
Lokasi |
Dusun Panggung, Desa Argomulyo, Kecamatan Cangkringan, Kabupaten Sleman (TS 2) Dusun Tambakan, Desa Sindumartani, Kecamatan Ngemplak, Kabupaten Sleman (TS3) |
Dukuh Gabugan, Desa Donokerto, Kecamatan Turi, Kabupaten Sleman |
Dukuh Somohitan, Desa Girikerto, Kecamatan Turi, Kabupaten Sleman |
|
Ketinggian (m dpl) |
413-440 (TS 2) 282-296 (TS 3) |
389-407 |
513 |
|
Arus |
Deras (TS 2 dan TS 3) |
Deras |
Deras |
|
Substrat |
Batuan dan kerikil (TS 2) Batuan, kerikil, dan pasir (TS 3) |
Batuan |
Pasir dan batuan |
Bahan dan alat
Bahan yang digunakan dalam penelitian ini adalah sampel udang yang telah dikoleksi dan
ethanol 96% yang digunakan untuk preservasi sampel. Bahan yang digunakan dalam identifikasi molekular antara lain daging dari abdomen
spesimen, ethanol 70%, 180 μL buffer ATL, QIAGEN DNeasy Blood & Tissue Kits, Tris-EDTA (TE), DNA ladder, agarose, pewarna Florosafe DNA Stain, akuades, akuabides, Bioline MyTaq Red Mix, primer 16 Sar (5’CGCCTGTTTATCAAAAACAT-3’) sebagai primer forward dan 16 Sbr
(5’CCGGTCTGAACTCAGATCACGT-3’) sebagai primer reverse.
Alat yang digunakan untuk pengambilan data morfologis antara lain jaring untuk sampling spesimen udang, mikroskop digital untuk mengamati bagian tubuh sampel yang berukuran kecil, caliper untuk mengukur morfometri spesimen, dan pinset untuk mengambil dan memegang sampel.Alat yang digunakan dalam identifikasi molekuler antara lain gunting, microcentrifuge tube (1,5-2 mL), DNeasy Mini spin, collection tube,mikropipet (Biohi, Germany) ukuran 0.5-10 μL, 10-100 μL, 1001000 μL, freezer, microwave (LG, Korea Selatan), pipet tip, vortex mixer (Germany, Taiwan), electrophoresis chamber (GE-100, China), centrifuge (GyroZen, Korea Selatan), timbangan digital (Ohaus Scout Pro, USA), UV lamp transilluminator dengan Gel-Doc (Daihan, Korea), dan thermocycler (Open PCR).
Metode
Karakter morfologis yang diamati pada penelitian ini meliputi karakter morfologi, meristik, dan morfometri. Karakter morfologis yang digunakan untuk identifikasi berupa bentuk, ada tidaknya struktur khusus, jumlah suatu struktur tubuh, dan ukuran suatu bagian tubuh. Karakter yang digunakan ini berdasarkan referensi dari jurnal Chace & Bruce, 1993; Ou & Yeo, 1995; Wowor & Choy, 2001; dan Wowor et al., 2004.
Pengambilan data molekuler dibagi menjadi beberapa tahap. Pada tahap persiapan, dilakukan sterilisasi terhadap alat yang digunakan. Tahap isolasi DNA kemudian dilakukan dengan mengikuti protokol dari QIAGEN DNEasy Blood & Tissues Kits. Isolat DNA diamplifikasi menggunakan primer 16Sar (forward) dan 16Sbr (reverse) dengan Bioline MyTaqTM HS Mix. PCR
dilakukan menggunakanthermocycler dengan siklus amplifikasi DNA, yaitu predenaturation 95° selama 1 menit, denaturation 95° selama 15 detik, annealing 50° selama 30 detik, extention 72° selama 30 detik, final extention 72° selama 5 menit, dan hold 4°. Elektroforesis dilakukan menggunakan agarose 1%, tegangan 100 V, dengan buffer TAE 1x. Setelah proses running selesai, gel agarose diangkat dan didokumentasikan dengan kamera pada UV illuminator. Tahapansequencingdilakukan dengan mengirimkan sampel hasil amplifikasi ke1st Base.
Analisis data
Karakter morfologis dianalisis secara deskriptif dengan mengamati ciri morfologi, meristik, dan morfometri dari udang Macrobrachium. Karakter-karakter yang diamati tersebut kemudian dibandingkan dengan karakter yang dimiliki oleh anggota kelompok spesies M. pilimanus. Perbandingan karakter tersebut kemudian lebih difokuskan antara sampel yang diteliti dengan spesies anggota M. pilimanus yang memiliki distribusi di Pulau Jawa, untuk mengetahui kemiripan karakter di antara keduanya.
Analisis karakter molekuler dibagi menjadi beberapa tahap. Pertama, pengolahan hasil sequencing sampel dengan program Gene Studio. Analisis similaritas kemudian dilakukan dengan menggunakan Basic Local Alignment Search Tool (BLAST) yang dapat diakses melalui website NCBI (https://blast.ncbi.nlm.nih.gov). Konversi data GenBank ke dalam bentuk FASTA dilakukan dengan menggunakan program MESQUITE. Alignmentdilakukan denganmenggunakan program MEGA7. Program MEGA7 juga dapat digunakan untuk analisis jarak genetik dengan Bootstrap 1000 dan Kimura 2 parameter model, serta rekonstruksi pohon filogenetik dengan model yang sama dan metode Neighbor-Joining. Analisis variasi genetik dilakukan dengan menggunakan program DnaSP v.5.10.01 dan NoteTab Light.
HASIL
Karakterisasi Morfologis
Pada penelitian ini diperoleh 11 sampel udang yang terdiri dari 7 sampel dari sungai Opak, 2 sampel dari sungai Winongo, dan 2 sampel dari sungai Sempor. Sampel yang diperoleh di sungai Opak menunjukkan jumlah tertinggi karena dilakukan di dua titik sampling, sedangkan sungai Winongo dan Sempor hanya dilakukan di satu titik sampling. Berdasarkan hasil identifikasi morfologis yang meliputi karakter morfologi,
meristik, dan morfometri dari 11 sampel udang tersebut, semuanya hanya dapat diidentifikasi hingga tingkat genus yaitu Macrobrachium sp. Sampel udang tersebut juga dapat dimasukkan ke dalam satu kelompok spesies luas yaitu Macrobrachium pilimanus species group (Tabel 2). Pada Tabel 2 juga menunjukkan bahwa sampel OP5 dan OP6 berjenis kelamin betina, sedangkan sampel OP-OP4, WNM1, SM, dan SM1 masih tergolong juvenile. Sampel WNM tidak dapat diidentifikasi karena pleopod kedua mengalami kerusakan.
Tabel 2. Hasil identifikasi sampel udang dalam penelitian
|
No. |
Titik Sampling |
Singkatan |
Jenis Kelamin |
Spesies |
Species Group |
|
1. |
Hulu TS 2 Opak |
OP |
Juvenile |
Macrobrachium sp. |
Macrobrachium pilimanus |
|
2. |
Hulu TS 2 Opak |
OP1 |
Juvenile |
Macrobrachium sp. |
Macrobrachium pilimanus |
|
3. |
Hulu TS 2 Opak |
OP2 |
Juvenile |
Macrobrachium sp. |
Macrobrachium pilimanus |
|
4. |
Hulu TS 2 Opak |
OP3 |
Juvenile |
Macrobrachium sp. |
Macrobrachium pilimanus |
|
5. |
Hulu TS 2 Opak |
OP4 |
Juvenile |
Macrobrachium sp. |
Macrobrachium pilimanus |
|
6. |
Hulu TS 2 Opak |
OP5 |
Betina |
Macrobrachium sp. |
Macrobrachium pilimanus |
|
7. |
Hulu TS 3 Opak |
OP6 |
Betina |
Macrobrachium sp. |
Macrobrachium pilimanus |
|
8. |
Hulu TS 1 Winongo |
WNM |
- |
Macrobrachium sp. |
Macrobrachium pilimanus |
|
9. |
Hulu TS 1 Winongo |
WNM1 |
Juvenile |
Macrobrachium sp. |
Macrobrachium pilimanus |
|
10. |
Hulu TS1 Sempor |
SM |
Juvenile |
Macrobrachium sp. |
Macrobrachium pilimanus |
|
11. |
Hulu TS1 Sempor |
SM1 |
Juvenile |
Macrobrachium sp. |
Macrobrachium pilimanus |
|
Tabel 3. Karakter meristik dan morfometri sampel udang OP-SM1 | ||
|
No. |
Karakter |
Keterangan |
|
1. |
Rumus rostrum |
(4-5)+(6-8)/2-3 |
|
2. |
Jumlah gigi chela pereopod kedua |
8-13 |
|
3. |
Chela: carpus pereopod pertama |
0,64-0,75 |
|
4. |
Panjang jari: palma pereopod pertama |
0,8-1,2 |
|
5. |
Panjang jari: palma pereopod kedua |
0,68-0,93 |
|
6. |
ch (chela height): pl (propodus length) |
0,22-0,31 |
|
7. |
cw (chela width): pl (propodus length) |
0,17-0,23 |
|
8. |
dl (dactylus length): pl (propodus length) |
0,44-0,59 |
|
9. |
Chela: carpus pereopod mayor kedua |
3,1-4,5 |
|
10. |
Palma: carpus pereopod mayor kedua |
1,86-2,7 |
|
11. |
Carpus: merus pereopod mayor kedua |
0,5-0,67 |
|
12. |
Panjang: lebar carpus pereopod kedua |
1,33-1,73 |
|
13. |
Propodus: dactylus pereopod ketiga |
2,3-2,96 |
|
14. |
Panjang rostrum: panjang karapas |
0,35-0,45 |
Dalam penelitian ini, karakter meristik yang diamati adalah jumlah gigi rostum dan jumlah gigi chela pereopod kedua. Karakter morfometri yang diamati merupakan rasio atau perbandingan bagian tubuh satu dengan bagian tubuh lainnya. Hal ini dilakukan karena setiap sampel yang diamati memiliki umur dan perkembangan bagian tubuh yang berbeda-beda, sehingga perhitungan rasio tersebut diharapkan dapat menghasilkan nilai yang relatif lebih stabil. Tabel 3 menunjukkan hasil pengamatan karakter meristik dan morfometri pada sampel udang Macrobrachium sp.
Karakterisasi Molekular
Berdasarkan hasil analisis molekular dengan gen mitokondria 16S, diperoleh beberapa hasil berupa amplifikasi DNA, analisis similaritas dengan BLAST, jarak genetik, rekonstruksi pohon filogenetik, dan variasi genetik. Berdasarkan hasil amplifikasi, diperoleh panjang fragmen gen mitokondria 16S pada udang Macrobrachium dengan kode sampel WNM1 adalah 493 bp.
Sampel WNM1 kemudian dianalisis similaritas menggunakan program Nucleotide BLAST. Berdasarkan analisis similaritas, didapatkan hasil bahwa sampel WNM1 memiliki similaritas tertinggi dengan spesies M. forcipatum sebesar 87,63%, kemudian diikuti dengan spesies M.
dienbienphuense sebesar 87,02%. Nilai query cover dengan kedua spesies tersebut sebesar 99% (Tabel 4). Analisis dilanjutkan dengan program MEGA untuk mengetahui jarak genetik dan merekonstruksi pohon filogenetik. Berdasarkan Tabel 5, didapatkan hasil bahwa sampel WNM1 memiliki jarak genetik sebesar 13% dengan spesies M. forcipatum dan 14% dengan M. dienbienphuense dari database GenBank. Hasil rekonstruksi pohon filogenetik, menunjukkan bahwa sampel WNM1 memiliki hubungan paling dekat atau ancestor terdekat dengan spesies M. forcipatum dan M. dienbienphuense dengan nilai bootstrap sebesar 69% (Gambar 2).
M WNMl
2000 bp __ IOOObp __
500 bp _
250 bp IOObp
Gambar 1. Elektroforegram hasil PCR dari ekstraksi DNA sampel udang WNM1 berdasarkan gen mitokondria 16S (M= Marker, WNM1= udang Macrobrachium Sungai Winongo)
Tabel 4. Persentase (%) similaritas antara sampel WNM1 dengan sampel dari database GenBank
|
No. |
Sampel NCBI |
Spesimen | |
|
WNM1 | |||
|
1. |
JQ362454.1 Macrobrachium forcipatum |
Identity Query cover |
87,63% 99% |
|
2. |
JQ390474.1 Macrobrachium dienbienphuense |
Identity Query cover |
87,02% 99% |
Tabel 5. Jarak genetik (%) sampel udang WNM1 dengan sampel udang dari GenBank
|
WNM1 |
M. forcipatum JQ362454.1 |
M. dienbienphuense JQ390474.1 | |
|
WNM1 |
2** |
2** | |
|
M. forcipatum JQ362454.1 |
13* |
1** | |
|
M. dienbienphuense JQ390474.1 |
14* |
2* |
Keterangan : *) jarak genetik; **) standard error
82 i—JQ362454.1 Macrobrachiumforcipatum
θθ ∣—JQ390474.1 Macrobrachiumdienbienphuense
39 ------------------------------WNM1
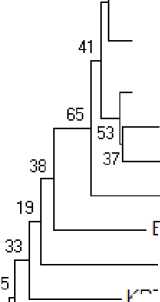
JQ390476.1 Macrobrachium hirsutimanus
AB646951.1 Macrobrachium amplimanus
DQ194954.1 Macrobrachium platycheles
EU493145.1 Macrobrachium esculentum
HM352463 1 Macrobrachium Crenulatum
FM986633.1 Macrobrachium pilimanus
100 ∣-GQ487497.1 Macrobrachium pilimanus
AB646964.1 Macrobrachiumeriocheirum
KP763702.1 Macrobrachium idella
EU493141.1 Macrobrachium japonicum
711----JF310718.1 Macrobrachium horstii
∣----EU493146.1 Macrobrachium jaroense
EU493142.1 Macrobrachium gracilirostre — FM986636.1 Macrobrachium placidum
D□681291 1 Macrobrachiumtolmerum
MK189912.1 Caridina malanda
0.050
Gambar 2. Rekonstruksi pohon filogenetik Neighbour-Joining 18 sekuen gen mitokondira 16S anggota
Macrobrachium menggunakan model Kimura 2-Parameter pada program MEGA 7
Analisis variasi genetik dilakukan dengan menggunakan program DNAsp dan NoteTab Light. Analisis ini digunakan untuk mengetahui variable sites, parsimony sites, nucleotide diversity, dan haplotype diversity. Berdasarkan hasil analisis variasi genetik (Tabel 6) antara sampel WNM1 dengan sampel M. forcipatum dan M. dienbienphuense dari GenBank, didapatkan tiga haplotipe dari tiga sekuen yang dianalisis. Berdasarkan tabel tersebut, diketahui juga bahwa
dari 494 bp sekuen gen mitokondria 16S yang diteliti, terdapat 67 variable site dan 0 parsimony site. Nilai keragaman haplotipe (Haplotype Diveristy) diperoleh sebesar 1,000± 0,272. Nilai keragaman nukleotida (Nucleotide Diversity) dalam penelitian ini didapatkan sebesar 0,09079± 0,03614. Pada Tabel 7 menunjukkan situs polimorfik yang terdiri dari 2 indel, 39 transisi, dan 28 transversi.
Tabel 6. Variasi genetik antara sampel udang Macrobrachium sp. WNM1 dengan M. forcipatum dan M. dienbienphuense berdasarkan gen 16S mitokondria
|
Spesies |
Bp |
Jumlah Individu |
Jumlah Haplotipe |
Variable Sites |
Parsimony Sites |
Haplotype Diversity (Hd) |
Nucleotide Diversity (π) |
|
Macrobrachium sp. (WNM1) |
494 |
3 |
3 |
67 |
0 |
1,000± 0,272 |
0,09079± 0,03614 |
Tabel 7. Hasil analisis situs polimorfik sampel WNM1 dari penelitian dan sekuen M. forcipatum dari GenBank
|
Sampel |
Haplotipe |
Situs Polimorfik | ||||||||||||||||||||||||||||||||||
|
1 |
1 |
1 |
1 |
1 |
2 |
2 |
2 |
2 |
2 |
2 |
2 |
2 |
2 |
2 |
3 |
3 |
3 |
3 |
3 |
3 |
3 |
3 |
3 |
3 |
3 |
3 |
3 |
3 |
3 |
3 |
3 | |||||
|
1 |
8 |
1 |
1 |
2 |
7 |
7 |
0 |
0 |
1 |
1 |
2 |
3 |
3 |
5 |
5 |
5 |
1 |
2 |
2 |
3 |
4 |
4 |
4 |
4 |
5 |
5 |
5 |
6 |
7 |
7 |
7 |
8 |
9 | |||
|
2 |
7 |
6 |
4 |
8 |
6 |
1 |
2 |
7 |
8 |
2 |
8 |
8 |
0 |
3 |
4 |
8 |
9 |
8 |
0 |
7 |
7 |
0 |
2 |
6 |
9 |
2 |
5 |
8 |
1 |
0 |
2 |
7 |
0 |
1 | ||
|
JQ362454.1 |
HT1 |
A |
C |
A |
A |
T |
T |
G |
C |
T |
G |
T |
T |
T |
A |
G |
A |
C |
A |
G |
T |
G |
A |
T |
T |
T |
G |
G |
T |
G |
- |
G |
T |
G |
T |
G |
|
JQ390474.1 |
HT2 |
. |
. |
G |
. |
. |
C |
A |
T |
. |
. |
G |
. |
. |
. |
A |
G |
. |
A |
A | ||||||||||||||||
|
WNM1 |
HT3 |
T |
- |
. |
G |
G |
C |
. |
T |
C |
A |
. |
C |
C |
G |
A |
. |
T |
G |
. |
C |
. |
G |
A |
G |
A |
A |
A |
A |
A |
T |
A |
A |
C |
G |
A |
|
Sampel |
Haplotipe |
Situs Polimorfik | ||||||||||||||||||||||||||||||||||
|
3 |
3 |
3 |
4 |
4 |
4 |
4 |
4 |
4 |
4 |
4 |
4 |
4 |
4 |
4 |
4 |
4 |
4 |
4 |
4 |
4 |
4 |
4 |
4 |
4 |
4 |
4 |
4 |
4 |
4 |
4 |
4 |
4 |
4 | |||
|
9 |
9 |
9 |
0 |
0 |
0 |
1 |
1 |
1 |
1 |
1 |
2 |
2 |
2 |
3 |
3 |
4 |
4 |
4 |
4 |
5 |
5 |
5 |
5 |
6 |
6 |
6 |
6 |
6 |
7 |
7 |
7 |
7 |
8 | |||
|
3 |
5 |
7 |
0 |
4 |
9 |
1 |
2 |
4 |
7 |
9 |
3 |
5 |
7 |
1 |
9 |
2 |
5 |
6 |
7 |
1 |
3 |
5 |
9 |
1 |
3 |
5 |
6 |
8 |
2 |
4 |
7 |
9 |
3 | |||
|
JQ362454.1 |
HT1 |
G |
G |
T |
T |
G |
G |
G |
T |
G |
G |
G |
T |
G |
T |
G |
G |
T |
G |
C |
T |
T |
T |
G |
C |
T |
G |
T |
A |
T |
T |
T |
T |
G |
C | |
|
JQ390474.1 |
HT2 |
. |
T |
T | ||||||||||||||||||||||||||||||||
|
WNM1 |
HT3 |
A |
T |
C |
A |
A |
A |
T |
A |
T |
C |
A |
C |
A |
G |
A |
T |
C |
C |
. |
A |
G |
A |
C |
T |
A |
C |
G |
G |
G |
C |
G |
C |
A |
T | |
Keterangan:

PEMBAHASAN
Karakterisasi Morfologis
Berdasarkan hasil pengamatan secara morfologis yang meliputi karakter morfologi, morfometri, dan meristik, seluruh sampel udang yang diperoleh dari Sungai Winongo, Opak, dan Sempor termasuk ke dalam Infraordo Caridea. Anggota infraordo ini dicirikan dengan pleura segmen abdominal kedua berbentuk buah pear dan melebar menutupi segmen pertama dan ketiga. Pada tingkat familia, sampel udang tersebut dimasukkan ke dalam familia Palaemonidae, yang dicirikan dengan bentuk karapas yang silindris dengan rostrum menonjol yang terkompresi lateral dan memiliki gigi pada bagian dorsal dan ventral, pasangan pereopod pertama dan kedua memiliki pincer dengan pereopod pertama lebih pendek dan ramping dari pereopod kedua, tiga pereopod terakhir tanpa pincer, dan individu jantan memiliki appendix masculina dan appendix interna pada endopod pleopod kedua (Fischer & Bianchi, 1984; Chace & Bruce, 1993). Pada tingkat genus, seluruh sampel udang ini dikelompokkan ke dalam genus Macrobrachium karena memiliki karakter antara lain karapas dengan spina hepatica, tetapi tanpa spina branchiostegal. Dactylus tiga pereopod terakhir sederhana (non bifida), pereopod kedua panjang dan kuat, dan pleopod pertama individu jantan dicirikan dengan tidak adanya appendix interna pada bagian endopodnya (Fischer &
Bianchi, 1984; Chace & Bruce, 1993; Short, 2004).
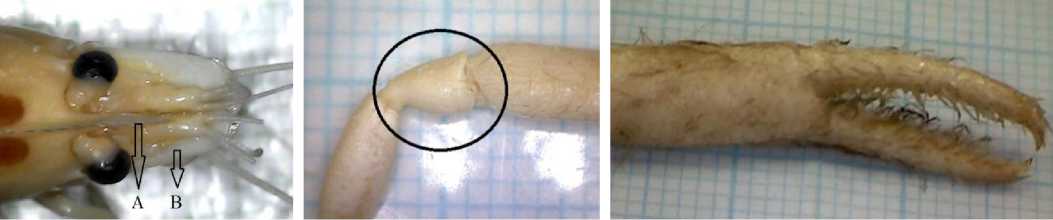
Identifikasi udang Macrobrachium hingga tingkat spesies memerlukan individu jantan dewasa. Dari 11 sampel udang yang diidentifikasi, dua di antaranya (OP5 dan OP6) berjenis kelamin betina karena tidak adanya appendix masculina pada endopod pleopod kedua, serta didukung dengan ditemukannya telur pada bagian abdomen. Sampel OP-OP4, WNM1, SM, dan SM1 masih dalam tahap juvenile karena appendix masculina tidak ada atau belum berkembang. Oleh karena itu, seluruh sampel udang yang diteliti tidak dapat diidentifikasi secara morfologi hingga tingkat spesies. Sampel udang tersebutdapat dimasukkan ke dalam satu kelompok spesies luas, yaitu kelompok spesies Macrobrachium pilimanus berdasarkan kesamaan karakter yang dimilikinya, antara lain memiliki rostrum yang tidak melebihi scaphocerite, carpus pereopod kedua berbentuk cangkir pendek, dan adanya pubescence lebat pada chela pereopod kedua (Gambar 3) (Ng, 1995).
Selain karakter-karakter yang dimiliki oleh semua anggota kelompok spesies M. pilimanus, diamati pula karakter morfologi lainnya. Seluruh sampel yang diteliti memiliki rostrum pendek yang tidak mencapai akhir segmen ketiga antennular peduncle danmemiliki epistome yang berbentuk trilobus dengan dua lekukan. Pada bagian telson, memiliki preanal carina pada inter-uropodal sclerite, terdapat mobile mesial spine
yang lebih pendek dari distolateral tooth, dan apeks posterior telson tidak melebihi spina posterolateral. Karakter meristik dan morfometri dari sampel udang dapat dilihat pada Tabel 3. Karakter morfologi, meristik, dan morfometri yang telah diamati dari sampel penelitian kemudian dibandingkan dengan seluruh spesies
anggota M. pilimanus species group, dan lebih difokuskan pada spesies yang terdistribusi di Pulau Jawa. Spesies anggota M. pilimanus species group yang terdistribusi di Pulau Jawa, antara lain M. pilimanus, M. empulipke, M. leptodactylus, dan M. poeti (Wowor et al., 2004; Cai et al., 2004; Wowor, 2010; Cai & Vidthayanon, 2016).
(a)
(b)
(c)
Gambar 3. Rostum (A) tidak melebihi scaphocerite (B) sampel WNM1 (a), Carpus pereopod kedua sampel WNM1 (b),
Pubescence chela pereopod kedua sampel WNM (c)
Sampel udang dalam penelitian ini memiliki banyak kesamaan dengan spesies M. pilimanus dan M. empulipke, antara lain memiliki rostrum pendek dan tidak mencapai akhir segmen ketiga antennular peduncle, memiliki preanal carina, dan memiliki mobile mesial spine yang lebih pendek dari distolateral tooth. (Cai et al., 2004; Wowor, 2010). Karakter meristik dari sampel penelitian, M. pilimanus, dan M. empulipke, yaitu masing-masing memiliki rumus rostrum (4-5)+(6-8)/2-3 vs (3-5)+(6-10)/1-3 vs (3-5)+(6-10)/ 2-3, serta memiliki gigi pada chela pereopod kedua sebanyak 8-13 vs 14-17 vs 12-15. Berdasarkan hal tersebut, M. pilimanus memiliki perbedaan karakter jumlah gigi pada chela pereopod kedua (Cai et al., 2004; Wowor, 2010).
Karakter morfometri antara sampel dengan M. pilimanus dan M. empulipke memiliki beberapa kesamaan dan perbedaan. Persamaan karakter morfometri antara sampel dengan M. pilimanus antara lain pada bagian pereopod pertama,yaitu rasio panjang chela terhadap carpus (0,64-0,75 vs 0,67), serta pada bagian pereopod kedua yaitu rasio jari terhadap palma (0,68-0,93 vs 0,83-1,12), rasio chela height terhadap propodus length (0,22-0,31 vs 0,17-0,31), rasio chela width terhadap propodus length (0,17-0,23 vs 0,15-0,2), rasio dactylus length terhadappropodus length
(0,44-0,59 vs 0,51-0,59), rasio palma terhadap carpus (1,86-2,7 vs 1,25 sampai lebih dari 2), dan rasio carpus terhadap merus (0,5-0,67 vs 0,50,67). Persamaan karakter ini memiliki arti yaitu beberapa karakter M. pilimanus masih masuk dalam rentang hasil pengukuran pada sampel. Perbedaan karakter antara sampel dengan M. pilimanus antara lain rasio chela terhadap carpus pereopod kedua sebesar 3,1-4,5 vs 5, rasio propodus terhadap dactylus pereopod kedua sebesar 2,3-2,96 vs 3,2, dan rasio rostrum terhadap karapas sebesar 0,35-0,45 vs 0,14-0,2 (Chace & Bruce, 1993; Ou & Yeo, 1995; Cai et al., 2004, Wowor, 2010).
Kesamaan karakter morfometri antara sampel dengan M. empulipke antara lain pada bagian pereopod pertama, yaitu rasio panjang chela terhadap carpus (0,64-0,75 vs 0,62-0,67) dan rasio panjang jari terhadap palma (0,8-1,2 vs jari sepanjang palma), serta pada bagian pereopod kedua yaitu rasio panjang chela terhadap carpus (3,1-4,5 vs 3,4-3,8) dan rasio panjang carpus terhadap merus (0,5-0,67 vs 0,62-0,73). Selain itu, rasio panjang rostrum terhadap karapas juga masih dalam rentang rasio pengukuran sampel (0,35-0,45 vs 0,35-0,47). Perbedaan antara sampel dengan M. empulipke yaitu pada rasio panjang jari terhadap palma pereopod kedua
sebesar 0,68-0,9 vs 1,4-2, rasio panjang palma terhadap carpus pereopod kedua sebesar 1,86-2,7 vs 1,59, dan rasio panjang carpus pereopod kedua terhadap lebarnya sebesar 1,33-1,73 vs 1,68-2,03 (Wowor, 2010).
Perbandingan antara sampel dengan M. leptodactylus dan M. poeti lebih banyak memiliki perbedaan daripada kesamaan karakter. Kesamaan karakter antara sampel dan M. leptodactylus yaitu memiliki rostrum pendek
yang tidak melebihi akhir segmen ketiga antennular peduncle dan bentuk epistome yang mirip (Gambar 4). Perbedaan antara sampel dengan spesies tersebut antara lain rumus gigi rostrum yaitu (4-5)+(6-8)/2-3 vs 7+7/2, gigi pada chela pereopod kedua sebanyak 8-13 vs 15, rasio carpus terhadap merus pereopod kedua sebesar 0,5-0,67 vs 0,78-0,9, dan rasio panjang carpus pereopod kedua terhadap lebarnya sebesar 1,331,73 vs 2,18-2,68 (Cai et al., 2004; Wowor, 2010).
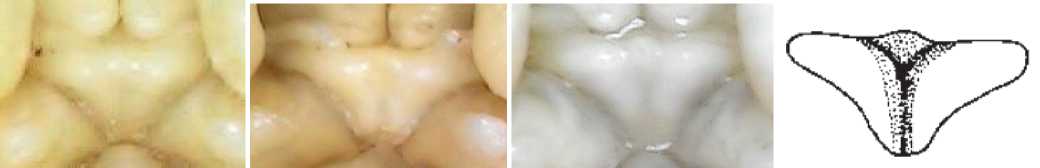
Gambar 4. Bentuk epistome sampel WNM1 (a), OP1 (b), SM (c) (Doc Pribadi, 2020), spesies M. leptodactylus (d) (Cai et al.,2004)
Sampel penelitian Macrobrachium sp. dibandingkan dengan spesies M. poeti, lebih banyak menunjukkan perbedaan. Spesies M. poeti hanya memiliki 1 gigi ventral pada rostrumnya, sedangkan dalam penelitian ini jumlah gigi rostrum ventral yang terhitung berkisar antara 23. Jumlah gigi pada cheliped pereopod kedua hanya beberapa, sedangkan pada sampel berkisar antara 8-13 buah. Karakter lain yang dimiliki oleh spesies M. poeti adalah memiliki merus pereopod kedua yang secara nyata lebih panjang dari palma, dan propodus pereopod ketiga tiga kali dactylus (Cai & Vidthayanon, 2016).
Sampel dalam penelitian ini juga memiliki beberapa kesamaan dengan spesies M. forcipatum, yaitu memiliki mobile mesial spine lebih pendek dari distolateral tooth, jumlah gigi rostrum ((4-5)+(6-8)/2-3 vs (3-5)+(6-10)/2-3), jumlah gigi pada chela pereopod kedua (8-13 vs 7-10), rasio jari dengan palma pereopod pertama (0,8-1,2 vs jari lebih pendek dari palma), dan rasio panjang propodus dengan dactylus pereopod ketiga (2,3-2,96 vs 2,6-3). Pada bagian pereopod kedua, terdapat kesamaan pada rasio panjang carpus terhadap merus (0,5-0,67 vs 0,5) dan rasio panjang carpus dengan lebarnya (1,33-1,73 vs 1,4) (Cai et al., 2004). Sampel WNM1 juga
memiliki beberapa kesamaan dengan spesies M. dienbienphuense, antara lain memiliki preanal carina, mobile mesial spine lebih pendek dari distolateral tooth, jumlah gigi rostrum ((4-5)+(6-8)/2-3 vs (3-5)+(5-9)/1-3), rasio jari terhadap palma pereopod pertama (0,8-1,2 vs jari sepanjang palma), rasio chela terhadap carpus pereopod kedua (3,1-4,5 vs 3,3), rasio palma terhadap carpus pereopod kedua (1,86-2,7 vs lebih dari 2), dan rasio propodus terhadap dactylus pereopod ketiga (2,3-2,96 vs 2,8).
Berdasarkan pengamatan morfologis, baik karakter morfologi, meristik, maupun morfometri spesies Macrobrachium sp. yang digunakan dalam penelitian ini menunjukkan adanya variasi karakter yang merujuk ke beberapa spesies anggota M. pilimanus species group. Perbandingan karakter sampel dengan M. pilimanus species group yang terdistribusi di Pulau Jawa, menunjukkan bahwa sampel penelitian memiliki kesamaan karakter yang lebih besar dengan spesies M. pilimanus dan M. empulipke. Hal ini diperkuat dengan kondisi habitat ditemukannya sampel udang tersebut, yaitu di daerah hulu sungai yang memiliki substrat berupa batu dan pasir dengan arus yang cukup deras. Menurut literatur, spesies M.
pilimanus dapat ditemukan di aliran sungai yang deras dengan substrat pasir dan batu dengan serasah daun di sepanjang tepian, tetapi tanpa vegetasi lebat (Ou & Yeo, 1995). SpesiesM. empulipke dapat ditemukan di bentangan sungai bagian tengah dan atas dengan campuran substrat bongkahan batu besar dan kecil, serta aliran air yang sedang hingga deras. Kadang-kadang juga mendiami badan air yang dalam (Wowor, 2010). Kondisi habitat antara sampel dengan M. pilimanus dan M. empulipke ini menunjukkan adanya kemiripan. Sampel udang dalam penelitian lebih banyak menunjukkan perbedaan dengan spesies M. poeti, salah satunya karena perbedaan karakter habitat antara keduanya. Macrobrachium poeti ditemukan di sungai di beberapa gua di Gunung Sewu (Cai & Vidthayanon, 2016).
Adanya variasi karakter pada sampel udang dalam penelitian ini diakibatkan karena sampel udang yang diamati belum mencapai tingkat jantan dewasa, sehingga karakter yang diamati bersifat kurang konsisten. Taksonomi dari kelompok spesies ini juga sangat rumit. Variabilitas spesies dalam kelompok spesies M. pilimanus juga sangat luar biasa. Kemungkinan hanya studi secara ekstensif dari berbagai lokasi di seluruh wilayah jelajahnya dan studi spesimen hidup di habitat aslinya, yang dapat memecahkan masalah ini (Fransen et al., 2010).
Karakterisasi Molekuler
Dalam analisis molekular, hanya satu sampel saja yang dapat dianalisis, yaitu sampel WNM1 yang merupakan sampel udang dari Sungai Winongo, DIY. Hal ini dikarenakan beberapa sampel lainnya tidak dapat diidentifikasi secara molekuler akibat kerusakan jaringan yang disebabkan preservasi pada suhu ruang. Berdasarkan hasil amplifikasi DNA, diperoleh panjang fragmen gen mitokondria 16S pada udang Macrobrachium sp.sampel WNM1 sebesar 493 bp (Gambar 1). Langkah selanjutnya adalah analisis similaritas dengan Nucleotide BLAST. Gen mitokondria 16S yang akan dianalisis di BLAST sebaiknya memiliki panjang 500 bp, atau terburuknya adalah sepanjang 400 bp (Yang et al.,
2014). Oleh karena itu, panjang fragmen gen 16S pada sampel memenuhi syarat untuk dilakukan analisis similaritas pada BLAST. Berdasarkan analisis similaritas sampel WNM1 (Tabel 4) diketahui bahwa sampel udang memiliki similaritas tertinggi dengan spesies M. forcipatum sebesar 87,63% dan diikuti dengan spesies M. dienbienphuense sebesat 87,02%. Menurut literatur, persentase similaritas gen 16S rRNA kurang dari 97% dianggap sebagai spesies baru (Pangastuti, 2006). Oleh karena itu, sampel WNM1 dianggap sebagai spesies baru yang belum terdaftar dalam database GenBank. Spesies M. forcipatum dan M. dienbienphuense merupakan anggota kelompok spesies M. pilimanus. Berdasarkan data tersebut dapat diketahui bahwa spesimen WNM1 memiliki kedekatan dengan anggota spesies M. pilimanus. Sampel WNM1 menunjukkan nilai query cover sebesar 99% dengan kedua spesies, yang menunjukkan bahwa 99% dari total urutan nukleotida sampel WNM1 dapat dibandingkan dengan sekuen M. forcipatum dan M. dienbienphuense.
Analisis jarak genetik dan rekonstruksi pohon filogenetik kemudian dilakukan dengan menggunakan program MEGA. Jarak genetik adalah tingkat perbedaan gen (genom) di antara spesies atau antara populasi dalam satu spesies, berupa proporsi situs nukleotida yang memiliki perbedaan antara dua sekuen yang dibandingkan (Nei, 1987). Jarak genetik yang kecil menunjukkan bahwa populasi tersebut memiliki banyak alel yang serupa, berhubungan dekat, dan memiliki nenek moyang yang sama (Nei & Roychoudhury, 1974). Berdasarkan Tabel 5, diketahui bahwa sampel WNM1 memiliki jarak genetik terdekat dengan spesies M. forcipatum yaitu sebesar 13%, dan diikuti dengan M. dienbienphuense sebesar 14%. Besarnya nilai jarak genetik ini menunjukkan sampel WNM1 memiliki perbedaan gen yang cukup besar dan hubungan kekerabatan yang kurang dekat dengan kedua spesies tersebut. Berdasarkan hasil rekonstruksi pohon filogenetik (Gambar 2), sampel WNM1 memiliki ancestor terdekat dengan spesies M. forcipatum dan M.
dienbienphuense dengan nilai bootstrap sebesar 69%. Kedekatan sampel WNM1 dengan M. forcipatum dan M. dienbienphuense ini didukung oleh jarak genetik yang paling dekat dibandingkan spesies lain, yaitu masing-masing sebesar 13% dan 14%. Pohon filogenetik ini juga menunjukkan bahwa sampel WNM1 membentuk takson monofiletik dengan spesies lain dari anggota genus Macrobrachium, yang artinya sampel tersebut memiliki nenek moyang yang sama yaitu genus Macrobrachium, familia Palaemonidae. Spesies Caridina malanda berada pada cabang batang out group karena berasal dari familia Atyidae.
Analisis variasi genetik interspesies, diperoleh hasil berupa variable sites, parsimony sites, nucleotide diversity, dan haplotype diversity. Berdasarkan Tabel 6, didapatkan tiga haplotipe dari tiga sekuen yang dianalisis. Haplotipe adalah sekelompok alel dalam suatu organime yang diturunkan bersama dari satu induk (Cox et al., 2016). Diketahui pulabahwa dari 494 bp sekuen gen mitokondria 16S yang diteliti, terdapat 67 variable site dan 0 parsimony site. Oleh karena itu, 67 situs nukleotida yang berbeda tersebut adalah singleton site. Nilai keragaman haplotipe (Haplotype Diveristy) diperoleh sebesar 1,000± 0,272. Haplotype diversity adalah frekuensi dan jumlah haplotipe antara individu. Nilai haplotype diversity dikatakan tinggi apabila nilai Hd > 0,5 (Grant dan Bowen, 1998). Nilai haplotipe sebesar 1 ini menunjukkan bahwa seluruh sampel yang dianalisis adalah haplotipe yang berbeda. Oleh karena itu, tiga sekuen sampel yang dianalisis ini memiliki tiga haplotipe yang berbeda.
Nilai keragaman nukleotida (Nucleotide Diversity) dalam penelitian ini didapatkan sebesar 0,09079± 0,03614. Nucleotide diversity (π) adalah jumlah rata-rata perbedaan nukleotida per situs antara dua sekuen DNA yang dipilih secara acak dalam populasi (Nei & Li, 1979). Nilai nucleotide diversity dikatakan rendah jika nilai π<0,005 atau 0,5% (Grant and Bowen, 1998). Oleh karena itu, keragaman nukleotida dalam penelitian dapat dikatakan cukup tinggi, yang artinya ada banyak situs nukleotida yang mengalami mutasi, yaitu pada 67 situs.
Berdasarkan Tabel 7, dari 67 situs polimorfik terdapat 2 indel, 39 transisi, dan 28 transversi. Indel adalah peristiwa evolusi spesifik yang dapat berupa penambahan (insersi) maupun penghapusan (delesi) pasangan nukleotida dalam suatu sekuens. Indel bertanggung jawab atas terbentuknya gap saat alignment sekuens (Cartwright, 2009). Transisi adalah mutasi DNA berupa pertukaran basa pirimidin dengan basa pirimidin lain, maupun pertukaran basa pirin dengan basa pirin lainnya. Transversi adalah mutasi DNA berupa pertukaran basa nukleotida dari purin ke pirimidin, dan sebaliknya (Guo et al., 2017).
SIMPULAN
Berdasarkan analisis morfologis, seluruh sampel udang dalam penelitian teridentifikasi sebagai Macrobrachium sp. dan dimasukkan ke dalam M. pilimanus species group. Berdasarkan analisis molekular, sampel WNM1 dengan M. forcipatum dan M. dienbienphuense memiliki similaritas masing-masing 87,63% dan 87,02%, serta jarak genetik masing-masing 13% dan 14%. Hasil analisis ini menunjukkan bahwa sampel yang diteliti berbeda dengan M. forcipatum dan M. dienbienphuense, tetapi masih masuk ke dalam anggota M. pilimanus species group.
UCAPAN TERIMA KASIH
Penelitian ini terlaksana atas bantuan dari Fakultas Biologi UGM yang telah memberikan dana penelitian atas nama Rury Eprilurahman, S.Si., M.Sc. melalui program Penelitian Kolaborasi Dosen dan Mahasiswa Tahun 2019 dengan nomor kontrak UGM/BI/1703/M/02/05. Penulis menyampaikan terima kasih kepada: Kepala Laboratorium Genetika & Pemuliaan dan Sistematika Hewan Fakultas Biologi UGM yang telah memberikan ijin menggunakan fasilitas laboratorium; Tim Macrobrachium DIY 2019 dan Bapak Susilo Irwanjasmoro yang telah memberikan waktu dan tenaga selama proses pengerjaan penelitian ini dari awal sampai akhir baik di lapangan maupun di laboratorium.
KEPUSTAKAAN
Cai Y, Vidthayanon C. 2016. Macrobrachium spelaeus, a new species of stygobitic freshwater prawn from Thailand (Decapoda: Palaemonidae). Raffles Bulletin of Zoology 64: 117-122
Cai Y, Naiyanetr P, Ng PKL. 2004. The
freshwater prawns of the genus Macrobrachium Bate, 1868, of Thailand (Crustacea: Decapoda: Palaemonidae).
Journal of Natural History 38(5): 581-649
Cartwright RA. 2009. Problems and Solutions for Estimating Indel Rates and Length Distributions. Molecular Biology and Evolution 26(2): 473-480
Chace FA, Bruce AJ. 1993. The Caridean shrimps (Crustacea: Decapoda) of the Albatross Philippine Expedition, 1907–1910, Part 6: Superfamily Palaemonoidea. Smithsonian Contributions to Zoology 543: 1–152
Chen RT, Tsai CF, Tzeng WN. 2009. 16S and 28S rDNA Sequences in Phylogenetic Analyses of Freshwater Prawns (Macrobrachium Bate, 1868) from Taiwan. Journal of Crustacean Biology 29(3): 400-412
Cox CB, Moore PD, Ladle R. 2016. Biogeography: An Ecological and
Evolutional Approach. Ninth Edition. John Wiley & Sons, Inc., Oxford. p. 34, 106.
Fischer W, Bianchi G (eds). 1984. FAO species identification sheets for fishery purposes: Western Indian Ocean; (Fishing Area 51). Food and Agricultural Organization of the United Nations. Rome.
Fransen C, de Grave S, Ng P. 2010. Studies on Malacostraca: Lipke Bijdeley Holthuis
Memorial Volume. Koninklijke Brill NV. Leiden, p. 716.
Grant WS, Bowen BW. 1998. Shallow Population Histories in Deep Evolutionary Lineages of Marine Fishes: Insights from Sardines and Anchovies and Lessons for Conservation. The American Genetic Association 89: 415426
Guo C, McDowell IC, Nodzenski M, Scholtens DM, Allen AS, Lowe WL, Reddy TE. 2017. Transversions have Larger Regulatory Effects than Transitions. BMC Genomics 18(394): 1-9
Holthuis LB. 1952. A general revision of the Palaemonidae (Crustacea, Decapoda,
Natantia) of the Americas. II. The subfamily Palaemoninae. Occasional Papers of the Allan Hancock Foundation 12: 1–396
Muphy NP, Austin CM. 2002. A Preliminary Study of 16S rRNA Sequence Variation in Australian Macrobrachium Shrimps (Palaemonidae: Decapoda) Reveals
Inconsistencies in Their Current Classification. Invertebrate Systematics 16: 697-701
Nei M. 1987. Molecular Evolutionary Genetics. Columbia University Press, New York, USA.
Nei M, Roychoudhury AK. 1974. Genic variation within and between the three major races of man, Caucasoids, Negroids, and Mongoloids. The American Journal of Human Genetics, 26: 421–443.
Nei M, Li W. 1979. Mathematical Model for Studying Genetic Variation in Terms of Restriction Endonucleases. Proceedings of the National Academy of Sciences of the United States of America, 76(10): 5269– 5273.
Ng PKL. 1995. Freshwater Decapod Crustaceans (Potamidae, Palaemonidae) of Temengor Forest Reserve, Hulu Perak, Malaysia. Malayan Nature Journal 48: 249-257
Ou ACT, Yeo DCJ. 1995. A New Species of Freshwater Prawn, Macrobrachium platycheles (Decapoda: Caridea:
Palaemonidae) from Singapore and Peninsular Malaysia. The Raffles Bulletin of Zoology 43(2): 299-308
Pangastuti A. 2006. Definisi Spesies Prokaryota Berdasarkan Urutan Basa Gen Penyandi 16S rRNA dan Gen Penyandi Protein. Biodiveristas 7(3): 292-296
Short JW. 2004. A revision of Australian river prawns, Macrobrachium (Crustacea: Decapoda: Palaemonidae). Hydrobiologia 525: 1-100
Wowor D, Choy SC. 2001. The Freshwater Prawns of The Genus Macrobrachium BATE, 1868 (Crustacea: Decapoda:
Palaemonidae) from Brunei Darussalam. The Raffles Bulletin of Zoology 49(2): 269-289
Wowor D, Cai Y, Ng PKL. 2004. Crustacea: Decapoda, Caridea. In: Yule, C.M., Sen, Y.H. (Eds.), Freshwater Invertebrates of the Malaysian Region. Academy of Sciences. Malaysia, Kuala Lumpur.
Wowor D, Muthu V, Meier R, Balke M, Cai Y, Ng PKL. 2009. Evolution of Life History Traits in Asian Freshwater Prawns of The Genus Macrobrachium (Crustacea:
Decapoda: Palaemonidae) based on
Multilocus Molecular Phylogenetic
Analysis. Molecular Phylogenetics and Evolution 52(2): 340-350
Wowor D. 2010. Macrobrachium empulipke, a New Freshwater Prawn Species (Decapoda, Palaemonidae) from Indonesia. Dalam: CRM 014 – Fransen et al., (eds), Lipke Bijdeley Holthuis Memorial. Leiden: Koninklijke Brill NV. pp. 715-726
Yang L, Tan Z, Wang D, Xue L, Guan M, Huang T, Li R. 2014. Species identification through mitochondrial rRNA genetic analysis. Scientific Reports 4(4089): 1-11
70
Discussion and feedback