Deteksi Escherichia coli O157 pada air minum di Kelurahan Sekaran Gunungpati Semarang
on
JURNAL BIOLOGI UDAYANA 24(2): 55-62
P ISSN: 1410-5292 E ISSN: 2599-2856
Deteksi Escherichia coli O157 pada air minum di Kelurahan Sekaran Gunungpati Semarang
Detection of Escherichia coli O157 in drinking waters at Sekaran Gunungpati Semarang
Devi Dwi Jayanti1*, R. Susanti1, Ari Yuniastuti1, I Wayan Suardana2
1Jurusan Biologi, Fakultas Matematika dan Ilmu Pengetahuan Alam Universitas Negeri Semarang, Kampus Sekaran Gunungpati Semarang, Kode Pos 50229
2Lab. Kesmavet Fakultas Kedokteran Hewan Universitas Udayana, Denpasar. Jl. PB. Sudirman Denpasar, Bali
*Email: devidwijayanti@gmail.com
Diterima 17 April 2020 Disetujui 20 Agustus 2020
INTISARI
Penelitian bertujuan untuk mendeteksi adanya bakteri Escherichia coli O157 pada air minum kemasan, air minum isi ulang, dan air sumur di Kelurahan Sekaran Gunungpati Semarang. Sampel yang diambil sebanyak 20 sampel yang terdiri atas 4 merk air minum kemasan, 8 sampel air minum isi ulang, dan 8 sampel air sumur. Penelitian diawali dengan tahap isolasi E. coli pada medium Eosin Methylen Blue Agar (EMBA), yang dilanjutkan ke medium Sorbitol MacConkey Agar (SMAC) untuk identifikasi E. coli O157 dilanjutkan uji lateks aglutinasi (OXOID) dan diakhiri dengan uji konfirmasi gen rfbE menggunakan teknik Polymerase Chain Reaction (PCR). Hasil penelitian menunjukkan 8 sampel yang positif E. coli pada medium SMAC menunjukkan positif E. coli O157 (colorless). Uji lateks aglutinasi juga menunjukkan 8 sampel positif E. coli O157 seperti kontrol ATCC 43894. E. coli ATCC 43894 dan 8 sampel E. coli dari berbagai air minum di Kelurahan Sekaran Gunungpati Semarang menunjukkan positif E. coli O157.
Kata kunci: Escherichia coli O157, rfbE, air minum kemasan, air minum isi ulang, air sumur
ABSTRACT
The aim of this study was to detect Escherichia coli O157 in drinking waters at Sekaran, Gunungpati Semarang. Twenty (20) water samples consisted of four bottled water, eight refilled water, and eight well water was collected. In this study the bacteria were isolated in Eosin Methylen Blue Agar (EMBA) medium, followed by screening for Escherichia coli O157 in a selective medium of Sorbitol MacConkey Agar (SMAC), confirmation of O157 was conducted by applying O157 latex agglutination test and Polymerase chain reaction on rfbE gene. The result showed that 8 samples were found to be positive containing E. coli O157 as indicated by colorless colonies on SMAC medium. O157 latex aglutination test showed that 8 samples were confirmed positive to contain E. coli O157, which was similar to the results shown by control ATCC 43894.
Keywords: Escherichia coli O157, rfbE, bottled water, refilled water, well water
PENDAHULUAN
Air merupakan materi yang sangat penting dalam kehidupan tumbuhan, hewan, dan manusia. Kebutuhan air bersih terutama air minum semakin
lama semakin meningkat seiring keperluan dan taraf kehidupan penduduk. Industri air minum kemasan berkembang sangat pesat di Indonesia, karena semakin banyak minat masyarakat
terhadap air minum kemasan. Hal itu mendorong pertumbuhan industri air minum kemasan buatan Indonesia yang ada di pasaran. Konsumen lebih memilih air minum kemasan karena dianggap memiliki kualitas dan mutu yang lebih baik daripada air minum isi ulang. Harga air minum kemasan relatif lebih tinggi dibandingkan air minum isi ulang, tetapi daya beli masyarakat pada air minum kemasan tetap tinggi. Hal ini terbukti dalam kurun waktu sepuluh tahun, produk air minum dalam kemasan buatan Indonesia berkembang pesat (Waluyo, 2009).
Air minum isi ulang saat ini juga berkembang pesat, karena kebutuhan air minum instan dibutuhkan untuk rumah tangga dan gerai dengan harga yang terjangkau. Disisi lain harga air minum isi ulang ini lebih murah, karena biaya lebih efisien (Kandou, 2009). Air minum isi ulang relatif murah, efisien, mudah diperoleh sehingga banyak masyarakat yang lebih memilih air minum isi ulang.
Produk air minum kemasan dan isi ulang di Indonesia berkembang pesat sehingga menimbulkan permasalahan tentang mutu dan kualitas air minum yang aman untuk dikonsumsi. Hasil penelitian menyebutkan bahwa 8,33% sampel air minum kemasan dan 25% air minum isi ulang di Makassar terkontaminasi Escherichia coli O157 (Kandou, 2009). Di Kecamatan Bungus Padang menunjukkan bahwa 55,6% depo air minum menghasilkan air minum dengan kualitas yang tidak memenuhi persyaratan mikrobiologi yang ditetapkan pemerintah (Wandrivel et al., 2012).
Air yang layak diminum harus memenuhi tiga persyaratan kelayakan dari segi fisik, kimia dan mikrobiologis. Syarat air minum harus aman diminum artinya bebas mikroba patogen dan zat berbahaya serta diterima dari segi warna, rasa, bau dan kekeruhannya (Soemirat, 2004). Standar kandungan bakteri koliform dalam air minum adalah 0 per 100 ml.
Escherichia coli (E. coli) pada produk air minum merupakan indikasi adanya kontaminasi (Odonkor & Ampofo, 2013). Salah satu bakteri koliform yang patogen adalah bakteri E. coli
O157; E. coli O157 adalah patogen yang menyebabkan penyakit serius pada manusia. E. coli O157 dapat beradaptasi dan bertahan di bawah kondisi rata-rata lingkungan termasuk perubahan suhu, pH rendah dan pengeringan.
Metode yang umum digunakan untuk pengujian air minum secara mikrobiologis adalah dengan menghitung jumlah bakteri koliform secara kuantitatif dan kualitatif. beberapa metode yang digunakan untuk mendeteksi E. coli O157 adalah medium agar Sorbitol MacConkay (SMAC) yang digunakan untuk isolasi E. coli O157, teknik PCR (Polymerase Chain Reaction) dan ELISA (Enzyme Linked Imunosorbent Assay) yang memanfaatkan antibodi monoklonal (4E8C12) spesifik untuk membran protein E. coli O157 (Odonkor & Ampofo, 2013).
Metode kultur adalah metode yang sederhana yang dapat digunakan untuk isolasi dan identifikasi E. coli O157. Koloni E. coli yang diidentifikasi sebagai E. coli O157 dicirikan dengan koloni jernih, tidak berwarna (colorless), atau bersifat sorbitol negatif (Bridson, 1998). Selain dilakukan metode kultur, dilakukan metode PCR sebagai konfirmasi molekuler. Konfirmasi molekuler perlu dilakukan untuk mengetahui adanya gen rfbE pada E. coli O157. Keuntungan utama dari metode PCR lebih spesifik, sensitif dan cepat daripada metode mikrobiologi konvensional (Bintari et al., 2014). Gen target untuk deteksi spesifik dengan teknik PCR biasanya terkait dengan faktor virulensi dari patogen. Gen target untuk E. coli O157 adalah gen rfbE untuk biosíntesis antigen O, gen terkait dengan glukoronidase (gen uidA), gen terkait dengan verotoksin (gen Shigalike toxins, stx1 dan stx2) dan gen yang terkait dengan protein yang berperanan dalam perlekatan (gen eaeA) (Suardana et al., 2010).
Penelitian ini dilakukan untuk pengujian kualitas air minum kemasan, isi ulang, dan air sumur secara mikrobiologis menggunakan teknik kultur dengan medium SMAC dan teknik PCR untuk mendeteksi keberadaan bakteri E. coli O157. Sebagai gen target adalah rfbE dengan posisi sekuens gen pada strand 5’ 7441-7680 3’,
ukuran produk PCR adalah 239 bp (Morin et al., 2004). Gen rfbE telah diidentifikasi sebagai penanda (marker) yang baik karena diturunkan pada semua fase pertumbuhan (growth phases) dari fase eksponensial awal sampai fase stasioner akhir. Gen rfbE merupakan gen target yang baik untuk mendeteksi kehadiran dari bakteri E. coli O157 dalam sampel (Liu et al., 2006).
Kelurahan Sekaran, Gunungpati, Semarang dipilih sebagai lokasi pengambilan sampel berbagai air minum karena berdasarkan data Puskesmas Sekaran pada tahun 2008-2016, kasus diare paling banyak terdapat di Kelurahan Sekaran, dibandingkan Kelurahan Ngijo, Patemon, Kalisegoro, dan Sukorejo. Pada tahun 2016 terdapat satu kasus diare berdarah di Kelurahan Sekaran. Diharapkan pada penelitian ini menjadi sumber informasi bagi masyarakat tentang kualitas air minum kemasan, isi ulang dan air sumur, di Kelurahan Sekaran, Gunungpati, Semarang serta diharapkan pula masyarakat dapat lebih selektif untuk memilih air minum yang dikonsumsi.
MATERI DAN METODE
Tempat dan waktu penelitian
Penelitian ini dilakukan pada bulan Juli-Oktober 2017 di Laboratorium Biologi Fakultas Matematika dan ilmu Pengetahuan Alam Universitas Negeri Semarang dan di Laboratorium Balai Besar Veteriner Denpasar Bali. Rancangan penelitian yang digunakan dalam penelitian ini adalah penelitian observasional dengan dilakukan pemeriksaan terhadap berbagai sampel air minum dengan teknik kultur dan PCR.
Alat dan bahan yang digunakan pada penelitian ini adalah cawan Petri, tabung reaksi, Erlenmeyer, gelas ukur, drigalski, ose lurus (Pyrex), Base Safety Cabinet (Innotech), microtube 1,5ml (Kartell), sentrifugator (Hettich Sentrifugen), vortex (Heidolph), inkubator (Memmert), Waterbath (Memmert), Mikropipet (AccupetPro), Spektrofotometer (ES Tab), Thermocycler (Peqlab), Tube PCR (Eppendorf),
Alat elektroforesis (Mupid exU), neraca analitik (Pioneer) UV transiluminator (Alpha Innotech), Nutrient Broth (OXOID), Eosin Metylen Blue Agar (OXOID), Nutrient Agar (OXOID), Sorbitol MacConkey Agar (OXOID), O157 Latex Aglutination Test (OXOID), Kontrol positif ATCC (American Type Culture Collection) 43894, GeneJET Genomic DNA Purification Kit (Thermo Scientific), Dreamtaq Green PCR master mix (Thermo Scientific), primer rfbE 239 bp, Gel agarosa 1,5% (Sigma), 0,5 mg/L ethidium bromida (BioChemica), Buffer TBE 1X, Low DNA mass ladder (Thermo Scientific), Loading dye (Thermo Scientific), parafilm (M roll size 4 in x 125 ft).
Kultur bakteri E. coli O157 dengan medium SMAC
Kultur E. coli O157 dilakukan dengan cara sampel diinokulasikan pada medium NB dan diinkubasi pada suhu 37°C selama 24 jam. Sampel yang tumbuh pada medium NB disebar pada medium EMBA steril dengan menggunakan gelas bengkok, diinkubasi pada suhu 37°C selama 24 jam. Hasil positif ditunjukkan dengan koloni berwarna hijau metalik dengan titik hitam di tengah. Koloni E. coli yang positif diinokulasikan pada medium NA miring. Selanjutnya dipindah ke medium selektif SMAC dan diinkubasi pada suhu 37°C selama 24 jam. Hasil positif menunjukkan koloni bakteri tidak berwarna (colorless). Konfirmasi terhadap uji SMAC dilakukan dengan uji O157 latex aglutination test (OXOID). Sebanyak 2-3 ose isolat presumtif E. coli O157 ditumbuhkan pada media Brain Heart Infussion (BHI) diinkubasi pada suhu 37°C selama 24 jam. Selanjutnya direaksikan dengan tetes pereaksi latex. Hasil uji positif ditandai dengan terbentuknya aglutinasi sesuai dengan kontrol positif yang tersedia.
Isolasi DNA genomik bakteri
Isolasi DNA bakteri dilakukan dengan menggunakan GeneJET Genomic DNA Purification Kit (Thermo Scientific). Sampel sebanyak 1 ml dikulturkan di dalam 9 ml medium NB dan diinkubasi pada suhu 37°C selama 24 jam. Sampel diambil 1,5 ml dalam microtube dan
disentrifugasi dengan kecepatan 5000 g selama 10 menit. Pellet yang diperoleh kemudian ditambahkan 180 µl Lysis buffer. Campuran tersebut kemudian divortex dan diinkubasi pada suhu 37°C selama 30 menit. Setelah itu ditambahkan 200 µl Lysis solution dan 20 µl Proteinase K. Kemudian divortex dan diinkubasi dalam waterbath pada suhu 56°C selama 30 menit. Setelah diinkubasi, ditambah dengan 20 µl RNAse A Solution dan divortex. Kemudian diinkubasi pada suhu ruang selama 10 menit.
Langkah selanjutnya yaitu penambahan 400 µl ethanol 50% dan divortex sampai homogen. Kemudian campuran tersebut dipindahkan ke dalam kolom dan disentrifugasi dengan kecepatan 6000 g selama 1 menit. Pemindahan kolom ke tube yang baru dan ditambahkan 500 µl wash buffer I dalam kolom tersebut, disentrifugasi dengan kecepatan 8000 g selama 1 menit. Selanjutnya supernatan dibuang dan ditambahkan 500 µl wash buffer II dalam kolom, kemudian disentrifugasi dengan kecepatan 12000 g selama 3 menit. Tube dibuang dan diganti dengan tube 1,5 ml. Penambahan 200 µl Ellusion buffer dalam kolom, kemudian diinkubasi pada suhu ruang selama 2 menit dan disentrifugasi dengan kecepatan 8000 g selama 1 menit. Selanjutnya tube yang berisi DNA hasil isolasi disimpan pada suhu -20°C.
Amplifikasi DNA dengan PCR
Hasil isolasi DNA dianalisis menggunakan primer rfbE dengan posisi sekuens gen pada strand 5’ 7441-7680 3’, ukuran produk PCR adalah 239 bp (Morin et al. 2004). Komposisi PCR adalah 2 μL DNA template, 25 μL PCR master mix (Thermo Scientific), 2 μL primer F, 2 μL primer R, 19 μL ddH2O dengan volume reaksi 50 μL. Setelah itu, dilakukan amplifikasi dengan menggunakan Thermocycler sebanyak 30 siklus setiap siklus terdiri dari pradenaturasi pada suhu 95ºC selama 5 menit, denaturasi pada suhu 94ºC selama 1 menit, annealing pada suhu 56ºC selama 1 menit, ekstensi pada suhu 72ºC selama 1 menit 30 detik, dan ekstensi akhir pada suhu 72ºC selama 10 menit.
Visualisasi produk PCR / elektroforesis
Masing-masing 5 μl produk amplifikasi dicampur dengan 2 μl loading dye. Setelah tercampur dengan baik, masing-masing dimasukkan ke dalam sumur gel agarosa 1% yang terendam dalam tanki yang berisi buffer Tris acetid acid-EDTA (TAE 1X). Dimasukkan juga marker DNA 100bp ke dalam sumur gel agarosa untuk mengetahui ukuran DNA produk PCR, kemudian elektroforesis dijalankan selama 30 menit dengan tegangan konstan 100 volt, dan gel diamati di bawah sinar Ultra Violet (UV). Hasil yang diperoleh berupa pola pita DNA (band DNA) yang menunjukkan jumlah dan pola yang berbeda. Data yang diperoleh dianalisis secara deskriptif, disajikan dalam bentuk tabel dan gambar.
HASIL
Koloni pada media EMBA yang positif E. coli selanjutnya diinokulasi ke media NA (Nutrien Agar) miring, dan selanjutnya ditumbuhkan pada medium SMAC (Sorbitol Mac Conkey Agar). SMAC merupakan media selektif untuk menumbuhkan E. coli O157. Sebanyak 8 sampel positif E. coli pada media EMBA, dan semua menunjukkan hasil positif E. coli O157 pada media SMAC ditunjukkan oleh koloni tidak berwarna seperti terlihat pada Gambar 1. Hasil ini menunjukkan bahwa 8 isolat tidak memfermentasikan sorbitol sehingga menghasilkan koloni yang tidak berwarna (colorless).
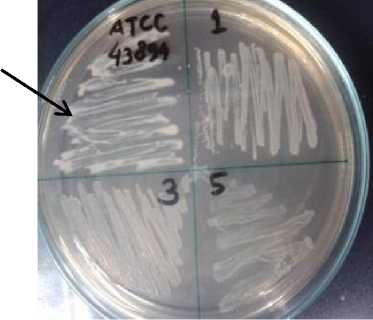
Gambar 1. Koloni E. coli O157 pada media SMAC ditunjukkan dengan warna colorless seperti pada kontrol E. coli O157 ATCC 43894.
Koloni bakteri colorless pada media SMAC selanjutnya diuji dengan O157 latex aglutination test (OXOID). Koloni E. coli O157 yang positif memperlihatkan reaksi aglutinasi terhadap antiserum E. coli O157. Delapan sampel positif pada medium SMAC menunjukkan hasil positif pula pada uji aglutinasi lateks yang disajikan pada Gambar 2.
Hasil positif pada uji aglutinasi lateks menunjukkan bahwa isolat presumtif (diduga) E. coli O157 pada media SMAC merupakan isolat E. coli O157.
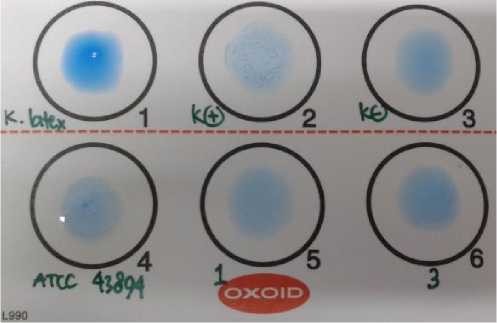
Gambar 2. Hasil uji O157 latex aglutination test dari isolat E. coli O157. Kontrol ATCC 43894 ditunjukkan dengan tanda panah yang membentuk adanya presipitasi.
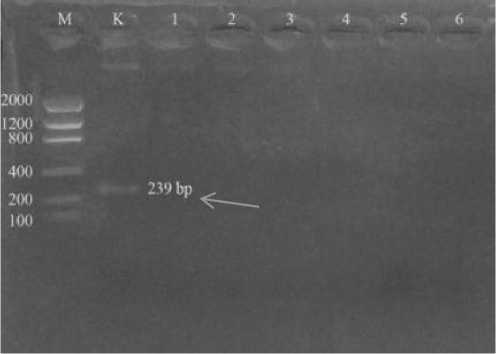
Gambar 3. Elektroforegram menggunakan gen rfbE sebagai gen target. Hasil PCR menunjukkan semua sampel negatif. M= low DNA mass ladder (Thermo scientific), K= kontrol positif ATCC 43894 yang menunjukkan ukuran 239 bp.
Konfirmasi molekuler dilakukan dengan metode PCR menggunakan primer spesifik gen rfbE dengan ukuran produk PCR 239 bp dan
sequence F : 5’- GTG CTT TTG ATA TTT CGA GTA CAT TGG -3’ serta sequence R: 5’- TTT ATA TCA CGA AAA CGT GAA ATT GCT GAT -3’. Semua sampel (20 sampel air) menunjukkan hasil negatif. Elektroforegram produk PCR dapat dilihat pada Gambar 3.
PEMBAHASAN
Air merupakan salah satu transmisi E. coli O157, air mudah terkontaminasi feses manusia atau hewan (Gibson, 2014). Air minum isi ulang diambil dari depot air minum isi ulang yang sumber airnya didapat dari Gunung Ungaran. Proses pengolahan (water treatment) air minum diproses melalui 3 tahap yaitu: penyaringan, desinfeksi, dan pengisian. Penyaringan untuk menghilangkan kotoran dan bau yang terkandung dalam air. Desinfeksi bertujuan untuk menghilangkan sebagian besar mikroba dan membunuh bakteri patogen dalam air, dengan menggunakan proses ozonisasi, ultraviolet (UV) dan reverse osmosis sistem (pemurnian air). Pengisian merupakan tahap akhir berupa pengemasan air yang telah diproses. Pada proses pengolahan air yang harus diperhatikan adalah kapasitas filter-filter pendukung, media yang digunakan, bahan tabung filter yang digunakan serta perawatan alat yang digunakan (Kandou, 2009).
Kemungkinan kontaminasi berasal dari proses pengolahan yang tidak memenuhi standar yang telah ditentukan dan sumber air yang telah tercemar. Sanitasi buruk dan higienitas rendah dari depot air minum isi ulang juga dapat menyebabkan kontaminasi. Di sisi lain kontaminasi dapat terjadi pada galon jika tidak dibersihkan dengan baik kemungkinan bakteri masih menempel pada dinding galon. Lokasi depot air minum isi ulang harus bebas dari pencemaran seperti tempat pembuangan sampah dan kotoran, serta pabrik lain yang dapat menimbulkan pencemaran pada air minum seperti bengkel, las dan cat (Natalia et.al., 2014). Konstruksi bangunan depot air minum isi ulang harus memenuhi persyaratan tata ruang dan syarat fisik. Syarat fisik meliputi kondisi lantai, kondisi
dinding, kondisi atap, harus kuat, aman, dan mudah dibersihkan (Taib, 2012).
Lokasi pengambilan sampel air sumur di rumah warga yang padat penduduk. Selain itu sumurnya terletak di dekat selokan. Sanitasi buruk dan higienitas rendah dari lingkungan sumur menyebabkan adanya kontaminasi. Air sumur kemungkinan terkontaminasi oleh feses manusia atau hewan. Lokasi air sumur juga harus bebas dari pencemaran seperti tempat pembuangan kotoran dan sampah. Air sumur berpeluang terhadap penyebaran bakteri E. coli O157 (Radji et al., 2010).
Berbeda dengan air isi ulang dan air sumur, air kemasan yang diambil dari 4 merk yang ada di pasaran menunjukkan hasil negatif. Hal itu dikarenakan proses yang telah terstandar dan sesuai dengan prosedur. Proses pengolahan air minum kemasan melalui 4 tahap yaitu proses water treatment system, proses water sterilization, proses quality control system dan proses pengemasan. Proses selanjutnya adalah distribusi, proses distribusi dilakukan 5-6 jam setelah proses pengemasan agar kondisi gas ozon yang terkandung dalam air menguap dan gas ozon tersebut kembali menjadi oksigen.
Konfirmasi molekuler menggunakan primer spesifik gen rfbE pada semua sampel (20 sampel) menunjukkan hasil negatif, tidak sama seperti pada kontrol positif ATCC 43894 (Gambar 3). Gen rfbE adalah salah satu gen yang terlibat dalam sintesis rantai samping O. Rantai samping O adalah bagian dari lipopolisakarida (LPS) yang esensial dari membran luar bakteri. Lipopolisakarida (LPS) sering terlibat dalam proses patologi bakteri Gram negatif dan berperan dalam infektivitas (kemampuan patogen menyebabkan infeksi) dari pathogen (Abongo & Momba, 2008). Gen rfbE digunakan sebagai gen target karena gen rfbE mengkode antigen O untuk E. coli O157. Gen rfbE juga digunakan untuk membedakan E. coli O157 dan E. coli non O157 (Suria, 2012).
Hasil PCR pada Gambar 3 menunjukkan bahwa seluruh sampel tidak menghasilkan pita DNA dengan ukuran 239bp. Primer yang digunakan
mengacu pada protokol (Morin et.al., 2004), bukan mendesain sendiri. Faktor yang mempengaruhi keberhasilan PCR adalah pemilihan primer. Keberhasilan teknik PCR didasarkan pada kesesuaian primer, efisiensi, dan optimasi PCR. Primer yang tidak spesifik dapat menyebabkan teramplifikasinya daerah lain di dalam genom non target atau tidak ada daerah genom yang teramplifikasi (Nugroho, 2015). Penelitian lain yang menggunakan gen rfbE sebagai gen target memiliki ukuran yang berbeda karena menggunakan sekuen primer yang berbeda. Hasil PCR yang negatif pada sampel positif E. coli O157 (media SMAC dan uji aglutinasi lateks) kemungkinan karena isolat E. coli O157 dalam penelitian ini memiliki sekuen gen rfbE yang berbeda, terutama pada situs pengenalan primer (nukleotida nomor 74417680). Pemilihan primer yang tidak sesuai pada penelitian ini sehingga semua sampel (20 sampel air minum) negatif gen rfbE. Optimasi suhu dan waktu annealing juga mempengaruhi hasil, optimasi annealing pada penelitian ini telah diulang sebanyak sepuluh kali.
Amplifikasi PCR pada tingkatan serotipe atau varietas memiliki tingkat kesulitan tinggi karena spesies tersebut kemungkinan mengalami mutasi (Nugroho, 2013). Varian E. coli O157 ada O157 serotipe non motil dan O157:H-; E. coli O157 serotipe H ada berbagai macam yaitu E. coli O157:H3, E. coli O157:H7, E. coli O157:H12, E. coli O157:H16, E. coli O157:H38, dan E. coli O157:H45 (Aris et.al., 2012). Banyaknya serotipe E. coli O157 mengakibatkan tingkat kesulitan yang tinggi untuk amplifikasi DNA.
SIMPULAN
Berdasarkan hasil deteksi Escherichia coli O157 pada beberapa air minum di Kelurahan Sekaran Gunungpati Semarang dapat disimpulkan sebanyak 8 sampel air minum dari 20 sampel (40%) positif E. coli O157 pada media SMAC Agar dan O157 latex aglutination test. Dari 8 sampel air positif E. coli O157, 4 sampel merupakan air sumur (20%) dan 4 sampel air minum isi ulang (20%).
UCAPAN TERIMA KASIH
Ucapan terima kasih disampaikan kepada Biologi FMIPA Universitas Negeri Semarang, Fakultas Kedokteran Hewan Universitas Udayana, Dinas Kesehatan Kota Semarang, Puskesmas Sekaran, dan Balai Besar Veteriner Denpasar Bali.
KEPUSTAKAAN
Abong’o BO, Momba MNB. 2008. Prevalence and potential link between E. coli O157:H7 isolated from drinking water, meat, and vegetables and stools of diarrheic confirmed and non-confirmed HIV/AIDS patients in the Amathole District-South Africa. J. Appl. Microbiol 105.
Aris M, Sukenda, Haris E, Sukadi MF, Yuhana M. 2013. Identifikasi molekuler bakteri patogen dan desain primer PCR. Jurnal Budidaya Perairan 1 (4).
Bintari SH, Fibriana F, Mustikanintyas D & Iswari RS. 2014. PCR approach for rapid detection of Escherichia coli in tempe using a specific primer. Journal of Biological Researches 19: 54-58.
Bridson EY. 1998. The Oxoid Manual. 8th Ed.
Franz E, Klerks MM, De Vos OJ, Termorshuizen AJ, van Bruggen AHC. 2007. Prevalence of shiga toxin-producing Escherichia coli stx1, stx2, eaeA, and rfbE genes and survival of E. coli O157:H7 in manure from organic and low-input conventional dairy farms. Applied and Environmental Microbiology 73: 21802190.
Gibson KE. 2014. Viral pathogen sin water: occurrence, public health impact, and available control strategies. Curr.Opin.Virol 4: 50–57.
Kandou FEF. 2009. Analisis Molekuler Escherichia coli serotype O157:H7 pada air minum dalam kemasan dan isi ulang
menggunakan teknik Polymerase Chain Reaction (PCR) dengan rfbE sebagai gen target. Chem. Prog. 2(1): 8-14.
Liu YZ, Gong N, Morin O, Pui M, Cheung H, Zhang, Li XF. 2006. Electronic deoxyribonucleic acid (DNA) microarray detection of viable pathogenic Escherichia coli, Vibrio cholerae, and Salmonella typhi. Anal. Chim. Acta 578:75-81.
Morin NJ, Gong Z & Li XF. 2004. Reverse Transcription-Multiplex PCR Assay for Simultaneous Detection of Escherichia coli O157: H7, Vibrio cholerae OI, and
Salmonella typhi. Clinical Chemistry 50(11): 2037-2044.
Natalia LA, Bintari SN, Mustikaningtyas D. 2014. Kajian kualitas bakteriologis air minum isi ulang di Kabupaten Blora. Unnes J. Life Sc. 3(1): 31-38.
Nugroho FAD. 2015. Identifikasi pola haplotipe DNA mitokondria udang jari (Metapenaeus elegans) Segara Anakan Kabupaten Cilacap Jawa Tengah menggunakan enzim restriksi HindIII. Skripsi. Malang: UIN Maliki Malang.
Odonkor ST, Ampofo JK. 2013. Escherichia coli as an indicator of bacteriological quality of water: an overview. Microbiology Research 4: 5-11.
Radji M, Puspaningrum A, Sumiati A. 2010. Deteksi cepat bakteri Escherichia coli dalam Sampel Air dengan metode Polymerase Chain Reaction menggunakan primer 16E1 dan 16E2. Makara Sains 14(1): 39-43.
Sharma VK. 2006. Real-time reverse transcription-multiplex PCR for
simultaneous and specific detection of rfbE and eae genes of Escherichia coli O157:H7. Molecular and Cellular Probes 20: 298-306.
Soemirat J. 2004. Kesehatan Lingkungan. Yogyakarta: Gadjah Mada University Press.
Suardana IW, Artana WT, Asmara W, Daryoni BS. 2010. Identifikasi Escherichia coli O157:H7 serta deteksi gen Shiga Like Toxin 1 dan 2 aal feses hewan, daging, dan feses manusia. Jurnal Veteriner 11(4): 264-270.
Suria MS. 2013. Multiplex Polymerase Chain Reaction (PCR) efficiency in detection of
pathogenic Escherichia coli O157:H7. International Research Journal 20(6).
Taib DA. 2012. Aspek kualitas dan hygiene sanitasi depot air minum isi ulang (DAMIU) di kecamatan kota utara kota Gorontalo. Public Health Journal 1(1):93-104.
Waluyo L. 2009. Mikrobiologi Lingkungan. Malang: UMM Press.
Wandrivel R, Suharti N, Lestari Y. 2012. Kualitas Air Minum yang diproduksi depot air minum isi ulang di Kecamatan Bungus Padang berdasarkan persyaratan mikrobiologi. Jurnal Kesehatan Andalas 1(3): 129-133.
Yaron S, Matthews KR. 2002. A reverse transcriptase-polymerase chain reaction assay for detection of viable Escherichia coli O157:H7: investigation of specific target genes. Journal of Applied Microbiology 92: 633-640.
62
Discussion and feedback