DESAIN IN SILICO DNA PROBE PENDETEKSI MUTASI DAERAH RESISTENSI QUINOLON Gen gyrA DAN gyrB Mycobacterium tuberculosis
on
SIMBIOSIS VII (2 ): 44-50 http://ojs.unud.ac.id/index.php/simbiosis
Program Studi Biologi FMIPA UNUD
ISSN: 2656-7784
September 2019
DESAIN IN SILICO DNA PROBE PENDETEKSI MUTASI DAERAH RESISTENSI QUINOLON Gen gyrA DAN gyrB Mycobacterium tuberculosis
IN SILICO DNA PROBE DESIGN FOR MUTATION DETECTION OF QUINOLON RESISTANCE AREA gyrA AND gyrB GENE Mycobacterium tuberculosis
Tasya Pramiswari, Jennifer Tamara, Ni Made Febrianti, Sagung Chandra Yowani Program Studi Farmasi, Fakultas Matematika dan Ilmu Pengetahuan Alam, Universitas Udayana, Bukit Jimbaran, Bali-Indonesia, 80361
Email: tasyapramiswari13@gmail.com
ABSTRAK
Fluorokuinolon (FQ) merupakan obat utama yang digunakan pada terapi MDR-TB sehingga resistensi terhadap FQ dapat menyebabkan kematian dan resiko kegagalan terapi pada pasien MDR-TB. Mutasi pada gen gyrA dan gen gyrB dari Mycobacterium tuberculosis bertanggung jawab terhadap terjadinya resistensi FQ. Mutasi region QRDR pada gen gyrA paling tinggi ditemukan pada kodon 94, sedangkan region QRDR pada gen gyrB ditemukan pada kodon 500. Penyakit M. tuberculosis yang resisten terhadap FQ dapat dideteksi menggunakan metode Real Time Polymerase Chain Reaction (RT-PCR) dengan DNA probe. Penelitian ini akan mendesain urutan nukleotida probe berjenis TaqMan menggunakan program Clone Manager Suite 9.2. Hasil rancangan DNA probe kemudian dianalisis 2 tahap yaitu berdasarkan kriteria probe secara umum dan berdasarkan kriteria pelabelan TaqMan probe. Rancangan DNA probe mutan menggunakan program menghasilkan 1 probe untuk mutasi spesifik Asp94Ala pada gen gyrA dan 33 probe untuk mutasi spesifik Asp500Ala pada gen gyrB. Setelah dianalisis dengan kedua kriteria, didapat probe A94MA1 dengan urutan 5’-TCGATCTACGCCAGCCTGGT-3’ dan probe B500MA12 dengan urutan 5’-TACCACAAGCTCGTGCTGATGGC-3’. Hasil probe tersebut memenuhi kedua kriteria dan dapat digunakan untuk mendeteksi mutasi pada kodon 94 gen gyrA dan kodon 500 gen gyrB Mycobacterium tuberculosis.
Kata kunci: MDR-TB, gen gyrA, gen gyrB, TaqMan probe, Real-Time PCR
ABSTRACT
Fluoroquinolone (FQ) is the main drug used in MDR-TB therapy resistance to FQ can cause death and increase the risk of treatment failure in MDR-TB patients. Mutations in gyrA gene and gyrB gene from Mycobacterium tuberculosis are responsible for the occurrence of FQ resistance. The highest mutation of gyrA gene in QRDR was found in codon 94, while mutations in gyrB gene was found in codon 500. M. Tuberculosis which resistant to FQ can be detected using the Real Time Polymerase Chain Reaction (RT-PCR) method with DNA probe. This study will design the nucleotide sequence of the TaqMan type probe using the Clone Manager Suite 9.2 program. The results of the DNA probe design were then analyzed in two stages, which is based on the probe criteria in general and based on the TaqMan probe labeling criteria. The design of the mutant probe DNA using the program produced 1 probe for Asp94Ala specific mutations in the gyrA gene and 33 probes for Asp500Ala specific mutations in the gyrB gene. After being analyzed by the two criteria, it was obtained the A94MA1 probe with the 5 '-TCGATCTACGCCAGCCTGGT-3' sequence and B500MA12 probe with the order of 5 '-TACCACAAGCTCGTGCTGATGGC-3'. The results of these probes meet both criteria and can be used to detect mutations in codon 94 gyrA genes and codons 500 gyrB genes of Mycobacterium tuberculosis.
Keywords: MDR-TB, gyrA gene, in silico, TaqMan probe, Real-Time PCR
PENDAHULUAN
XDR-TB (Extensively Drug-Resistant Tuberculosis) didefinisikan sebagai TB resisten Isoniazid (INH) dan Rifampisin (RIF) (MDR-TB) serta setidaknya resisten terhadap salah satu dari dua golongan obat antituberkulosis (OAT) lini kedua yang penting dalam pengobatan MDR-TB yaitu fluorokuinolon (FQ) dan OAT lini kedua dalam bentuk injeksi yaitu kapreomisin, kanamisin, atau amikasin. Pada 2017, terdapat 558.000 kasus MDR-TB ditingkat global dengan prevalensi terbanyak pada negara China, India dan Russia. Dari 127 negara anggota WHO, 113 negara menyatakan terjadi peningkatan penderita MDR-TB menjadi XDR-TB sebanyak 8,5% (WHO, 2018).
Pasien MDR-TB memerlukan waktu terapi sekitar 18-24 bulan (Nugrahaeni dan Malik, 2015). FQ adalah obat lini kedua utama dari pengobatan MDR-TBsehingga adanya resistensi terhadap FQ merupakan salah satu penanda dari kondisi XDR-TB yang dapat menyebabkan kematian dan meningkatkan risiko kegagalan pada pasien MDR-TB (Kaswa et al., 2014). FQ bekerja dengan menghambat DNA gyrase bakteri M. tuberculosis (Rinanda, 2015).
DNA gyrase dikode oleh dua subunit yaitu gen gyrA dan gen gyrB. Mutasi pada kedua gen dapat menyebabkan terjadinya resistensi FQ. Quinolone Resistance Determining Region (QRDR) adalah daerah penentu terjadinya resistensi FQ pada gen gyr (Zhang et al., 2014). QRDR gen gyrA berada pada daerah kodon 67 hingga 106 (Matrat et al., 2008) sedangkan gen gyrB memiliki QRDR yang terletak pada daerah kodon 500 hingga 540 (Disratthakit et al., 2016).
Mutasi paling tinggi ditemukan pada QRDR gen gyrA M. tuberculosis kodon 94 sebanyak 32,6%. Mutasi pada kodon 94 dapat menyebabkan perubahan asam amino yang bervariasi yaitu Asp94His, Asp94Tyr, A94Asn, Asp94Ala, Asp94Gly, dan Asp94Cys (Zhang etal., 2014). Mutasi pada QRDR gen gyrB M. tuberculosis paling sering terjadi pada kodon 500 dan 538 (Disratthakit et al., 2016). Mutasi gen gyrB pada kodon 500 dapat menyebabkan perubahan asam amino yang bervariasi yaitu Asp500Asn dan Asp500Ala. Salah satu cara mengetahui resistensi pada MDR-TB dapat dilakukan dengan pendeteksian cepat agar dapat memberikan terapi yang tepat. Deteksi M. tuberculosis yang resisten terhadap FQ dapat dilakukan menggunakan metode real-time polymerase chain reaction (RT-PCR) dengan DNA probe mutan. Salah satu sistem deteksi DNA probe mutan yang dapat digunakan untuk RT-PCR adalah TaqMan probe (Navarro et al., 2015). Deteksi mutasi dan skiring subtipe spesifik dari mikroba patogen dapat juga menggunakan TaqMan probe. TaqMan probe merupakan oligonukleotida yang memiliki pewarna reporter fluoresen pada ujung 5’ dan pewarna quencher pada ujung 3’.
Desain DNA probe mutan dibuat menggunakan software Clone Manager Suite 9.2. Perancangan dengan software dapat mengoptimalkan hasil desain dibandingkan perancangan manual, memaksimalkan probabilitas keberhasilan hibridisasi DNA probe dengan urutan DNA target serta mampu meminimalkan waktu yang diperlukan (Yilmaz et al., 2011).
Desain DNA probe mutan yang dihasilkan digunakan untuk mendeteksi mutasi pada kodon 94 gen gyrAdan kodon
500 gen gyrBM. Tuberculosis sehingga dapat menentukan rejimen obat yang sesuai. Mutasi pada kedua gen ini menunjukkan adanya resistensi terhadap FQ, yang merupakan salah satu obat dalam rejimen lini kedua OAT bagi penderita MDR-TB. DNA probe mutan dapat mendeteksi adanya mutasi lebih awal sehingga penggunaan FQ pada pasien dapat dihentikan dan digantikan dengan obat lini ketiga.
MATERI DAN METODE
Alat yang diperlukan seperti laptop (Windows 10 32 bit), modem dan program Clone Manager Suite 9.2 untuk perancangan serta analisis DNA probe. Bahan yang digunakan yaitu urutan nukleotida gen gyrA dan gyrB M. tuberculosis H37Rv (wild-type) (Accession No. NC_000962) yang diperoleh dari database www.ncbi.nlm.nih.gov dan sepasang primer. Primer digunakan untuk menganalisis hasil rancangan probe yang mana terdiri dari primer forward serta reverse.
Primer gen gyrA terdiri dari primer forward (gen-gyrA-F) dengan urutan 5’-GCAGCTACATCGACTATGC-3’ dan primer reverse (gen-gyrA-R) dengan urutan 5’-GGCTTCGGTGTACCTCATC-3’ sedangkan primer gen gyrB terdiri dari primer forward (gen-gyrB-F) dengan
urutan 5' GAGAGTTGGTGCGGCGTAA 3' dan primer reverse (gen-gyrB-R) dengan urutan 5' GCGGTCGGAGTATGCGAAT-3'.
Penentuan urutan nukleotida target
Urutan DNA probe mutan terpilih yaitu kodon 94 gyrA dan 500 gyrB gen M. tuberculosis. Hal ini didasarkan pada
prevalensi mutasi tertinggi yang terjadi pada gen gyrA dan gen gyrB. DNA probe mutan dirancang di dalam daerah amplifikasi pada posisi nukleotida no 77396 untuk gen gyrA dan posisi nukleotida no. 1271-1755 untuk gen gyrB. Daerah amplifikasi dibatasi oleh sepasang primer untuk masing-masing gen.
Analisis primer
Primer yang digunakan diperoleh dari studi pustaka pada penelitian Sasmitha (2017) dan Dewi (2018). Primer gen gyrA terdiri dari primer forward (gen-gyrA-F) dengan urutan 5’-
GCAGCTACATCGACTATGC-3’ dan primer reverse (gen-gyrA-R) dengan urutan 5’-GGCTTCGGTGTACCTCATC-3’ sedangkan primer gen gyrB terdiri dari primer forward (gen-gyrB-F) dengan urutan 5' GAGAGTTGGTGCGGCGTAA 3' dan primer reverse (gen-gyrB-R) dengan urutan 5' GCGGTCGGAGTATGCGAAT 3'.
Perancangan DNA probe
Perancangan DNA probe mutan dilakukan secara in silico menggunakan software Clone Manager Suite 9.2. Urutan nukleotida gen gyrA dan gyrB diinput ke dalam software untuk diolah sehingga didapatkan hasil berupa urutan nukleotida probe mutan. Pada tahap ini dihasilkan beberapa rancangan urutan nukleotida DNA probe mutan.
Analisis hasil perancangan DNA probe
Hasil rancangan DNA probe yang diperoleh kemudian dianalisis
menggunakan software Clone Manager Suite 9.2 sesuai dengan kriteria DNA probe mutan secara umum dan kriteria pelabelan TaqMan probe. Kriteria pelabelan TaqMan probe meliputi tidak terdapat basa G pada
ujung 5’, dan mengandung basa C lebih banyak daripada basa G (McPherson and Moller, 2006).
HASIL
Hasil DNA probe mutan dianalisis sesuai dengan kriteria probe secara umum dan kriteria pelabelan TaqMan probe. Hasil analisis probe berdasarkan kriteria secara umum menghasilkan 1 buah DNA probe mutan gyrA sedangkan terdapat 33 buah DNA probe mutan gyrB. Hasil dapat dilihat pada Tabel 1. Setelah itu dilakukan analisis kembali sesuai kriteria pelabelan TaqMan probe dan didapat DNA probe yang sesuai kriteria yaitu probe A94AM1 dengan urutan 5’-CGATCTACGCCAGCCTGGT-3’ untuk mendeteksi mutasi pada kodon 94 gen gyrA dan probe B500MA12 dengan urutan5’TACCACAAGCTCGTGCTGAT GGC-3’ untuk mendeteksi mutasi pada kodon 500 gen gyrB.
PEMBAHASAN
DNA probe mutan dirancang pada kodon 94 gyrA dan kodon 500 gyrB M. tuberculosis. Primer yang digunakan diperoleh dari studi pustaka. Perancangan DNA probe mutan dilakukan dengan menggunakan urutan nukleotida wild-type dari gen gyrA dan gyrB M. tuberculosis H37Rv Accession No. NC_000962 dari database www.ncbi.nlm.nih.gov.
Probe adalah urutan nukleotida yang memiliki afinitas kuat dengan target spesifik sehingga dapat digunakan untuk mendeteksi adanya mutasi. Desain probe berjenis TaqMan memiliki spesifisitas yang tinggi, kemampuan untuk reaksi multiple tetapi memerlukan biaya yang tinggi (Anonim, 2006). Perancangan DNA probe mutan menggunakan software Clone Manager Suite 9.2.
Analisa DNA probe mutan dilakukan melalui dua tahap yaitu analisis sesuai dengan kriteria probe secara umum dan pelabelan TaqMan Probe. Analisa tahap awal dilakukan berdasarkan kriteria DNA probe secara umum. Kriteria umum dapat dilihat pada Tabel 1.
Tabel 1. Kriteria Probe Umum
|
Kriteria |
Keterangan |
|
Panjang %GC Tm Dimer Runs Repeats Hairpin |
18-30 basa 40-60% 5-10°C ≥ Tm primer (optimal 70°C) Tidak ada ≤ 4 ≤ 4 Tidak ada |
Tabel 1. memperlihatkan hasil analisis kriteria probe secara umum yang mana semua memenuhi kriteria DNA probe secara umum. Setelah itu dilakukan analisis sesuai kriteria TaqMan probe. TaqMan probe merupakan probe yang memiliki pewarna reporter fluoresen yang melekat pada ujung 5’ dan pewarna quencher pada ujung 3’.
Kriteria pertama dari pelabelan TaqMan probe yaitu tidak boleh ada basa G pada posisi nukleotida pertama dan kedua dari ujung 5’ (Rychlik, 2010). Kriteria kedua yaitu DNA probe mengandung jumlah basa C lebih banyak atau sama dengan basa G (McPherson and Moller, 2006). DNA probe mutanyang memenuhi kedua kriteria yaitu probe A94MA1, B500MA4, B500MA7,B500MA12,B500MA13, B500MA14,B500MA15,B500MA16,B500 MA18,B500MA24,B500MA25,B500MA2 6,B500MA27,B500M32 dan probe B500MA33. Probe dapat ditempelkan label
yang terdiri dari reporter dan quencher. Label yang biasanya digunakan untuk TaqMan probe adalah FAM sebagai reporter dan TAMRA sebagai quencher (Behlke et al., 2005; Didenko, 2006). Kedua label reporter dan quencher ditempelkan pada ujung berlawanan pada urutan nukleotida probe mutan (Livak et al., 1995). Pelabelan pewarna direkomendasikan agar dilekatkan pada basa C atau T, tetapi umumnya dilekatkan pada basa T (Bishop et al., 2015). Dari ke-14 DNA probe pada gen gyrB yang memenuhi kriteria pelabelan TaqMan, dipilih salah satu DNA probe mutan yaitu B500MA12 karena memiliki posisi mutasi ada ditengah urutan probe.

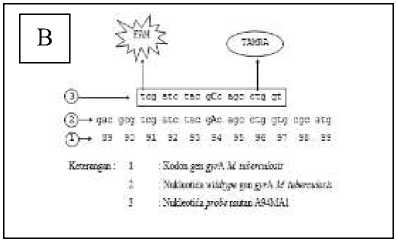
Gambar 1. Pelabelan desain DNA probe (A) DNA Probe A94MA1 dan (B) DNA Probe B500MA12 spesifik terhadap
perubahan A → C pada kodon 94 dan 500.
Berdasarkan desain yang telah dilakukan, dihasilkan DNA probe mutan pada gyrA yaitu 5’-TCGATCTACGCCAGCCTGGT-3’ yang dapat mendeteksi mutasi pada
kodon ke-94 dengan perubahan nukleotida GAC menjadi GCT sedangkan desain yang dilakukan pada gen gyrB menghasilkan urutan5’TACCACAAGCTCGTGCTGAT GGC-3’ dapat mendeteksi adanya mutasi nukleotida ke-500 dengan perubahan nuleotida GAT menjadi GCT.
KESIMPULAN
Rancangan DNA probe mutan menggunakan software menghasilkan 1 probe untuk mutasi spesifik Asp94Ala pada gen gyrA dan 33 probe untuk mutasi spesifik Asp500Ala pada gen gyrB. Setelah dianalisis dengan kedua kriteria, didapat probe A94MA1 dengan urutan 5’-TCGATCTACGCCAGCCTGGT-3’ dan probe B500MA12 dengan urutan 5’-TACCACAAGCTCGTGCTGATGGC-3’. Hasil probe tersebut memenuhi kedua kriteria dan dapat digunakan untuk mendeteksi mutasi pada kodon 94 gen gyrA dan kodon 500 gen gyrB Mycobacterium tuberculosis.
DAFTAR PUSTAKA
Anonim a. 2006. Real-Time PCR Applications Guide. Bulletin 5279.USA: Bio-Rad Laboratories
Inc. (Diakses pada 19 Juni 2019).
Behlke, M. A., L. Huang, L. Bogh, S. Rose, and E. J. Devor. 2005. Fluorescence and Fluorescence Applications. United States: Integrated DNA
Technologies.
Bishop, J. L., Campbell, S. A., Farrell, P., Fitzgerald, M., Haugen, M., Kocmond, W., Madden, D. E., Murray, W. E., and Persing. D. H. 2015. Designing Real-Time Assays on the SmartCycler® II System, United States: Cepheid Technical
Support.pp 1-8. (Diakses pada 19 Juni 2019).
Borah, P. 2011. Primer Designing for PCR. Science Vision. 11: 134-136.
Dewi, D.S.W. 2018. Identifikasi Mutasi Gen gyrA dan gyrB pada Isolat Klinik Multidrug Resistant Tuberculosis (MDR-TB) sebagai Penanda Extensively Drug-Resistant
Tuberculosis (XDR-TB) dengan Metode Multiplex Polymerase Chain Reaction. Skripsi. Program Studi Farmasi, FMIPA, Universitas Udayana.
Didenko, V. V. 2001. DNA Probes Using Fluorescence Resonance Energy Transfer (FRET): Designs and
Applications. JournalBiotechniques. 31. pp 1106-1121.
Disratthakit, A., T. Prammananan, C. Tribuddharat, I. Thaipisuttkul, N, M. Leechawengwongs, dan A.
Chalprasert. 2016. Role of gyrB Mutation in PreExtensively and Extensively Drug Resistant
Tuberculosis in Thai Clinical Isolates.American Society for
Microbiology. 60(9): 5189-5197.
Kaswa, M. K., Aloni, M., Nkuku, L., Bakoko, B., Lebeke, R., Nzita, A. 2014. Pseudo-Outbreak of Pre-Extensively Drug-Resistant (Pre-XDR) Tuberculosis in Kinshasa: Collateral Damage Caused by False Detection of Fluoroquinolone Resistance by GenoType MTBDRsl. Journal of Clinical Microbiology. 52: 2876-2880.
Livak, K. J., Flood, S. J. A., Marmaro, J., Glusti, W., and Deetz, K. 1991. Oligonucleotides with Fluorescent Dyes at Opposite Ends Provide a Quenched Probe System Useful for
Detecting PCR Product and Nucleic Acid Hybridization. Journal Genome Research. 4. pp 357-362.
Matrat, S., A. Aubry, C. Mayer, V. Jarlier, and E. Cambau. 2008. Mutagenesis in the α3α4 GyrA Helix and in the Toprim Domain of GyrB Refines the Contribution of Mycobacterium tuberculosis DNA Gyrase to Intrinsic Resistance to Quinolones. Journal Antimicrobial Agents and
Chemotherapy. 52(8): 2909-2914.
McPherson, M. dan S. Moller. 2006. PCR. Edisi 2. New York: Taylor and Francis Group. 1-30.
Navarro, E., G. S. Heras, M. J. Castano, dan J. Solera. 2015. Real-Time PCR Detection Chemistry. Journal Clinica Chimica Acta. 439 (1). pp 231-250.
NCBI. 2018. Mycobacterium tuberculosis H37Rv Complete Genome.Available at: www.ncbi.nlm.nih.gov. (Cited 13 September 2018).
Nugrahaeni, D. K. dan U. S. Malik. 2015. Analisis Penyebab Resistensi OAT (Obat Anti Tuberkulosis). Jurnal Kesehatan Masyarakat. 11(1): 8-15.
Rinanda, T. 2015. Kajian Molekuler Mengenai Mekanisme Resistensi
Mycobacterium tuberculosis, Jurnal Kedokteran Syiah Kuala. 15(3): 162167.
Rychlik, W. 2010. OLIGO Primer Analysis Software Version 7. USA: Molecular Biology Insights, Inc.
Sasmitha, L. V. 2017. Eksplorasi Mutasi Gen gyrA sebagai Penanda
Extensively Drugs Resistant Tuberculosis pada Isolat Klinik Multidrug-Resistant Tuberculosis di Bali menggunakan Metode PCR (Polymerase Chain Reaction).
Skripsi. Program Studi Farmasi, FMIPA, Universitas Udayana.
Walker, J. M. and Rapley, R. 2005. Medical Biomethods of Handbook. New
Jersey: Humana Press Inc.
World Health Organization. 2018. Global of Tuberculosis Report 2018. Prancis: WHO.
Yilmaz, L. S., S. Parnerkar, dan DD. R. Noguera. 2011. mathFISH. a Web Tool That Uses ThermodynamicsBased Mathematical Models for In
Silico Evaluation of Oligonucleotide Probes for Fluorescence In Situ Hybridization. Journal Applied and Environmental Microbiology. 77(3). pp 1118-1122.
Zhang, Z., J. Lu, Y. Wang, Y. Pang, dan Y. Zhao. 2014. Prevalence and Molecular Characterization of Floroquinolone-Resistant Mycobacterium tuberculosis Isolates in China, Jornal ASM org. 58(1): 364-369.
50
Discussion and feedback