Identifikasi Burr Cell dalam Eritrosit Menggunakan Region Properties pada Citra Mikroskop
on
MERPATI VOL.6 NO.1 APRIL 2018
ISSN: 2252-3006
Identifikasi Burr Cell dalam Eritrosit Menggunakan Region Properties pada Citra Mikroskop
Putu Andre Mahaputra, I Made Suwija Putra
Program Studi Teknologi Informasi, Fakultas Teknik, Universitas Udayana
Bukit Jimbaran, Bali, Indonesia, telp. (0361) 701806
e-mail: andremahaputra6868@yahoo.co.id , putrasuwija@unud.ac.id
Abstrak
Uremia adalah sindroma klinik yang terjadi pada semua organ akibat penurunan fungsi ginjal. Secara laboratoris, uremia ditandai dengan peningkatan kadar ureum dan kreatinin dalam darah. Kadar ureum yang tinggi akan menyebabkan perubahan morfologi pada eritrosit. Oleh sebab itu salah satu cara untuk mendiagnosis penyakit ini adalah dengan melakukan pemeriksaan apusan darah tepi untuk melihat ada tidaknya kelainan morfologi eritrosit yaitu burr cell. Hasil pemeriksaan berupa citra ini kemudian diagnosis oleh ahli patologi klinik atau dokter untuk mendapatkan hasil dengan cara manual. Berdasarkan hal tersebut penelitian ini dilakukan untuk mengimplementasikan deteksi secara visual ke dalam komputer yang dapat membantu dokter maupun tenaga laboratorium untuk mendeteksi penyakit uremia berdasarkan citra hasil apusan darah tepi. Aplikasi deteksi burr cell menggunakan Region Properties Filtering sebagai ekstraksi ciri penyakit uremia dan sekaligus pendeteksi dari citra tersebut. Citra yang diuji sebanyak 6 buah citra eritrosit dengan hasil pengujian aplikasi mendapatkan rata-rata akurasi sebesar 89,13%.
Kata Kunci: Uremia, Eritrosit, Burr Cell, Region Properties, Filtering
Abstract
Uremia is a clinical syndrome that occurs in all organs due to decreased renal function. In laboratory, uremia is characterized by elevated levels of urea and creatinine in the blood. High levels of urea will cause morphological changes in erythrocytes. Therefore, one way to diagnose this disease is to examine the peripheral blood smear to see whether there is an erythrocyte morphological abnormality that is burr cell. The results of the examination in the form of this image then diagnosis by clinical pathologists or doctors to get the results manually. Based on this study was conducted to implement detection visually into the computer that can help doctors and laboratory personnel to detect the disease of uremia based on the peripheral blood smear image. Application of burr cell detection using Region Properties Filtering as extraction characteristic of uremia disease and also detection of the image. The image tested as many as 6 pieces of erythrocyte images with the results of application testing get an average accuracy of 89.13%.
Keyword: Uremia, Erythrocytes, Burr Cell, Region Properties, Filtering
Uremia adalah suatu sindrom klinis dan laboratorik yang terjadi pada semua organ akibat penurunan fungsi ginjal, dimana terjadi retensi sisa pembuangan metabolisme protein, yang ditandai dengan peningkatan kadar ureum. Uremia lebih sering terjadi pada Gagal Ginjal Kronis (GGK) [1]. Penyakit Gagal Ginjal kronik merupakan penyakit yang sangat berbahaya karena penyakit ini dapat berlangsung lama dan mematikan. The United States Renal Data System (USRDS) mencatat bahwa jumlah pasien yang dirawat karena end stage renal disease (ESRD) atau gagal ginjal kronis global diperkirakan 3.010.000 pada tahun 2012 dengan tingkat pertumbuhan 7%. Perhimpunan Nefrologi Indonesia (PERNEFRI) tahun 2012 menyatakan bahwa Tingginya prevalensi gagal ginjal kronis juga terjadi di Indonesia, karena angka ini dari tahun ke tahun terus mengalami kenaikan. Jumlah penderita gagal ginjal kronis di Indonesia pada tahun 2011 tercatat 22.304 dengan 68,8% kasus baru dan pada tahun 2012 meningkat menjadi 28.782 dengan 68,1% kasus baru [2]. Secara laboratoris, uremia ditandai dengan
peningkatan kadar ureum dan kreatinin dalam darah. Salah satu tanda akibat uremia dan juga komplikasi penyakit ginjal kronik adalah perubahan morfologi eritrosit. Sebagai salah satu diagnosis penyakit uremia, apusan darah tepi dibuat dan dibaca dengan menggunakan mikroskop untuk melihat adanya salah satu kelainan morfologi eritrosit yaitu burr cell [3] [4].
Metodologi penelitian Aplikasi Identifikasi Burr Cell dalam Eritrosit Menggunakan Region Properties pada Citra Mikroskop yang terdiri dari 8 tahapan yaitu melakukan studi literatur dengan mencari teori-teori penunjang pembuatan aplikasi melalui (buku, jurnal, artikel dan laporan penelitian), identifikasi masalah, penentuan tujuan, perancangan solusi, demo, pengujian, pembahasan, dan kesimpulan.
Aplikasi Identifikasi Burr Cell dalam Eritrosit Menggunakan Region Properties pada Citra Mikroskop merupakan suatu aplikasi pengenalan citra medis berupa sel darah merah (eritrosit) yang dihasilkan dari pengamatan mikroskop untuk mengidentifikasi adanya Burr Cell di dalam citra tersebut dengan menggunakan algoritma Region Properties, guna untuk medeteksi adanya kemungkinan mengidap penyakit Uremia. Gambaran umum pada penelitian mengenai Identifikasi Identifikasi Burr Cell dalam Eritrosit Menggunakan Region Properties pada Citra Mikroskop dapat dilihat pada Gambar 1 di bawah ini.
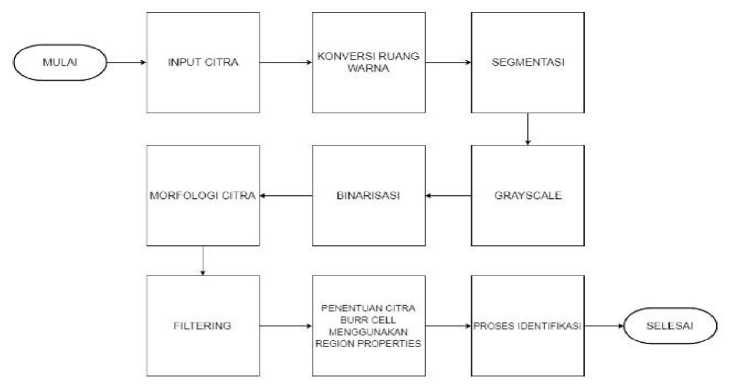
Gambar 1 Gambaran umum aplikasi
Tahap awal sistem dimulai dengan melakukan input citra yaitu mengambil citra mikroskop yang telah disiapkan yaitu citra sel eritrosit dengan format citra .jpg, Citra yang sudah diinputkan tersebut dilakukan proses konversi ruang warna citra menggunakan ruang warna L*a*b guna membuat warna citra bisa terlihat jelas, Citra yang sudah mendapatkan proses konversi warna dilakukan proses segmentasi dengan menggunakan metode K-Means Clustering pada komponen warna a dan b, citra hasil clustering diubah menjadi grayscale agar preprocessing bisa berjalan dengan baik dan diproses binerisasi, Hasil citra binerisasi diberikan proses morfologi citra menggunakan fungsi Imfill dengan tujuan perbaikan citra untuk menutup lubang-lubang objek citra, kemudian dilakukan proses filtering untuk menghilangkan noise pada citra, Citra yang telah mengalami filterisasi sudah bisa dilakukan proses penentuan citra Burr Cell menggunakan region properties, dimana pada proses tersebut memerlukan parameter metric untuk mendapatkan nilai metric pada setiap objek citra yang telah ditandai dengan region properties. Proses identifikasi untuk menentukan Burr Cell dengan Normal Cell, denga menggunakan nilai metric untuk membedakan objek pada citra.
-
3. Kajian Pustaka
-
3.1 Uremia
-
Uremia adalah sindrom klinis yang berhubungan dengan ketidakseimbangan cairan, elektrolit, hormon dan kelainan metabolik, yang berkembang secara paralel dengan penurunan fungsi ginjal [5]. Manifestasi klinis uremia adalah kelelahan, neuropati perifer, penurunan ketajaman penglihatan, kejang, anoreksia dan mual, penurunan indera penciuman dan perasa, gelisah, gangguan tidur, koma, gatal, cegukan dan anemia [6].
Penyebab dari uremia dapat dibagi menjadi tiga, yaitu prerenal, renal, dan post renal. Gagal ginjal merupakan salah satu penyebab Uremia yaitu renal. Uremia renal terjadi akibat gagal ginjal yang dapat menyebabkan gangguan ekskresi urea sehingga urea akan tertahan di dalam darah, hal ini akan menyebabkan intoksikasi oleh urea dalam konsentrasi tinggi yang disebut dengan uremia [2]. Uremia dapat menyebabkan kelainan morfologi eritrosit pada penyakit ginjal kronik. Morfologi eritrosit dapat dilihat dengan apusan darah tepi yang dimana kelainan tersbut dinamakan dengan burr cell [7].
-
3.2 Burr Cell
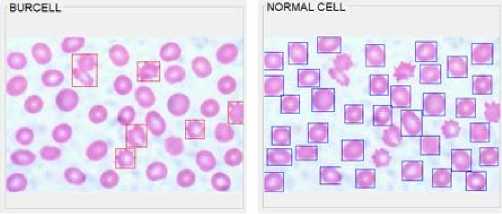
Apusan darah tepi penderita penyakit ginjal kronik akan ditemukan burr cell. Burr cell merupakan salah satu kelainan bentuk eritrosit dimana sel ini mempunyai 10-30 duri-duri pendek yang biasanya memenuhi permukaan eritrosit dan memiliki ukuran yang berbeda-beda. Eritrosit menjadi kasar dan berduri, kadang berbentuk seperti bintang, terlihat seperti Gambar 2.
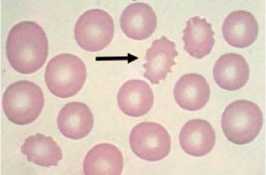
Gambar 2 Burr Cell
Perubahan bentuk ini mungkin karena peroksidasi membran lipid oleh radikal bebas sehingga membran eritrosit menjadi berubah dan akan mempengaruhi bentuk eritrosit [8].
-
3.3 K-Means Clustering
Clustering adalah metode untuk membagi sekumpulan data ke dalam sejumlah kelompok tertentu. k-means clustering adalah salah satu metode clustering yang populer. Dalam k-means clustering, memilah kumpulan data ke dalam kelompok data [9]. Algoritma K-Means adalah metode yang dapat digunakan untuk mengelompokkan objek sebanyak n oleh atribut kesamaan ke dalam sejumlah kelompok k, di mana k < n. Algoritma bekerja dengan mencari jarak terdekat antara centroid dan data latihan berulang. Algoritma K-Means diimplementasikan dengan langkah-langkah berikut.
-
a. Inisialisasi Jumlah klaster.
Dalam langkah ini, tentukan jumlah kelompok atau segmen yang akan dihasilkan. Jumlah ini akan disebut k yang merupakan bilangan bulat positif. Nilai k biasanya ditentukan oleh heuristik atau studi kasus.
-
b. Tentukan posisi Centroid.
Pertama pilih nomor acak dari objek k dari kumpulan data sebagai centroid awal.
-
c. Tentukan Jarak Antara Centroid dan Objek Lainnya.
Semua objek data non-centroid dihitung dalam jarak ke semua centroid. Perhitungan jarak umumnya dilakukan dengan metode jarak Euclidean.
-
d. Klasifikasikan Objek ke Cluster Terdekat.
Setelah satu objek memiliki nilai jarak k, kemudian masukkan objek ke salah satu cluster terdekat antara k.
-
e. Ubah posisi Centroid.
Untuk memperbarui nilai centroid, rumus berikut digunakan.
^k ~ ~ ^?=iXq (1)
Setelah semua centroid diperbarui, lalu dibandingkan dengan nilai centroid sebelumnya. Jika posisinya tidak berubah maka algoritma berakhir. Namun, jika terjadi perubahan posisi centroid, maka kembali ke proses C dan D. Selanjutnya diulangi hingga semua centroid stabil (posisi tidak berubah) [10].
-
3.4 Region Properties
Region properties (regionprops) adalah metode yang digunakan untuk mengukur sekumpulan properti – properti dari setiap region yang telah dilabeli dalam matriks label. Bilangan integer positif yang merupakan elemen dari matriks berkorespondensi dengan region yang bersesuaian. Area, panjang major axis, dan panjang minor axis yang digunakan merupakan sebagian dari properti yang dihasilkan fungsi regionprops.
Daerah yang berdekatan juga disebut objek, komponen yang terhubung, atau
gumpalan. Matriks label yang berisi daerah bersebelahan mungkin terlihat seperti ini:
1 1 0 2 2 0 3 3
1 1 0 2 2 0 3 3
Elemen L sama dengan 1 milik daerah bersebelahan pertama atau komponen yang terhubung; elemen L sama dengan 2 milik komponen terhubung kedua; dan seterusnya. Daerah tidak jelas adalah daerah yang mungkin berisi beberapa komponen yang terhubung. Sebuah matriks label yang berisi daerah yang tidak beraturan mungkin terlihat seperti ini:
1 1 0 1 1 0 2 2
1 1 0 1 1 0 2 2
Elemen L sama dengan 1 milik wilayah pertama, yang tidak jelas dan berisi dua komponen yang terhubung. Elemen L sama dengan 2 milik wilayah kedua, yang merupakan komponen terhubung tunggal.[11]
Sumber data yang digunakan adalah sampel yang diambil dari situs Google Images yaitu dengan melakukan pencarian citra Burr Cell dimana dalam satu citra terdapat dua objek yaitu Burr Cell dan Normal Cell. Citra sampel yang digunakan pada percobaan yaitu berjumlah 5 citra dan 1 citra yang merupakan Normal Cell (tidak mengalami Burr Cell) dapat dilihat pada Gambar 3.
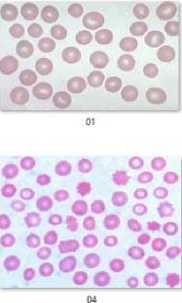
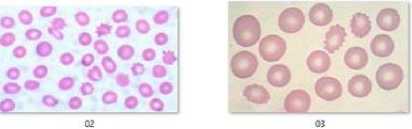
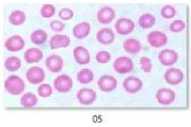
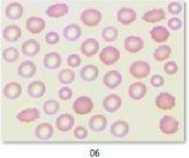
Gambar 3 Sampel data dinama no 01 merupakan eritrosit normal dan no 02-06 eritrosit dengan burr cell
Proses pembuatan Aplikasi Identifikasi Burr Cell dalam Eritrosit Menggunakan Region Properties pada Citra Mikroskop menggunakan bahasa pemrograman Matlab dengan versi MATLAB R2017a. Berikut ini merupakan GUI (Graphic User Interface) dari Aplikasi Identifikasi Burr Cell dalam Eritrosit Menggunakan Region Properties pada Citra Mikroskop.
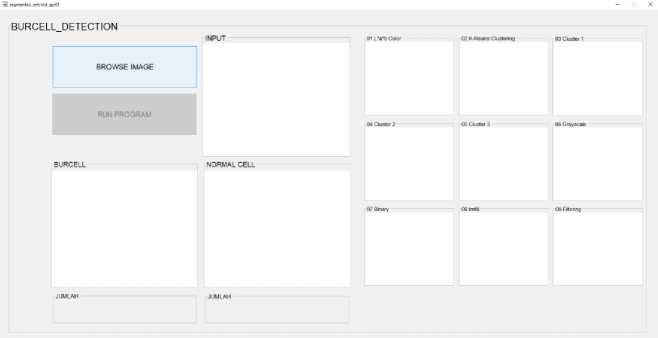
Gambar 4 GUI (Graphic User Interface) Tampilan Awal
Gambar 4.1 merupakan tampilan GUI (Graphic User Interface) dari system Identifikasi Burr Cell Menggunakan Region Properties Pada Citra Mikroskop, dimana pada tampilan ini terdapat 1 Axes untuk menampilkan citra asli, 9 Axes untuk menampilkan hasil proses segmentasi, preprocessing dan morfologi, 2 Axes untuk menampilkan citra hasil proses yaitu diteksi Burr Cell dan Normal Cell, 2 button dan 2 statistic text untuk menampilkan jumlah Burr Cell dan Normal Cell.
-
4.2.1 Open Image
Berikut merupakan kode program yang digunakan untuk menjalankan perintah jika user memilih button BROWSE IMAGE, yaitu digunakan untuk memilih citra asli yang ingin diidentifikasi. Output yang dihasilkan jika user memilih BROWSE IMAGE dan memilih citra yang akan diuji, adalah citra akan tampil di kolom input seperti pada Gambar 5.
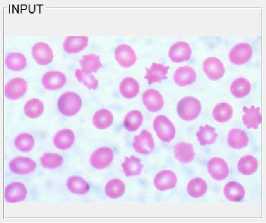
Gambar 5 Tampilan citra input yang akan di uji
Ruang Warna L*a*b* (CIELAB) adalah ruang warna yang paling lengkap yang ditetapkan oleh Komisi Internasional tentang illuminasi warna. LAB singkatan dari Luminance (atau kecerahan) dan A dan B (yang merupakan komponen berwarna). Menurut model ini A berkisar dari hijau ke merah, dan rentang B dari biru menjadi kuning [12]. Konversi Warna L*a*b merupakan proses yang paling pertama dilakukan sebelum proses segmentasi dilakukan karena warna citra terlebih dahulu harus dibuat berbeda dan tampak lebih kontras, agar dalam proses segmentasi bisa mendapatkan hasil yang lebih baik.
Dalam Matlab untuk merubah warna citra menjadi Warna L*a*b dilakukan dengan menngunakan fungsi makecform untuk membuat struktur transformasi warna yang mendefinisikan konversi ruang warna kedalam ruang warna srgb2lab. srgb2lab merupakan fungsi yang digunakan melakukan konversi warna L*a*b pada citra asli.

Gambar 6 Output konversi warna L*a*b
Gambar 6 merupakan output yang dihasilkan oleh proses konversi warna L*a*b dimana citra asli dirubah menjadi warna dengan komponen L (light), a (hijau-merah) dan b (biru-kuning).
-
4.2.3 K-Means Clustering
Proses segmentasi pada aplikasi ini menggunakan metode K-Means Clustering. K-means clustering merupakan metode yang paling populer digunakan untuk mendapatkan deskripsi dari sekumpulan data dengan cara mengungkapkan kecenderungan setiap data lainnya [13]. Dengan K-Means ini diharapakan dihasilkan citra output yang sudah tersegemnetasi menurut cluster nya.
%variabel ab digunakan untuk menyimpan nilai komponen warna a dan b
%variabel nrows dan ncols digunakan untuk menyimpan nilai ukuran pada %citra ab dimana nrows sebagai baris dan ncols sebagai kolom ab = double(lab(:,:,2:3));
nrows = size(ab,1);
ncols = size(ab,2);
%fungsi reshape digunakan untuk mendapatkan nilai pixel dari citra ab %dimana nilai pixel disimpan dalam bentuk array pada baris berjumlah %nrows*ncols dan kolom berjumlah 2
ab = reshape(ab,nrows*ncols,2);
%kluster yang digunakan berjumlah 3
nColors = 3;
%proses segmentasi dengan K-Means Clustering menggunakan metode %Euclidean
Distance untuk menghitung jarak nilai pixel
[cluster_idx, cluster_center] = kmeans(ab,nColors,'distance','sqEuclidean',
...
'Replicates',3);
%merubah label matrix menjadi citra RGB
pixel_labels = reshape(cluster_idx,nrows,ncols);
RGB = label2rgb(pixel_labels);
%menampilkan hasil clustering pada axes G3
axes(handles.G3);
imshow(RGB,[]);
% menggunakan data pixel_labels, lalu memisahkan objek dalam citra
%berdasarkan warna, yang akan menghasilkan 3 gambar.
segmented_images = cell(1,2,3);
rgb_label = repmat(pixel_labels,[1 1 3]);
%menggunakan perulangan sebanyak jumlah kluster for k = 1:nColors
color = Img;
color(rgb_label ~= k) = 0;
segmented_images{k} = color;
Kode Program 1 Fungsi K-Means Clustering
Kode Program 1 merupakan kode program yang berguna untuk melakukan segmentasi yaitu menggunakan metode K-Means Clustering. Jumlah kluster adalah k=3 dimana akan ditampilkan 4 citra yaitu citra hasil clustering dari komponen warna a dan b, citra hasli clustering pertama, kedua dan ketiga.

Gambar 7 Hasil Clustering K-Means
Gambar 7 Hasil Clustering K-Means merupakan hasil dari proses segmentasi dimana terdapat 4 citra yaitu citra clustering dari komponen warna a dan b, lalu hasil dari masing-masing kluster yaitu cluster 1, cluster 2 dan cluster 3. Citra yang digunakan sebagai parameter untuk proses selanjutnya adalah cluster 2.
-
4.2.4 Grayscale dan Binerisasi
Tahapan selajutnya adalah merubah citra menjadi Grayscale dan Binerisasi. Grayscale adalah suatu citra dimana nilai dari setiap pixel merupakan sample tunggal. Format ini sangat membantu dalam pemrograman karena manipulasi bit yang tidak terlalu banyak [14]. Grayscale membantu untuk menyederhanakan warna yang ada pada citra. Dalam Matlab proses merubah citra hasil segmentasi sebelumnya menjadi Grayscale dengan memanggil fungsi rgb2gray di dalam Matlab. Citra cluster 2 yang berwarna sebagai input proses Grayscale diproses sehingga menghasilkan warna keabuan dan bisa diproses ke tahap binerisasi.
Binerisasi adalah sebuah proses transformasi citra keabuan menjadi citra hitam dan putih dengan menggunakan nilai ambang batas (threshold) tertentu [15]. Fungsi dari binerisasi sendiri adalah untuk mempermudah proses pengenalan pola, karena pola akan lebih mudah terdeteksi pada citra yang mengandung lebih sedikit warna [16]. Dalam Matlab fungsi yang digunakan untuk merubah citra menjadi citra biner adalah dengan memanggil fungsi. Imbinerize.

a

b
Gambar 8 a)Hasil Grayscale, b) Hasil Binerisasi
Gambar 8a merupakan output yang dihasilkan pada proses Grayscale dan Gambar 8a Hasil dari proses binerisasi yang dilakukan pada citra Grayscale yang terdapat didalam variabel Ig dengan nilai threshold 0.0.
Proses selanjutnya adalah melakukan proses morphologi citra dimana pada aplikasi ini menggunakan Dilasi dan Imfill agar citra objek yang memiliki lubang dapat tertutup. Dilasi digunakan untuk mempertebal objek dengan menambahkan 1 piksel pada batas antar objek dan Imfill bertujuan untuk mengisi 1 piksel dalam objek yang kosong. Implementasi Dilasi dan Imfill dengan memanggil fungsi yang ada Matlab yaitu imdilate dan mengisi lubang didalam wilayah objek piksel 1 dengan memanggil fungsi yang ada Matlab yaitu imfill. Median filtering memberikan hasil yang sangat bagus untuk citra yang terkena noise impluse bipolar dan unipolar [17]. Noise dalam pengolahan citra adalah piksel atau gambar yang dapat menganggu kualitas citra [18]. Dalam Matlab median filtering digunakan dengan memanggil fungsi medfilt2. Hasil dari proses morphologi dan filtering dapat di lihat pada Gambar 9.
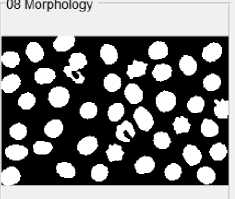
a.

b.
Gambar 9 a)Hasil Morphologi Citra, b) Hasil Filtering
Region properties (regionprops) adalah metode yang digunakan untuk mengukur sekumpulan properti – properti dari setiap region yang telah dilabeli dalam matriks label [17]. Region properties digunakan untuk membantu dalam proses penetuan nilai centroid dan penentuan daerah bounding box [19].
|
%menjalankan fungsi bwboundaries yaitu mengembalikan matriks objek dan lubang diberi label. |
label L di %mana |
|
[segmented_images{2},L] = bwboundaries(K,'noholes'); | |
|
%membuat variabel bursel dan normal untuk menyimpan nilai yang %digunakan | |
|
untuk menghitung objek Burr Cell dan Normal Cell yang telah % |
diberi label |
|
menggunakan region properties | |
|
bursel = 0; | |
|
normal = 0; | |
|
%regionprops digunakan untuk menjalankan fungsi region properties | |
|
stats = regionprops(L,'All'); | |
|
%menyiapkan sebuah wadah matrix menggunakan variabel bw2, lal |
u %menggunakan |
|
bwlabel untuk mengembalikan label matrix num yang berisi %label untuk 8- | |
|
connected object yang ditemukan dalam BW | |
|
bw2 = zeros(size(Img,1),size(Img,2)); | |
|
[~,num] = bwlabel(bw2,8); | |
|
%Membuat variabel untuk menyimpan data citra asli namun nilai |
matrix %telah di |
|
set menjadi 0 | |
|
RGB2 = Img; | |
|
RGB3 = Img; | |
|
RGB4 = Img; | |
|
RGB5 = Img; | |
|
%sebuah perulangan yang dilakukan sepanjang citra didalam variabel | |
|
%segmented_images{2} lalu digunakan untuk proses klasifikasi |
%menggunakan |
|
parameter metric | |
|
for k = 1:length(segmented_images{2}) | |
|
boundary = segmented_images{2}{k}; | |
|
delta_sq = diff(boundary).^2; | |
|
perimeter = stats(k).Perimeter; | |
|
area = stats(k).Area; | |
|
metric = 4*pi*area/perimeter^2; | |
bbox = stats(k).BoundingBox;
eccentricity = stats(k).Eccentricity;
MajorAxisLength = stats(k).MajorAxisLength;
MinorAxisLength = stats(k).MinorAxisLength;
d = MajorAxisLength/MinorAxisLength;
position = [boundary(1,2),boundary(1,1)];
%sebuah kondisi yang digunakan untuk melakukan klasifikasi yaitu %menentukan Burr Cell dan Normal Cell pada citra
%menggunakan fungsi insertObjectAnnotation untuk memberi label berupa
%rectangle berwarna merah kepada objek yang dinyatakan sebagai Burr %Cell yang
disimpan kedalam variabel RGB4
RGB4 =
insertObjectAnnotation(RGB4,'rectangle',bbox,'','FontSize',10,'LineWidth',2,'C olor',{'red'});
%menghitung jumlah objek Burr Cell
bursel = bursel+1;
%namun jika nilai metric pada masing-masing objek yaitu lebih dari 0,88 %maka objek tersebut dinyatakan sebagai Normal Cell
elseif metric >= 0.88
bw2(L==k) = 1;
%menggunakan fungsi insertObjectAnnotation untuk memberi label berupa %rectangle berwarna biru kepada objek yang dinyatakan sebagai Normal %Cell yang disimpan kedalam variabel RGB5
RGB5 =
insertObjectAnnotation(RGB5,'rectangle',bbox,'','FontSize',10,'LineWidth',2,'C olor',{'blue'});
%menghitung jumlah objek Normal Cell normal = normal +1 end end
%menampilkan hasil klasifikasi Burr Cell pada axes G11 axes(handles.G11);
imshow(RGB4);
%menampilkan hasil klasifikasi Normal Cell pada axes G12 axes(handles.G12);
imshow(RGB5);
Kode Program 2 Fungsi Region Properties
Kode Program 2 merupakan kode program yang digunakan untuk menjalankan fungsi region properties dimana dengan fungsi region properties, klasifikasi citra bisa dilakukan yaitu memanfaatkan nilai area dan nilai perimeter untuk menggunakan parameter metric. Metric merupakan sebuah besaran yang menunjukkan tingkat kebulatan bentuk suatu objek. Nilai metric = 4π x luas objek/(keliling objek)2. Nilai metric berkisar antara 0 hingga 1. Semakin bulat suatu objek, maka nilai metric-nya semakin mendekati 1 [20]. Dengan rumus 4π x area/perimeter^2 untuk mendapatkan nilai pada objek (Burr Cell), Parameter metric digunakan untuk membuat suatu kondisi dimana jika nilai objek kurang dari 0,88 maka dapat objek tersebut dinyatakan Burr cell, namun jika sebaliknya maka objek tersebut adalah Normal Cell.
Gambar 10 Hasil Proses region properties
Gambar 10 merupakan hasil yang didapat dari fungsi region properties yang digunakan untuk proses klasifikasi menggunakan parameter metric dengan memanfaatkan nilai metric pada masing-masing objek lalu dilakukan suatu kondisi dimana jika nilai metric kurang dari 0,88 maka objek dinyatakan Burr Cell, namun jika sebaliknya maka dinyatakan sebagai Normal Cell. Proses yang terakhir adalah menghitung jumlah Burr Cell dan Normal Cell yang telah diberi label menggunakan Region Properties. Perhitungan jumlah masing-masing sel ini didapatkan dengan membuat variabel jumlah_label burrcell untuk menyimpan data string yang digunakan untuk jumlah objek Burr Cell yang ada pada variabel bursel dan membuat variabel jumlah_label normal untuk menyimpan data string yang digunakan untuk jumlah objek Normal Cell yang ada pada variabel normal. Objek dihitung sesuai dengan label yang diberikan oleh fungsi region properties, dengan begitu peneliti dapat mengetahui jumlah Burr Cell dan Jumlah Normal Cell dengan baik.
Hasil uji coba adalah proses untuk mendapatkan hasil akurasi ketepatan Aplikasi Identifikasi Burr Cell dalam Eritrosit Menggunakan Region Properties pada Citra Mikroskop dalam melakukan deteksi Burr Cell pada citra Eritrosit. Perhitungan akurasi dilakukan dengan membandingkan jumlah Burr Cell yang didapat dari aplikasi dengan jumlah Burr Cell yang didapat dari hasil pengamatan mata dari citra yang diujikan. Dengan persamaan sebagai berikut.
, . rn,∖ Yburrcellterdeteksi
(2)
akurasi (.%J = =----------------x 100%
ZtburrcβU pengamatan
Dari Persamaan 2 tersebut hasil uji coba ke enam citra uji dapat dilihat pada Tabel 1.
|
No. |
Citra Deteksi Burr Cell |
Citra Deteksi Normal Cell |
Jumlah Burr Cell |
Jumlah Burr Cell Terdeteksi |
Status |
Akurasi Program | |
|
1 |
BURC ELX OOo ∙β OO ⅛oo^o∙* ocoo ooα |
NORMAL CELL |
0 |
0 |
Benar |
100% | |
|
2 |
3URCELL k⅞o4o∙ o°O O rso ∙ I o*∕<≡Λ∙∙ b oo0oo |
NORMAL CELL- — J1-1 1 □ -l∣ ,α |
6 |
6 |
Benar |
100% | |
|
3 |
r BURCELL--- A 0‰0 z⅛⅛0 ^⅛ooθ |
NORMAL CELL ⅛□□u |
3 |
2 |
Salah |
80,1% | |
|
4 |
BURCELL oθ-ow aoo .¾A√⅛ i¾So.⅛∙ *θ⅞⅜* a OO |
NORMAL CELL 1 π⅛Bπ ⅛≡¾κS |
8 |
6 |
Salah |
89,6% | |
|
5 |
B□RCELL Λ Oo O- ⅛k∙8∙∙ ⅞ |
NORMAL CELL ~=U1 1L = uc□π τ |
2 |
2 |
Benar |
100% |
|
6 |
B□RCELL □ □ o π n n aU π d |
NORMAL CELL H≡h□ ∏⅛P |
6 |
11 |
Salah |
65,1% |
Berdasarkan hasil uji coba sistem Identifikasi Burr Cell Menggunakan Region Properties Pada Citra Mikroskop, maka didapat sebuah kesimpulan bahwa sistem ini dapat melakukan proses identifikasi Burr Cell pada 6 citra eritrosit sebesar 89,13%. Nilai akurasi tersebut didapat dari penjumlahan total nilai persentase akurasi pada setiap citra yang dilakukan uji coba, lalu hasil penjumlahan dibagi 6 maka didapat nilai akurasi program. Nilai akurasi yang di atas 80 % menujukkan bahwa aplikasi ini berhasil dalam melakukan identifikasi Burr Cell dalam Sel Darah Merah (Eritrosit)
Daftar Pustaka
-
[1] T. Deguchi, K. Isozaki, K. Yousuke, T. Terasaki, and M. Otagiri, “Involvement of organic
anion transporters in the efflux of uremic toxins across the blood-brain barrier,” J. Neurochem., pp. 1051–1059, 2006.
-
[2] A. A. Alfonso, A. E. Mongan, and M. F. Memah, “Gambaran Kadar Ureum Serum pada
Pasien Penyakit Ginjal Kronik Stadium 5 Non Dialisis,” J. e-Biomedik, vol. 4, no. 2, 2016.
-
[3] B. J. Bain, “Diagnosis from the Blood Smear,” N. Engl. J. Med., pp. 498–507, 2005.
-
[4] J. F. Lesesve et al., “Comparative Evaluation of Schistocyte Counting by an Automated
Method and by Microscopic Determination,” Am. J. Clin. Pathol., pp. 739–745, 2004.
-
[5] M. B. Rivara, C. H. Chen, A. Nair, D. Cobb, J. Himmelfarb, and R. Mehrotra, “Indication
for Dialysis Initiation and Mortality in Patients With Chronic Kidney Failure: A Retrospective Cohort Study,” Am J Kidney Dis, vol. 69, no. 1, pp. 41–50, 2017.
-
[6] T. W. Meyer and T. H. Hostetter, “Uremia,” N. Engl. J. Med., pp. 1316–1325, 2007.
-
[7] S. Wahyu and I. Vitasari, “Hubungan Kadar Ureum Dengan Anemia Dan Kelainan
Bentuk Eritrosit Pada Penderita Penyakit Ginjal Kronik,” Mandala Heal., vol. 5, no. 1, pp. 87–219, 2011.
-
[8] G. Weiss and L. T. Goodnough, “Anemia of Chronic Disease.,” N. Engl. J. Med., pp.
1011–1023, 2005.
-
[9] N. Dhanachandra, K. Manglem, and Y. J. Chanu, “Image Segmentation Using K -means
Clustering Algorithm and Subtractive Clustering Algorithm,” Procedia Comput. Sci., 2015.
-
[10] A. E. Amalia, G. Airlangga, and A. N. A. Thohari, “Breast Cancer Image Segmentation Using K-Means Clustering Based on GPU Cuda Parallel Computing,” J. Infotel, vol. 10, no. 1, pp. 33–38, 2018.
-
[11] Z. A. Al-balqa and M. H. Al-balqa, “A Methodology to Analyze Objects in Digital Image using Matlab A Methodology to Analyze Objects in Digital Image using Matlab,” vol. 5, no. November, pp. 21–28, 2016.
-
[12] Y. Hapsari and M. F. Hidayattullah, “Deteksi Wajah Dari Berbagai Ras Manusia Menggunakan Warna Kulit Berbasis Ruang Warna L * A * B,” vol. 2013, no. November, pp. 409–414, 2013.
-
[13] A. Padmo A.M and M. Murinto, “Segmentasi Citra Batik Berdasarkan Fitur Tekstur Menggunakan Metode Filter Gabor Dan K-Means Clustering,” J. Inform., vol. 10, no. 1, pp. 1173–1179, 2016.
-
[14] H. Al Fatta, “Konversi Format Citra Rgb Ke Format Grayscale Menggunakan Visual Basic,” vol. 2007, no. November, pp. 1–6, 2007.
-
[15] E. Paulus, “Analisa dan Evaluasi Metode Binerisasi pada Tulisan Tangan Sunda kuno,” J. Tek. Inform., vol. 9, no. 1, pp. 33–37, 2017.
-
[16] M. Hatta and I. G. Susrama, “Counting Sperma Aktif Menggunakan Metode Otsu Threshold dan Local Adaptive Threshold,” Tek. Eng. Sains J., vol. 1, no. 1, pp. 47–54, 2017.
-
[17] C. J. Pramana, “Implementasi Metode Thresholding dan Metode Regionprops untuk
Mendeteksi Marka Jalan Secara Live Video,” J. Ilm. Ilk., vol. 1, no. 1, pp. 1–8, 2015.
-
[18] I. Maulana, P. N. Andono, P. Studi, T. Informatika, F. I. Komputer, and U. D.
Nuswantoro, “Analisa Perbandingan Adaptif Median Filter Dan Median Filter Dalam Reduksi Noise Salt & Pepper,” vol. 2, no. 2, pp. 157–166, 2016.
-
[19] A. Yudhana and S. Saifullah, “Perbandingan Segmentasi Pada Citra Asli dan Kompresi
Wavalet Untuk Identifikasi Telur,” J. Ilm. Ilk., vol. 8, no. Desember, pp. 190–196, 2016.
-
[20] N. Mirnasari and A. Kusworo, “Aplikasi Metode Otsu untuk Indentifikasi Bakteri
Tuberkulosis Secara Otomatis,” Youngster Phys. J., vol. 02, no. 1, pp. 13–20, 2013.
Identifikasi Burr Cell dalam Eritrosit Menggunakan Region Properties pada Citra Mikroskop
(Putu Andre Mahaputra)
12
Discussion and feedback