Brain Tumor Segmentation Based on Magnetic Resonance Imaging Images Using the U-NET Method
on
Majalah Ilmiah Teknologi Elektro, Vol. 19, No. 2, Juli - Desember 2020 DOI: https://doi.org/10.24843/MITE.2020.v19i02.P05
151
Segmentasi Tumor Otak Berdasarkan Citra Magnetic Resonance Imaging Dengan Menggunakan Metode U-NET
Ida Bagus Leo Mahadya Suta1, Made Sudarma2, I Nyoman Satya Kumara3
[Submission: 11-02-2020, Accepted: 13-11-2020]
Abstract—Brain tumor is a deadly disease where 3.7% per 100,000 patients have malignant tumors. To analyze brain tumors can be done through magnetic resonance imaging (MRI) image segmentation. Automatic image analysis process is needed to save time and improve accuracy of doctor diagnoses. Automatic segmentation can be done with deep learning. U-NET is one of the methods used to segment medical images because it works at pixel level. By applying the ReLU and Adam Optimizer activation function, this method can solve the problem of segmenting brain tumors. Dataset for the training and validation process using BRATS 2017. Several hyperparameters are applied to this method: learning rate (lr) = 0.0001, batch size (bz) = 5, epoch = 80 and beta (b1) = 0.9. From a series of processes carried out, accuracy of the U-NET method is calculated by Dice Coefficient formula and results in following accuracy values, during training of 90.22% (Full Tumor), 78.09% (Core Tumor) and 80.20% (Enhancing Tumor).
Intisari— Salah satu penyakit yang paling mematikan adalah tumor otak dengan tingkat kematian mencapai 3.7% per 100.000 pasien. Untuk menganalisa tumor otak dapat dilakukan melalui segmentasi citra Magnetic Resonance Imaging (MRI). Proses analisa citra secara otomatis dibutuhkan untuk menghemat waktu dan meningkatkan akurasi dari diagnosa yang dilakukan. Segmentasi secara otomatis dapat dilakukan dengan deep learning. Khusus pada citra medis metode yang digunakan harus mampu bekerja pada pixel level agar dapat mencapai akurasi yang baik, salah satu metode tersebut adalah metode U-NET. Dengan menerapkan fungsi aktivasi ReLU dan Adam Optimizer, metode ini dapat menyelesaikan permasalahan segmentasi tumor otak. Dataset untuk proses training dan validation menggunakan BRATS 2017. Beberapa hyperparameter diterapkan pada metode ini yaitu, learning rate (lr) = 0.0001, batch size (bz) = 5, epoch = 80 dan beta (b1) = 0.9. Dari serangkaian proses yang dilakukan, akurasi metode U-NET dihitung dengan rumus Dice Coefficient dan menghasilkan nilai akurasi sebagai berikut: 90.22% (Full Tumor), 78.09% (Core Tumor) dan 80.20% (Enhancing Tumor).
Kata Kunci— Tumor Otak, MRI, citra medis, deep learning, U-Net, ReLU, Adam Optimizer, Training, Validation
Petumbuhan sel yang abnormal dan tidak terkendali pada otak merupakan penyebab dari tumor otak. Tumor otak diklasifikasikan menjadi dua jenis, yaitu tumor otak ganas dan tumor otak jinak. Tumor jinak memiliki tingkat pertumbuhan sel yang abnormal lebih lambat jika dibandingkan dengan tumor ganas [1]. Insiden tahunan tumor ganas adalah 3.700 pada laki-laki dan 2.600 pada wanita [1].
Untuk deteksi dini penyakit tumor otak dapat dilakukan melalui pengambilan citra Magnetic Resonance Imaging (MRI). MRI merupakan pilihan yang dianggap terbaik karena tingkat sensitifitasnya sangat tinggi sehingga mampu memberikan informasi yang akurat [2]. Dalam tumor otak mengandung soft tissue dan hard tissue, kedua bagian ini terlihat jelas pada citra MRI [3]. Dokter dapat melakukan diagnosa melalui citra MRI untuk menentukan tindakan medis yang harus dilakukan. Diagnosa tidak dapat dilakukan dalam waktu yang singkat karena dokter harus melakukan pemetaan/segementasi wilayah tumor otak secara manual melalui citra MRI [4]. Untuk mengatasi permasalahan tersebut dibutuhkan segmentasi citra secara otomatis dengan menggunakan bantuan komputer untuk mempersingkat waktu yang dibutuhkan dalam diagnosa penyakit tumor otak. Pengolahan citra digital dengan bantuan komputer memberikan imbas yang besar dalam bidang medis [5].
Kemampuan komputer untuk menyelesaikan permasalahan berdasarkan data yang dimiliki dikategorikan sebagai machine learning (ML). Pada penelitian sebelumnya mengenai segmentasi tumor otak dengan ML dikategorikan menjadi metode un-supervised learning [6]–[13] dan supervised learning [14]–[16]. Kedua metode tersebut dibuat untuk menyelesaikan permasalahan secara khusus, metode ini tidak didesain untuk pembelajaran otomatis dari informasi yang terkandung dalam data. Pembelajaran otomatis dapat diselesaikan dengan Artificial Neural Network (ANN). Terkait dengan penelitian ini metode yang sering digunakan untuk menyelesaikan permasalahan segmentasi dan analisa citra digital adalah metode Convolutional Neural Network (CNN) yang merupakan pengembangan dari metode ANN itu sendiri.
Dalam penelitian sebelumnya [17]–[19] metode CNN berfokus pada jumlah dan ukuran kernel dalam lapisan konvolusi untuk menawarkan akurasi yang baik pada segmentasi gambar biomedis. Dalam penelitian [19], [20] menggunakan ukuran kernel kecil untuk membuat metode ini bekerja dengan baik pada pixel level, tetapi dampak negatifnya adalah waktu penyelesaian yang lebih lama.
Permasalahan yang dihadapi oleh metode CNN konvensional dalam deep learning citra biomedik adalah akurasi dari segmentasi citra yang dipengaruhi oleh ukuran kernel, semakin kecil ukuran kernel maka akurasi semakin
p-ISSN:1693 – 2951; e-ISSN: 2503-2372

baik. Namun, hal tersebut berdampak pada waktu penyelesaian metode relatif lebih lama. Dalam penelitian ini [21] menawarkan metode U-NET CNN yang mampu bekerja pada pixel level dan memanfaatkan fungsi aktivasi ReLU dan berhasil memberikan akurasi yang baik dan waktu penyelesaian yang lebih singkat pada gambar sel mikroskopis. Sehingga, pada penelitian ini akan menggunakan metode U-NET dalam segmentasi citra biomedis MRI tumor otak.
-
A. Tumor Otak
Pertumbuhan sel yang tidak normal dan tidak terkendali pada otak merupakan penyebab tumor otak. Jenis tumor otak yang paling umum ditemukan adalah Glioma yang memiliki tingkat kematian yang tinggi. Glioma dibagi menjadi dua jenis, yaitu High Grade Glioma (HGG) dan Low Grade Glioma (LGG). Umumnya, pasien yang menderita HGG memiliki harapan hidup yang lebih rendah dibandingkan dengan LGG. Biasanya, pasien yang menderita LGG memiliki harapan hidup antara 6 sampai 15 tahun, namun pasien yang menderita HGG hanya memiliki harapan hidup sekitar 15 bulan [22].
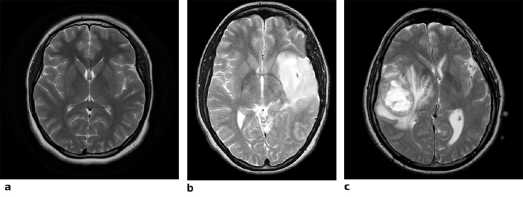
Gambar 1: (a) Otak Normal, (b) Low Grade Glioma, (c) High Grade Glioma
-
B. U-Net Convolutional Neural Network
CNN merupakan pengembangan dari ANN dan terdiri dari neuron yang memiliki bobot, bias dan fungsi aktivasi. CNN memiliki convolution, pooling dan fully connected layer [23]– [26]. Citra input akan menghasilkan output dari proses konvolusi, pada tahapan ini disebut dengan Convolution Layer. Proses konvolusi mengekstrak fitur dari citra input, proses konvolusi menggunakan fungsi aktivasi untuk menentukan jaringan yang aktif atau tidak. Pada penelitian ini fungsi aktivasi yang digunakan adalah fungsi nonlinier ReLU. Aktivasi ReLu (Rectifed Linear Unit) merupakan layer aktivasi yang mengaplikasikan fungsi R(x) = max(0, z) yang artinya fungsi ini melakukan filterisasi dengan nilai nol terhadap nilai bobot pada citra input. Gambar 2 menunjukkan aktivasi ReLU, dimana untuk setiap bobot dengan nilai minus akan dirubah menjadi nilai bobot nol.
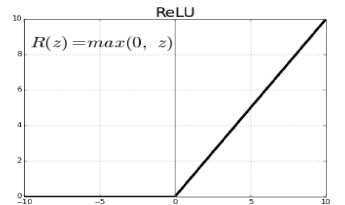
Gambar 2 Fungsi Aktivasi Non-Linier ReLU
Proses selanjutnya adalah pooling layer, Pooling layer memiliki tujuan untuk mengurangi ukuran sebuah citra. Selain itu Pooling juga dapat meningkatkan invariasi posisi dari fitur. Max pooling dan averange pooling adalah metode yang biasa digunakan pada proses ini [23].
Agar dapat diklasifikasikan secara linier, transformasi data satu dimensi dilakukan pada tahapan fully connected layer dengan cara menjumlahkan weight input dan nilai bias kemudian mengaplikasikannya kedalam fungsi aktivasi ReLU. Untuk memperoleh nilai rentang probabilitas fungsi softmax akan diterapkan. Jumlah propabilitas yang dihasilkan sama dengan satu dengan rentang antara 0 sampai 1.
Arsitektur U-Net dibangun di atas Fully Convolutional Network (FCN) dan dimodifikasi sedemikian rupa sehingga menghasilkan segmentasi yang lebih baik dalam pencitraan medis. Dibandingkan dengan FCN, dua perbedaan utamanya adalah (1) U-NET simetris dan (2) koneksi lompatan (skip connection) antara jalur downsampling dan jalur upsampling yang menerapkan operator gabungan daripada penjumlahan. Skip connection ini bertujuan untuk memberikan informasi lokal ke informasi global selama upsampling. Karena simetris, jaringan memiliki sejumlah besar feature map di jalur upsampling, yang memungkinkan untuk mentransfer informasi. Sebagai perbandingan, arsitektur FCN dasar hanya memiliki sejumlah peta fitur kelas di jalur upsampling-nya.
128 64 64
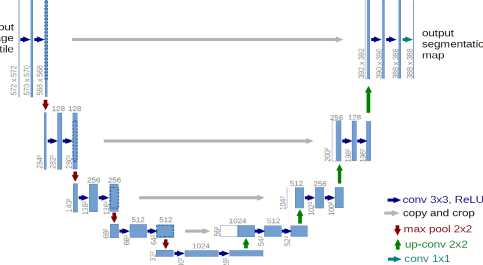
Gambar 3. Arsitektur U-NET
Sumber: Diambil dari [21]
Gambar 3 menggambarkan arsitektur U-NET yang dibagi menjadi 3 bagian:
-
1. Jalur contracting/downsamling
-
2. Bottleneck
-
3. Jalur expanding/upsampling
Contracting atau downsamling terdiri dari 4 blok, dan setiap blok tersusun atas
-
• 3x3 Convolution Layer + fungsi aktivasi
-
• 3x3 Convolution Layer + fungsi aktivasi
-
• 2x2 Max Pooling
Perlu diperhatikan bahwa jumlah feature map akan berlipat ganda untuk setiap pooling, dimulai dengan 64 feature map pada blok pertama, 128 untuk blok kedua dan seterusnya. Contracting bertujuan untuk menangkap konteks gambar input agar dapat melakukan segmentasi. Informasi kontekstual kasar ini kemudian akan ditransfer ke jalur upsampling dengan cara skip connection.
DOI: https://doi.org/10.24843/MITE.2020.v19i02.P05
Bottleneck adalah bagian yang terdapat di antara jalur upsampling dan downsampling. Bottleneck dibangun dari hanya 2 convolutional layer (dengan batch normalization).
Expanding atau Upsampling memiliki tujuan untuk memungkinkan lokalisasi yang tepat dikombinasikan dengan informasi kontekstual dari jalur contracting. Terdiri dari 4 blok dan setiap blok terdiri dari:
-
• Deconvolution layer dengan 2 stride
-
• Penggabungan dengan feature map yang dipotong dari jalur contracting
-
• 3x3 Convolution Layer + fungsi aktivasi (dengan
normalisasi batch)
-
• 3x3 Convolution Layer + fungsi aktivasi (dengan
normalisasi batch)
-
A. Sumber Data
Penelitian ini menggunakan data primer yang berasal dari Multi Brain Tumor Image Segmentation (BRATS) 2017. BRATS 2017 memiliki citra MRI dari 210 pasien yang menderita tumor otak HGG dan 75 pasien tumor otak LGG yang akan dibagi menjadi 20% data validation dan 80% data training. Data multimodal MRI tersedia dengan 4 urutan pemindaian untuk setiap pasien, yaitu T1-weighted (T1), citra T1-weighted dengan penambahan kontras gadolinium (T1c), T2-weighted (T2) dan Fluid Attenuated Inversion Recovery (FLAIR). Selain itu, segmentasi manual dengan empat kelas intra-tumoral tersedia untuk setiap pasien; (1) necrosis, (2) edema, (3) non-enhancing, (4) enhancing tumor. Segmentasi manual ini digunakan sebagai dasar kebenaran pengujian (ground truth) dalam segmentasi model pada saat training maupun pada saat validation.
-
B. Gambaran Umum Sistem
Diagram blok berikut adalah gambaran umum dari alur proses sistem diagnosa tumor otak berdasarkan citra MRI yang dapat dilihat pada gambar 4.
Gambar 4. Gambaran Umum Sistem
Pada alur diagram diatas, dapat dilihat sebuah citra MRI sebelum diproses oleh algortima U-NET akan melalui beberapa tahapan terlebih dahulu. Diawali dengan proses bias correction N4ITK untuk menghilangkan noise yang ada pada citra MRI, tahapan ini sangat berguna untuk meminimalisir kesalahan pada proses klasifikasi. Kemudian dilakukan proses data augmentation untuk memperbanyak dataset, dalam penelitian ini proses augmentation yang dilakukan dengan flip vertical dan flip horizontal. Algortima U-NET akan melakukan proses pembelajaran melalui fase training dan
153 validation, sehingga menghasilkan Data Model. Data model adalah kumpulan dataset citra MRI tumor otak yang telah diolah sebelumnya oleh algoritma U-NET, untuk menghasilkan kumpulan data array yang tersimpan sebagai data pembelajaran. Selanjutnya, algortima U-NET akan mensegmentasi citra inputan dan menentukan wilayah tumor otak.
Selama pengujian berlangsung akurasi dan loss akan dihitung setiap perulangan (epoch) dengan menggunakan rumus Dice Coefficient. Dice Coefficient merupakan ukuran overlap antara dua sampel. Ukuran ini berkisar dari 0 sampai 1, dimana Dice Coefficient bernilai 1 adalah yang terbaik. Fungsi ini awalnya dikembangkan untuk data biner dan dapat dihitung berdasarkan rumus berikut:
Dimana ∣Λ ∩ B| mewakili elemen antara set A dan B, ∣Λ∣ mewakili jumlah elemen dalam set A dan |B| mewakili jumlah elemen dalam set B. Sedangkan untuk menghitung loss atau kesalahan yang dihasilkan oleh model ini, kami menggunakan fungsi Soft Dice Coefficient yang menggunakan perhitungan 1 - Dice.
Pengujian dilakukan dengan menggunakan dataset BRATS 2017, yang dibagi menjadi dataset training dan validation dengan rasio 80:20. Pada algortima U-Net yang akan diujikan, terdapat beberapa hyperparameter dan juga parameter pada fungsi optimasi Adam Optimizer. Adapun nilai pada hyperparameter adalah sebagai berikut, learning rate (lr) = 0.0001, batch size (bz) = 5, epoch = 80 dan beta (b1) = 0.9.
Matrik evaluasi dibagi menjadi tiga wilayah tumor yang bernama a) Complete/Full Tumor (necrosis, edama, enhancing dan non enhancing tumor), wilayah kedua adalah Core Tumor (sama seperti Complete Tumor tanpa edama) dan yang terakhir adalah Enhancing Tumor.
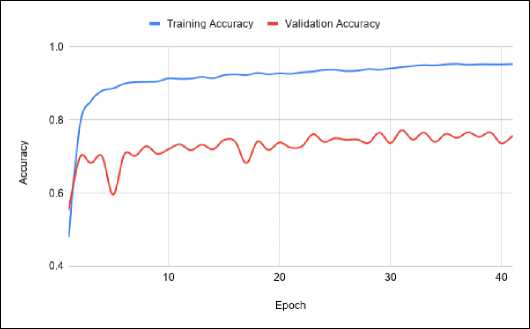
Gambar 5. Grafik Perbandingan Akurasi Fase Training dan Validation Pada Proses Segmentasi Full Tumor
Ida Bagus Leo Mahadya Suta: Segmentasi Tumor Otak Berdasarkan…
p-ISSN:1693 – 2951; e-ISSN: 2503-2372
.

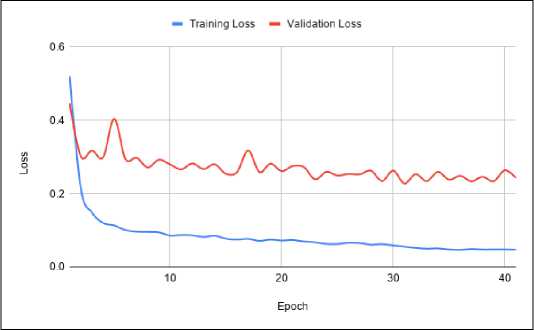
Gambar 6. Grafik Perbandingan Loss Fase Training dan Validation Pada Proses Segmentasi Full Tumor
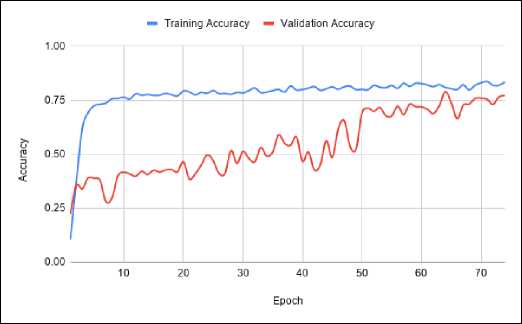
Gambar 9. Grafik Perbandingan Akurasi Fase Training dan Validation Pada Proses Segmentasi ET Tumor
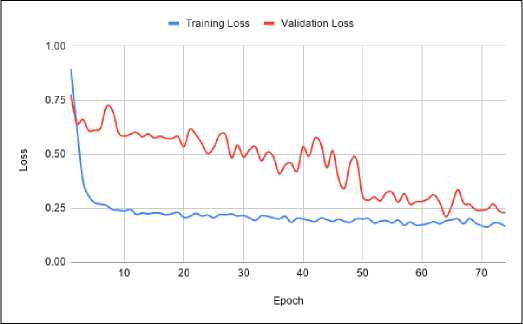
Gambar 10. Grafik Perbandingan Loss Selama Fase Validation dan Testing Pada Proses Segmentasi ET Tumor
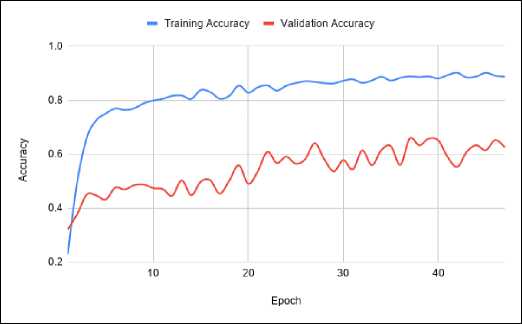
Gambar 7. Grafik Perbandingan Akurasi Fase Training dan Validation Pada Proses Segmentasi Core Tumor
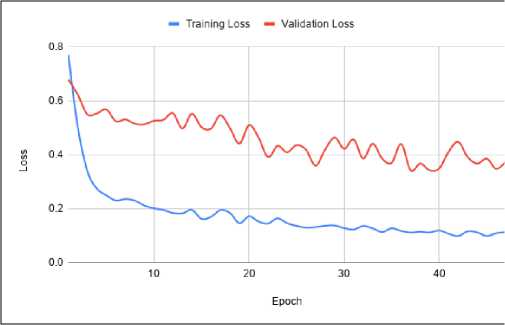
Gambar 8. Grafik Perbandingan Loss Fase Training dan Validation Pada Proses Segmentasi Core Tumor
Berdasarkan Gambar 5 - 10, menunjukkan bahwa grafik dari model U-Net tidak menunjukkan kondisi overfitting maupun nderfitting, melainkan menuju pada grafik yang menuju onvergen. Underfitting merupakan kejadian dimana model ang telah terbentuk tidak mampu untuk melihat logika dibalik dataset, sehingga model tidak dapat melakukan prediksi engan hasil yang akurat pada data training maupun data alidation. Sedangkan Overfitting terjadi ketika model yang ibuat terlalu fokus pada training dataset tertentu, sehingga idak bisa melakukan prediksi dengan benar apabila diberikan dataset lain yang serupa. Dalam penelitian ini jumlah epoch = 0, namun karena keterbatasan perangkat, peneliti menerapkan arly stop monitor. Early stop monitor merupakan kondisi dimana fase training dihentikan sebelum keseluruhan epoch selesai. Kondisi yang diterapkan adalah, apabila dalam setiap 10 epoch terjadi penurunan akurasi maka fase training akan dihentikan.
Ketika proses pembelajaran segmentasi full tumor, metode U-NET tidak mengalami peningkatan akurasi pada epoch ke-47, sehingga U-NET berhenti pada epoch tersebut. Kondisi yang sama ditunjukkan juga ketika melakukan proses pembelajaran pada wilayah core tumor yang tidak mengalami peningkatakan akurasi pada epoch ke-47 yang menyebabkan proses pembelajaran terhenti pada epoch tersebut. Pada enhancing tumor terhenti pada epoch ke-73. Proses selanjutnya dengan melakukan evaluasi masing masing model yang
Majalah Ilmiah Teknologi Elektro, Vol. 19, No. 2, Juli - Desember 2020
DOI: https://doi.org/10.24843/MITE.2020.v19i02.P05 155
dihasilkan dengan menggunakan fungsi model.evaluate() Berdasarkan Tabel I, proses segmentasi Full Tumor sehingga mengetahui rata-rata akurasi dan loss yang dihasilkan. memperoleh hasil akurasi yang paling tinggi dibandingkan dengan yang lainnya dikarenakan area tumornya paling besar dan paling jelas dibandingkan dengan area tumor lainnya. Gambar 11 menunjukkan hasil prediksi segmentasi tumor otak yang dibandingkan dengan segmentasi manual dari dokter. Dapat dilihat bahwa hasil segmentasi otomatis memberikan kemiripan yang tinggi dengan segmentasi manual.
TABEL I
UHasil Akurasi Dan Loss Selama Penelitian
|
Segmentasi |
Epoch |
Accuracy |
Loss |
|
Full Tumor |
41 |
90.22% |
0.1 |
|
Core Tumor |
47 |
78.09% |
0.22 |
|
Enhancing Tumor |
74 |
80.20% |
0.2 |
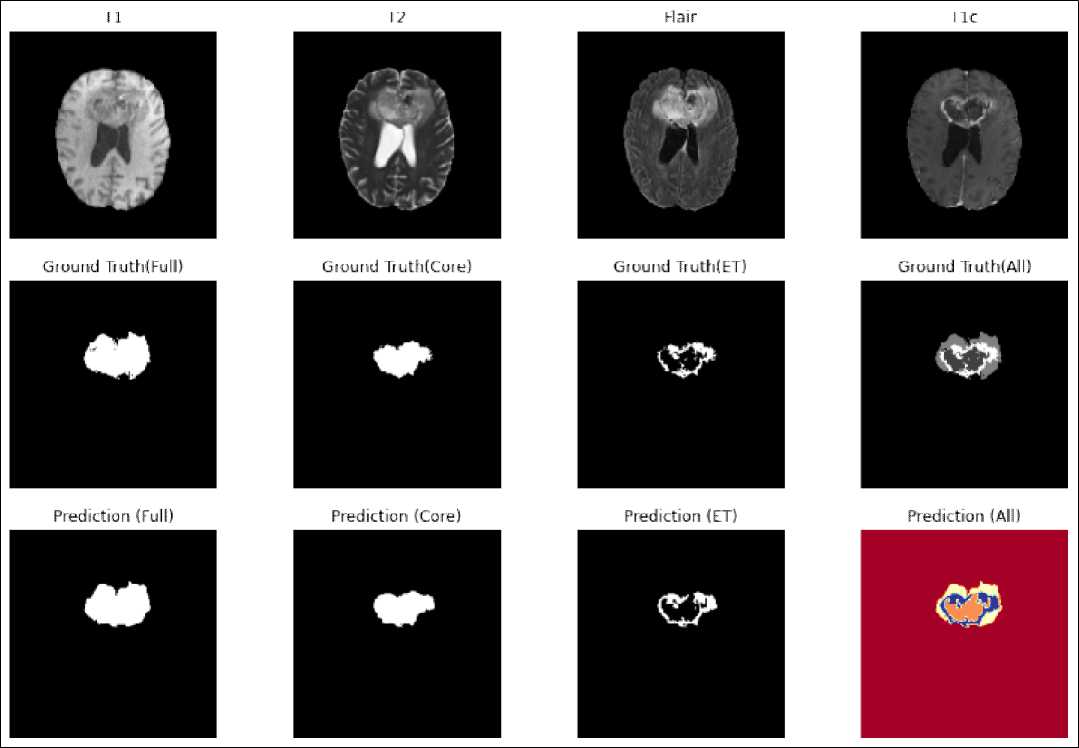
Gambar 11 Hasil Prediksi Dari Model U-NET Dengan Fungsi Aktivasi ReLU
-
V. KESIMPULAN [1]
Berdasarkan pengujian yang dilakukan dan hasil yang ditunjukkan pada Tabel I, nilai akurasi pada saat validasi [2] untuk full tumor, core tumor dan enhancing tumor berturut-turut adalah sebagai berikut: 90.22%, 78.09%, 80.20%. [3]
Untuk penelitian selanjutnya, disarankan agar merubah fungsi optimasi seperti LReLU, PReLU atau ELU serta menambah variasi data augmentation (rotasi, shift, blur dan sebagainya) untuk memperkaya dataset dengan harapan dapat [4] memperbaiki hasil akurasi untuk permasalahan segmentasi citra tumor otak.
[5]
Referensi
Ida Bagus Leo Mahadya Suta: Segmentasi Tumor Otak Berdasarkan…
D. Ph et al., “Brain Tumor Epidemiology: Consensus from the Brain Tumor Epidemiology Consortium (BTEC),” NIH Public Access, vol. 113, no. 1, pp. 1953–1968, 2010.
R. Riley, J. Murphy, and T. Higgins, “MRI imaging in pediatric appendicitis,” J. Pediatr. Surg. Case Reports, vol. 31, no. January, pp. 88–89, 2018.
I. Bagus, L. Mahadya, R. S. Hartati, and Y. Divayana, “Diagnosa Tumor Otak Berdasarkan Citra MRI ( Magnetic Resonance Imaging ),” Maj. Ilm. Teknol. Elektro, vol. 18, no. 2, pp. 149–154, 2019.
Tech Wire Asia, “Medical diagnosis AI in Beijing beats real doctors,” Tech Wire Asia, 2018. [Online]. Available: https://techwireasia.com/2018/07/medical-diagnosis-ai-in-beijing-beats-real-doctors/.
P. P. Winangun, I. M. O. Widyantara, and R. S. Hartati, “Learning Machine dengan Kernel Linear untuk Mengklasifikasi Kelainan Paru-Paru,” Maj. Ilm. Teknol. Elektro, vol. 19, no. 1, pp. 83–88,
p-ISSN:1693 – 2951; e-ISSN: 2503-2372

2020.
-
[6] A. Pinto, H. Correia, J. Oliveira, D. M. L. D. Rasteiro, and C. A. Silva, “Brain Tumour Segmentation based on Extremely Randomized Forest with High-Level Features,” pp. 3037–3040, 2015.
-
[7] N. Subbanna, D. Precup, and T. Arbel, “Iterative multilevel MRF leveraging context and voxel information for brain tumour segmentation in MRI,” Proc. IEEE Comput. Soc. Conf. Comput. Vis. Pattern Recognit., pp. 400–405, 2014.
-
[8] M. Soltaninejad et al., “Automated brain tumour detection and segmentation using superpixel-based extremely randomized trees in FLAIR MRI,” Int. J. Comput. Assist. Radiol. Surg., vol. 12, no. 2, pp. 183–203, 2017.
-
[9] W. Wu, A. Y. C. Chen, L. Zhao, and J. J. Corso, “Brain tumor detection and segmentation in a CRF (conditional random fields) framework with pixel-pairwise affinity and superpixel-level features,” Int. J. Comput. Assist. Radiol. Surg., vol. 9, no. 2, pp. 241–253, 2014.
-
[10] S. Shen, W. A. Sandham, M. H. Granat, M. F. Dempsey, and J. Patterson, “A New Approach to Brain Tumour Diagnosis Using Fuzzy Logic Based Genetic Programming,” in Proceedings of the 25th Annual International Conference of the IEEE EMBS, 2004, vol. 1, pp. 870–873.
-
[11] A. Das and M. Bhattacharya, “A study on prognosis of brain tumors using fuzzy logic and genetic algorithm based techniques,” in Proceedings - 2009 International Joint Conference on Bioinformatics, Systems Biology and Intelligent Computing, IJCBS 2009, 2009, no. 1, pp. 348–351.
-
[12] L. Szilagyi, L. Lefkovits, and B. Benyo, “Automatic Brain Tumor Segmentation in multispectral MRI volumes using a fuzzy c-means cascade algorithm,” 2015 12th Int. Conf. Fuzzy Syst. Knowl. Discov. FSKD 2015, pp. 285–291, 2016.
-
[13] J. Belghese and S. Agustin, “Brain Tumor Segmentation using Pattern Neural Networks with MRI Images,” IJSTE -International J. Sci. Technol. Eng., vol. 3, no. 09, pp. 641–644, 2017.
-
[14] S. Singh, “Classification of Human Brain Tumors from MRI Using K-NN Algorithm,” Int. J. Adv. Sci. Eng. Technol., vol. 3, no. 1, pp. 34–37, 2015.
-
[15] R. M. Azawi and I. T. Ibrahim, “A Hybrid Approach for Classification of MRI Brain Tumors Using Genetic Algorithm , K-
Nearest Neighbor and Probabilistic Neural Network,” Int. J. Comput. Sci. Inf. Secur., vol. 16, no. 5, pp. 74–85, 2018.
-
[16] A. Arora, P. Roy, S. Venktesan, and R. Babu, “k-NN Based Classification of Brain MRI Images using DWT and PCA to Detect Different Types of Brain Tumour,” Int. J. Med. Res. Heal. Sci., vol.
-
6, no. 9, pp. 15–20, 2017.
-
[17] W. Mengqiao, Y. Jie, C. Yilei, and W. Hao, “The multimodal brain tumor image segmentation based on convolutional neural networks, 2017 2nd IEEE Int. Conf. Comput. Intell. Appl., pp. 336–339, 2017.
-
[18] S. Hussain, S. M. Anwar, and M. Majid, “Brain Tumor Segmentation using Cascaded Deep Convolutional Neural Network, 39th Annu. Int. Conf. IEEE Eng. Med. Biol. Soc., pp. 1998–2001, 2017.
-
[19] A. Pinto, V. Alves, and C. A. Silva, “Brain Tumor Segmentation using Convolutional Neural Networks in MRI Images,” IEEE Trans. Med. Imaging, vol. 35, no. 5, pp. 1240–1251, 2016.
-
[20] J. W. Ha et al., “Predicting high-risk prognosis from diagnostic histories of adult disease patients via deep recurrent neural networks,” 2017 IEEE Int. Conf. Big Data Smart Comput. BigComp 2017, pp. 394–399, 2017.
-
[21] O. Ronneberger, P. Fischer, and T. Brox, “U-net: Convolutional networks for biomedical image segmentation,” Lect. Notes Comput. Sci. (including Subser. Lect. Notes Artif. Intell. Lect. Notes Bioinformatics), vol. 9351, pp. 234–241, 2015.
-
[22] L. Dirven, N. K. Aaronson, J. J. Heimans, and M. J. B. Taphoorn, “Health-related quality of life in high-grade glioma patients,” Chin. J. Cancer, vol. 33, no. 1, pp. 40–45, 2014.
-
[23] W. Rawat, “Deep Convolutional Neural Networks for Image Classification : A Comprehensive Review,” vol. 2449, pp. 23522449, 2017.
-
[24] G. Ö. Yiğit and B. M. Özyildirim, “Comparison of convolutional neural network models for food image classification,” vol. 1839, 2018.
-
[25] N. Sharma, V. Jain, and A. Mishra, “An Analysis of Convolutional Neural Networks for Image Classification,” Procedia Comput. Sci., vol. 132, no. Iccids, pp. 377–384, 2018.
-
[26] N. M. Balasooriya and R. D. Nawarathna, “A sophisticated convolutional neural network model for brain tumor classification,” 2017 IEEE Int. Conf. Ind. Inf. Syst., pp. 1–5, 2017.
ISSN 1693 – 2951
Ida Bagus Leo Mahadya Suta: Segmentasi Tumor Otak Berdasarkan...
Discussion and feedback