Keragaman Genetik Ikan Tongkol Abu-Abu (Thunnus tonggol) yang Didaratkan di Pasar Ikan Sagulung, Batam, Kepulauan Riau Berdasarkan DNA Mitokondria
on
Journal of Marine and Aquatic Sciences 7(2), 176-183 (2021)
Keragaman Genetik Ikan Tongkol Abu-Abu (Thunnus tonggol) yang Didaratkan di Pasar Ikan Sagulung, Batam, Kepulauan Riau Berdasarkan DNA Mitokondria
Suryany Devy a*, Ida Ayu Astarini b, I Nyoman Giri Putra a, Andrianus Sembiring c, Ni Luh Astria Yusmalinda c, M. Danie Al Malik c, Ni Putu Dian Pertiwi c
a Program Studi Ilmu Kelautan, Fakultas Kelautan dan Perikanan, Universitas Udayana, Badung, Bali – Indonesia b Program Studi Ilmu Biologi, Universitas Udayana, Badung, Bali - Indonesia
c Biodiversitas Indonesia (Bionesia), Denpasar, Bali – Indonesia
* Penulis koresponden. Tel.: +62-895-6173-22575
Alamat e-mail: suryanydevy@gmail.com
Diterima (received) 18 Januari 2021; disetujui (accepted) 23 Agustus 2021; tersedia secara online (available online) 1 Desember 2021
Abstract
Longtail Tuna (Thunnus tonggol) is one of the neritic species of tuna from the Scrombridae family. T. tonggol is oceanicromus and ussually found in tropical and subtropical waters in the Indo-Pacific region. Although known as one of the tuna species, the information of this species is very lacking, especially in Indonesia. Therefore, this study aims to determine the genetic diversity and kinship relationships of longtail tuna (T. tonggol) collected at Sagulung fish market, Batam, Riau Islands. Molecular analysis were carried out using genetic markers namely mitochondrial D-loop (control regian) with the stages of DNA extraction, PCR (Polymerase Chain Reaction), electrophoresis, sequencing, and data analysis. A total of 21 longtail tuna samples were identified using molecularly and confirmed as T. tonggol species, which have a base length ranging from 482 - 523 bp (base pairs). The value of haplotype diversity (Hd) of longtail tuna was 1,00000 and the value of nucleotide diversity (π) was 0,01654. The results of phylogenetic tree reconstruction showed that all samples collected were in the same group (clade) with the average genetic distance in one T. tonggol clade obtained a value of 0.010 which has a close relationship between samples. This result can be use as additional information for longtail tuna species in Indonesia.
Keywords: control region; polymerase chain reaction; mitochondrial DNA; genetic conservation; phylogenetic
Abstrak
Ikan tongkol abu-abu (Thunnus tonggol) merupakan salah satu spesies neritik tuna dari famili Scrombridae. T. tonggol adalah ikan oseanodromus dan biasanya ditemukan di perairan tropis dan subtropis di wilayah Indo-Pasifik. Meskipun dikenal sebagai salah satu spesies tuna, informasi spesies ini sangat kurang, terutama di Indonesia. Oleh karena itu, penelitian ini bertujuan untuk mengetahui keragaman genetik dan hubungan kekerabatan ikan tongkol abu-abu (T. tonggol) yang didaratkan di pasar ikan Sagulung, Batam, Kepulauan Riau. Analisis molekuler dilakukan menggunakan penanda genetik yaitu D-loop (control region) mitokondria dengan tahapan ekstraksi DNA, PCR (Polymerase Chain Reaction), elektroforesis, sekuensing, dan analisis data. Sebanyak 21 sampel ikan tongkol abu-abu berhasil di identifikasi secara molekuler dan dikonfirmasi sebagai spesies T. tonggol, yang memiliki panjang basa berkisar antara 482 – 523 bp (base pairs). Nilai keragaman haplotipe (Hd) ikan tongkol abu-abu (T. tonggol) sebesar 1,00000 dan nilai keragaman nukleotida (π) sebesar 0,01654. Hasil rekontruksi pohon filogenetik menunjukkan bahwa keseluruhan sampel yang dikoleksi berada dalam 1 kelompok yang sama (clade) dengan rata-rata jarak genetik dalam satu clade T. tonggol diperoleh nilai 0.010 yang memiliki hubungan kekerabatan yang dekat anatar sampel. Hasil ini dapat digunakan sebagai informasi tambahan untuk spesies Ikan tongkol abu-abu di Indonesia.
Kata Kunci: control region; polymerase chain reaction; DNA mitokondria; konservasi genetic; filogenetik
Negara Indonesia terdiri atas 17.502 buah pulau, dan memiliki garis pantai sepanjang 81.000 km2 dengan luas wilayah perikanan di laut sekitar 5,8 juta km2, yang terdiri dari perairan kepulauan dan teritorial seluas 3,1 juta km2 serta perairan Zona Ekonomi Eksklusif Indonesia (ZEEI) seluas 2,7 juta km2 (Yahya dkk., 2013). Hal ini menunjukkan bahwa prospek pembangunan perikanan dan kelautan Indonesia dinilai sangat tinggi dan menjadi salah satu kegiatan ekonomi yang strategis. Di perairan laut Indonesia terdapat beberapa jenis ikan bernilai ekonomis tinggi antara lain cakalang, tongkol, tenggiri, kakap, cumi-cumi, ikan-ikan karang, ikan hias, dan (Yaskun dan Sugiarto, 2017).
Tuna merupakan salah satu jenis ikan yang memiliki tingkat ekspor yang tinggi dan menjadi komoditi utama kegiatan ekspor perikanan Indonesia. Pada tahun 2013 Indonesia telah mengekspor ikan tuna sebesar 209,410 ton dengan nilai produksi 768,4 juta USD (Alfajri dan Pahlawan, 2017). Ikan tuna termasuk kelompok ikan pelagis yang aktif dan memiliki pergerakan yang luas. Tuna adalah ikan laut yang terdiri atas beberapa spesies dari famili Scombridae, terutama genus Thunnus. Tuna dari genus Thunnus terdiri dari 7 spesies yaitu tuna albakora (T. alalunga), tuna sirip kuning (T. albacares), tuna sirip biru selatan (T. macoyii), tuna sirip biru utara (T. thynnus), tuna sirip hitam (T. atlanticus), tuna mata besar (T. obesus) dan tongkol abu-abu (T. tonggol) (Sulistyaningsih dkk., 2016).
Menurut Hernawan (2017), sepanjang wilayah Samudera Hindia produksi tangkapan ikan tongkol abu-abu di Indonesia pada tahun 2013 produksi mencapai sebesar 28.384 ton/tahun dan mengalami penurun pada tahun 2015 menjadi sebesar 22.691 ton/tahun. Penurunan populasi tongkol abu-abu ini, dikhawatirkan akan menyebabkan penurunan variasi genetik tongkol abu-abu. Untuk mengantisipasi hal tersebut diperlukan suatu program konservasi, untuk melindungi ikan tongkol abu-abu dari kelangkaan. Salah satu upaya yang dapat dilakukan melalui kajian tentang keragaman genetik populasi ikan tongkol abu-abu. Kajian keragaman genetik bertujuan untuk mengetahui gambaran dalam menetukan spesies dan status hidup populasi. Selain itu Informasi genetik pada ikan dengan migrasi yang tinggi seperti tuna sangat penting diketahui untuk pemanfaatan yang bersifat lestari (Santos dkk., 2010).
Identifikasi molekuler dapat digunakan untuk mengetahui hubungan kekerabatan dalam suatu populasi ikan di suatu daerah, yaitu dengan menggunakan metode filogenetik. Filogenetik merupakan suatu metode yang digunakan untuk melihat dan memodelkan kedekatan suatu spesies dengan spesies lainnya. Filogenetik adalah studi mengenai hubungan antara organisme dan memperlihatkan hubungan kekerabatan (phylogenetic relationship) satu sama lain secara genetik (Saleky et al., 2016). Penggunaan sekuen DNA dalam penelitian filogenetik telah meningkat pesat dan telah dilakukan pada semua tingkatan taksonomi, misalnya famili, genus, dan spesies.
Penggunaan DNA mitokondria (mtDNA) banyak dilakukan untuk identifikasi suatu spesies karena memiliki beberapa kelebihan yaitu berevolusi lebih cepat dan terdapat dalam jumlah yang lebih banyak dibandingkan dengan DNA inti (Satiyarti dkk., 2017). Pada DNA mitokondria (mtDNA) lokus control region memiliki tingkat polimorfisme tinggi dan mempunyai laju mutasi yang lebih cepat dibandingkan dengan ruas lainnya, sehingga menyebabkan urutan nukleotida control region sangat bervariasi antar individu (Satiyarti dan Anggita, 2018). Analisis pada daerah control region dapat digunakan untuk melihat keragaman genetik antar subspesies ataupun antar populasi.
Penelitian ini penting dilakukan karena seiring dengan meningkatnya permintaan pasar dari tahun ke tahun, maka semakin tinggi pula eksploitasi terhadap ikan tuna khususnya jenis ikan tongkol abu-abu. Oleh karena itu, di Samudra Hindia bagian Barat Sumatera, khususnya di Kepulauan Riau, penelitian mengenai keragaman genetik dan filogenetik ikan tongkol abu-abu (Thunnus tonggol) yang didaratkan di Pasar ikan Sagulung, Batam Kepulauan Riau belum pernah dilakukan. Hal ini akan berguna sebagai tambahan informasi dari unit biologi yang sangat penting untuk menentukan keputusan manajemen perikanan yang tepat. Oleh karena itu, informasi mengenai keragaman genetik akan dapat membantu dalam proses pengelolaan kawasan perlindungan laut secara berkelanjutan.
Pengambilan data dilaksanakan di Pasar Ikan Sagulung, Batam Kepulauan Riau (Gambar 1). Pengambilan sampel ikan tongkol abu-abu (T.
tonggol) dilakukan pada bulan Maret sampai April
2019. Analisis molekuler dilakukan di
Laboratorium Yayasan Biodiversitas Indonesia (BIONESIA), Bali.
Gambar 1. Peta Lokasi Pengambilan Sampel
-
2.2 Alat dan Bahan
Alat yang digunakan pada penelitian ini, antara lain glove, tary, plate numeric, tube, microtubbe, strips tube PCR, cetakan, sisir (comb), api bunsen, pinset, mikro pipet, tips, gelas beker, vortex, centrifuge, heating block, UV transiluminator, thermal cycler, neraca analitik, mesin PCR, mesin elektroforesis, microwave dan kamera. Bahan yang digunakan Etanol 96%, larutan chelex 10%, ddH2O, 10X PCR buffer, DNTPs, MgCL2, primer CRK, primer CRE, PE Amplitaq, bubuk agarose, loading dye, biotium dan low mass ladder.
-
2.3 Pengambilan Sampel Jaringan
Sampel jaringan diambil pada bagian sirip pektoral (sirip dada) yang berukuran ± 5 cm.
Sampel disimpan dalam tube yang beriisi larutan etanol 96% untuk pengawetan. Kemudian sampel diberi label pada masing-masing tube. Jumlah total sampel jaringan yang berhasil dikoleksi sebanyak 21 sampel.
-
2.4 Analisis Molekuler
-
2.4.1. Ekstraksi
-
Ekstraksi dilakukan dengan menggunakan larutan Chelex 10% (Akbar dkk., 2018). Jaringan sampel diambil ± 2 mm dan dimasukkan kedalam tube berisi larutan Chelex 10%, kemudian divortex selama ± 15 detik dan centrifuge selama ± 1 menit.
Setelah itu, sampel dipanaskan didalam heating block dengan suhu 95°C selama 60 menit dan divortex dan centrifuge selama selama ± 15 detik
-
2.4.2. Polymerase Chain Reaction (PCR)
Proses amplifikasi DNA dilakukan pada mtDNA control region menggunakan primer forward CRK 5’-AGC TCA GCG CCA GAG CGC CGG TCT TGT AAA-3’ dan primer reverse CRE 5’-CCT GAA GTA GGA ACC AGA TG-3’ (Lee et al., 1995). Pengaturan tahapan PCR meliputi pradenaturasi pada suhu 80°C selama 10 detik, denaturasi pada suhu 94°C selama 30 detik, annealing pada suhu 50°C selama 30 detik, extension pada suhu 72°C selama 45 detik. Tahapan PCR ini diulang sebanyak 38 siklus.
-
2.4.3. Elektroforesis
Elektroforesis bertujuan untuk mengetahui ada tidaknya hasil amplifikasi DNA dalam produk PCR. Sebelum melakukan elektroforesis, terlebih dahulu dibuat media elektroforesis yaitu gel agarosa 1%. Pembuatan gel agarose yang mencampurkan 0.75 gram bubuk agarose dan 75 ml larutan buffer dimasukan kedalam gelas beker lalu dipanaskan didalam microwave selama 4 menit. Gel agarose dituangkan ke cetakan yang telah dipasang sisir pembuat sumur (well) dan diamkan selama 60 menit. Tambahkan ke masing – masing sampel 2μl loading dye, biotium dan ladder. Mesin elektroforesis dinyalakan selama 45 menit dengan kecepatan arus listrik 100V dan 80 watt. Selanjutnya dilakukan visualisasi fragmen DNA dengan LED Blue Light Transilluminator.
-
2.4.4. DNA Sekuensing
Produk PCR yang positif mengandung DNA kemudian dikirim ke DNA Sequencing Facility. Sekuensing DNA dilakukan untuk menentukan urutan nukleotida yang terdapat dalam DNA. Sekuensing DNA dilakukan dengan metode Sanger Sequencing.
-
2.5 Analisis Data
Analisis molekuler sekuen mtDNA control region menggunakan program MEGA5. Sekuen hasil penjajaran (alignment) dianalis dengan menggunakan Clustal W kemudian dibandingkan dengan sekuen yang ada di GenBank melalui
program BLAST (Basic Local Alignment Search Tool) dengan situs web (http://www.ncbi.nih.gov). Analisis Tingkat keragaman genetik pada data sekuen mtDNA control region menggunakan program DnaSP 5.10 (Fakhri dkk., 2015). Deskripsi hasil analisis keragaman genetik ditampilkan dengan nilai keragaman haplotipe (Hd) dan keragaman nuleotida (π); sedangkan analisis filogenetik dilakukan mengunakan program MEGA5, dengan metode Neighbor Joining (kimura 2 – parameter) dengan replikasi bootstraps 1000 (Tamura et al., 2011).
Hasil identifikasi molekuler menunjukkan bahwa 21 sampel ikan tongkol abu-abu berhasil diidentifikasi secara molekuler. Hal ini menunjukkan pada proses amplifikasi, primer yang digunakan menempel pada DNA target secara spesifik. Menurut Wardani dkk. (2017), keberhasilan amplifikasi DNA lebih didasarkan pada kesesuaian primer serta efisiensi dan
optimasi proses PCR. Primer merupakan bagian penting dalam reaksi PCR karena primer merupakan inisiator pada sintesis DNA target.
Pencocokan data sekuen dilakukan menggunakan Basic Local Alignment Search Tool (BLAST) pada situs GenBank (http://www.ncbi.nih.gov). BLAST merupakan program untuk mencari dan menganalisis kemiripan (homologi) sekuen suatu organisme. Hasil analisis BLAST (Tabel 1) menunjukkan bahwa ikan tongkol abu-abu yang diperoleh dari Pasar Ikan Sagulung Batam dikonfirmasi sebagai spesies T. tonggol yang mempunyai nama nasional tongkol abu-abu dan memiliki panjang basa berkisar antara 482 hingga 523 bp (base pairs), dengan nilai identity yang menunjukkan besarnya persentase kesamaan basa nukleotida sampel dengan data GenBank berkisar antara 97%-99%. Nilai persentase identity adalah perbandingan kemiripan sekuen antara spesies yang diujikan dengan data spesies yang ada di genbank (Sahari dkk., 2017). Seluruh sampel memiliki Query cover yang menunjukkan nilai sebesar 100%. Menurut Wardani dkk. (2017), query cover adalah persentase
Tabel 1
Hasil BLAST Sampel Ikan tongkol abu-abu (Thunnus tonggol)
|
No. |
Kode Sampel |
Hasil BLAST |
Panjang Sekuen |
Ident |
Query cover |
Accession |
|
1 |
BIO06_050_001_SAGULUNG |
Thunnus tonggol |
523 bp |
98.28% |
100% | |
|
2 |
BIO06_050_002_SAGULUNG |
Thunnus tonggol |
523 bp |
97.71% |
100% | |
|
3 |
BIO06_050_003_SAGULUNG |
Thunnus tonggol |
523 bp |
97.71% |
100% | |
|
4 |
BIO06_050_004_SAGULUNG |
Thunnus tonggol |
523 bp |
98.09% |
100% | |
|
5 |
BIO06_050_005_SAGULUNG |
Thunnus tonggol |
523 bp |
97.90% |
100% | |
|
6 |
BIO06_050_006_SAGULUNG |
Thunnus tonggol |
523 bp |
97.71% |
100% | |
|
7 |
BIO06_050_007_SAGULUNG |
Thunnus tonggol |
523 bp |
97.90% |
100% | |
|
8 |
BIO06_050_008_SAGULUNG |
Thunnus tonggol |
523 bp |
97.71% |
100% | |
|
9 |
BIO06_050_009_SAGULUNG |
Thunnus tonggol |
523 bp |
97.71% |
100% | |
|
10 |
BIO06_050_010_SAGULUNG |
Thunnus tonggol |
523 bp |
98.09% |
100% | |
|
11 |
BIO06_050_012_SAGULUNG |
Thunnus tonggol |
523 bp |
98.09% |
100% | |
|
12 |
BIO06_050_014_SAGULUNG |
Thunnus tonggol |
523 bp |
97.90% |
100% | |
|
13 |
BIO06_050_022_SAGULUNG |
Thunnus tonggol |
523 bp |
98.09% |
100% | |
|
14 |
BIO06_050_023_SAGULUNG |
Thunnus tonggol |
482 bp |
98.13% |
100% | |
|
15 |
BIO06_050_025_SAGULUNG |
Thunnus tonggol |
523 bp |
99.24% |
100% | |
|
16 |
BIO06_050_027_SAGULUNG |
Thunnus tonggol |
523 bp |
98.28% |
100% | |
|
17 |
BIO06_050_029_SAGULUNG |
Thunnus tonggol |
523 bp |
98.10% |
100% | |
|
18 |
BIO06_050_031_SAGULUNG |
Thunnus tonggol |
523 bp |
98.66% |
100% | |
|
19 |
BIO06_050_032_SAGULUNG |
Thunnus tonggol |
482 bp |
97.93% |
100% | |
|
20 |
BIO06_050_034_SAGULUNG |
Thunnus tonggol |
523 bp |
97.32% |
100% | |
|
21 |
BIO06_050_035_SAGULUNG |
Thunnus tonggol |
523 bp |
99.04% |
100% |
panjang nukleotida sampel yang sesuai dengan urutan nukleotida yang ada di Genbank. Semua data dibandingkan dengan sekuen T. tonggol yang teregistrasi di genbank dengan Accession Number HQ425780.1. Accession Number adalah pengenal rekaman urutan DNA atau protein untuk memungkinkan pelacakan berbagai versi urutan dan urutan yang terkait dengan urutan tersebut (NCBI, 2013).
-
3.2 Keragaman Genetik
Hasil analisis keragaman genetik yang didapatkan dari 21 sampel menghasilkan nilai keragaman haplotipe (Hd) sebesar 1,00000 dan nilai keragaman nukleotida (π) sebesar 0,01654. Nilai ini menunjukkan bahwa spesies tuna ini memiliki keragaman haplotipe yang tinggi dengan jumlah 21 haplotipe, sementara mempunyai keragaman nukleotida yang rendah. Keragaman nukleotida yang rendah menunjukkan hanya terdapat 36 situs nukleotida yang bersifat polimorfik (variable site) dari 523 total situs nukleotida. Tingginya keragaman haplotipe dapat diasumsikan bahwa populasi ikan tongkol abu-abu di pasar Ikan Sagulung Batam memiliki peluang hidup yang lebih baik dalam merespon perubahan lingkungan. Jatmiko dkk. (2018), mengatakan bahwa keragaman haplotipe yang tinggi pada sebagian besar spesies tuna merupakan tipe pola genetik ikan famili Scombridae. Keragaman genetik ikan tuna yang tinggi juga telah dilaporkan oleh beberapa penelitian lainnya yang disajikan pada (Tabel 2). Keragaman haplotipe ikan tongkol abu-
abu (T. tonggol) yang tinggi ini diduga disebabkan oleh dua faktor, pertama yaitu populasi ikan tongkol abu-abu ini berukuran besar sehingga sangat memungkinkan terjadinya perkawinan acak antar individu untuk bertemu dengan individu lain, baik dengan genotipe yang sama maupun berbeda dengannya. Dengan adanya sistem perkawinan acak ini, dapat membantu dalam meningkatkan frekuensi alel pada generasi keturunan berikutnya.
Faktor kedua yaitu kemampuan migrasi genetik, dimana keragaman genetik suatu populasi akan meningkat jika terdapat suatu masukan genetik dari populasi lain. Selain itu akan menyebabkan terjadinya pertemuan antar populasi yang besar sehingga terjadinya perkawinan silang dan memungkinkan adanya aliran gen (gen flow) antar populasi yang berbeda, yang akan diperoleh variasi gen yang berbeda-beda. Hal ini disebabkan karena tongkol abu-abu termasuk salah satu spesies tuna neritik yang bersifat oseanodromus dan pola migrasi mengikuti arus perairan. Oleh karena itu, sirkulasi arus yang terdapat di Indonesia merupakan salah satu faktor yang berpengaruh terhadap keragaman genetik ikan tongkol abu-abu. Selain itu arus laut juga berpengaruh terhadap persebaran larva dan memberikan peranan yang sangat penting dalam proses pertukaran genetik antar populasi. Tangke dkk. (2017), mengatakan arus dapat memberikan pengaruh yang besar pada keberadaan ikan karena dapat mempengaruhi rute migrasi ikan, tingkah laku ikan, distribusi makan, penyebaran dan kelimpahan ikan serta dapat membawa telur dan
Tabel 2
Perbandingan keragaman genetik antar spesies tuna
|
No. |
Spesies |
Marka |
Keragaman Nukleotida (π) |
Keragaman Haplotipe (Hd) |
Sumber |
|
1. |
Longtail Tuna |
Control |
0,0187 |
0,998 |
Kunal et al., 2014 |
|
(Thunnus tunggol) |
region | ||||
|
2 |
Bluefin Tuna |
Control |
0,0179 |
0,987 |
Boustany et al., |
|
(Thunnus thynnus) |
region |
2008 | |||
|
3. |
Bluefin Tuna |
Control |
0,015 |
0,991 |
Carlsson et al., |
|
(Thunnus thynnus) |
region |
2004 | |||
|
4. |
Tuna Mata Besar |
Control |
0,043 |
0,999 |
Chiang et al., |
|
(Thunnus obesus) |
region |
2008 | |||
|
5. |
Tuna Mata Besar |
Control |
0,026 |
0.997 |
Akbar et al., |
|
(Thunnus obesus) |
region |
2018 | |||
|
6. |
Tuna Sirip Kuning (Thunnus |
Control |
0,069 |
0,998 |
Kunal et al., 2013 |
|
albacares) |
region |
larva ikan dari tempat pemijahan ke tempat asuhan.
Menurut Moria dkk. (2009), bahwa sifat migrasi ikan tuna yang bergerombol (schooling) cenderung menghasilkan nilai keragaman genetik yang tinggi. Hasil keragaman genetik yang didapatkan pada ikan tongkol abu-abu yang didaratkan di pasar ikan Sagulung, Batam memberikan gambaran bahwa populasi ikan ini masih dalam kondisi baik, tingginya peluang bertahan hidup dan mampu beradaptasi terhadap gangguan kualitas lingkungan. Walaupun diketahui bahwa sumberdaya ikan tuna adalah spesies yang dijadikan ikan target dalam operasi penangkapan dan telah lama dieksploitasi karena memiliki nilai komersil yang tinggi. Meskipun demikian kegiatan penangkapan tidak bisa dibiarkan secara terus menerus karena dapat mempengaruhi struktur populasi yang berakibat turunnya keragaman genetik suatu spesies. Hal ini memberikan
padangan bahwa perlu adanya strategi untuk melindungi keanekaragaman hayati yang diperlukan melalui konservasi genetik.
Kunci penting untuk melestarikan keragaman genetik dapat dilakukan dengan cara preventif diantaranya adalah, pertama memberikan regulasi tentang pelaksanaan batas ukuran minimum tangkapan pada setiap operasi penangkapan, dimana ikan yang ditangkap adalah ikan dewasa dengan proporsi ukuran yang telah ditentukan. Hal ini guna memberikan peluang ikan kecil untuk hidup dan berkembang menjadi dewasa, dengan demikian proses regenerasi tetap berjalan. Kedua adalah pembuatan suatu kawasan reservasi yang dilindungi otoritas terkait dan melibatkan masyarakat sekitarnya. Kawasan ini penting didalam kegiatan konservasi genetik karena berperan untuk menjaga kelangsungan hidup populasi ikan tuna di suatu perairan. Kehadiran kawasan ini juga dapat meningkatkan reproduksi dan menjaga keseimbangan genetik ikan agar tetap tumbuh dan berkembang di habitatnya.
-
3.3 Analisis Filogenetik dan Jarak Genetik
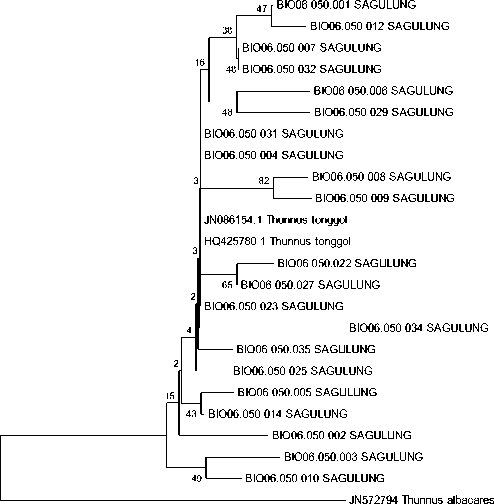
Hasil rekonstruksi pohon filogenetik menunjukkan bahwa semua sampel ikan tongkol abu-abu dikelompokkan menjadi 1 clade yang sama (Gambar 2). Pada clade T. tonggol, terbentuk cabang kecil yang membuktikan bahwa adanya perbedaan interspesies. Perbedaan interspesies dipengaruhi oleh perbedaan komposisi basa pada
urutan nukleotida antar individu namun tidak mempengaruhi kesamaan spesies antar populasi.
Hasil analisis pohon filogenetik menggunakan metode neighbor joining menunjukkan sampel BIO06.050.034_SAGULUNG memiliki garis panjang cabang yang lebih panjang dibandingkan antara sampel yang lain. Panjang suatu cabang yang berbeda-beda antar spesies menunjukkan perbedaan basa nukleotida. Semakin panjang cabang yang terbentuk menunjukkan spesies tersebut memiliki perbedaan basa DNA yang lebih besar dari pada spesies lainnya. Sebaliknya, semakin pendek suatu cabang menunjukkan perbedaan basa nukleotida yang lebih sedikit dibuktikan oleh bentuk pohon filogenetik (Fahmi dan Budiharjo, 2014).
Pada setiap percabangan yang membentuk clade atau kelompok yang didukung oleh nilai boostrap juga didukung oleh nilai jarak genetik. Terlihat pada hasil yang didapat, nilai bootstrap yang terdapat hasil percabangan T. tonggol menunjukkan nilai bootstrap kurang dari 70%. Menurut Wirdateti dan Semiadi (2017), Jika nilai boostrap kurang dari 70% maka peluang terjadinya perubahaan susunan clade adalah tinggi, sehingga ketika dilakukan analisis, percabangan dan pohon yang dibentuk masih dapat berubah-ubah dan sebaliknya. Meskipun pohon filogenetik yang terbentuk memiliki nilai boostrap kurang dari 70%, tetapi topologi pohon filogentik yang terbentuk sudah menunjukkan bahwa keseluruhan sampel yang dikoleksi berada dalam 1 kelompok yang sama (clade) dengan T. tonggol yang diperoleh dari database genbank, sehingga dapat dinyatakan sebagai spesies T. tonggol. Selain itu, di dalam pembentukan pohon filogenetik, tuna sirip kuning juga digunakan sebagai outgroup, dan keseluruhan sampel berada dalam clade yang berbeda dengan outgrup.
Hasil analisis rata-rata jarak genetik dalam satu clade T. tonggol diperoleh nilai 0.010 yang artinya semua sampel memiliki nilai jarak genetik yang kecil. Semakin kecil rentang nilai yang didapatkan maka semakin dekat pula tingkat kekerabatannya antar spesies. Sebaliknya, semakin besar rentang nilai yang didapatkan maka semakin jauh pula tingkat kekerabatan antar spesies tersebut (Jefri dkk., 2015). Sedangkan untuk perbedaan rata-rata jarak genetik antar clade T. tonggol dengan outgrup T. albacares diperoleh nilai 0.045.
0.005
Clade 1
Gambar 2. Hasil pohon filogenetik ikan tuna menggunakan metode Neighbor Joining dengan Kimura 2 – parameter .
Hasil analisis keragaman genetik menunjukan bahwa sampel ikan tongkol abu-abu (Thunnus tonggol) memiliki nilai keragaman haplotipe (Hd) sebesar 1,00000 dan keragaman nukleotida (π) 0,01654. Hubungan kekerabatan filogenetik ikan tongkol abu-abu dikelompokkan menjadi 1 clade yang sama dengan rata-rata jarak genetik dalam satu clade T. tonggol diperoleh nilai 0.010, nilai ini menunjukkan bahwa keseluruhan sampel yang dikoleksi merupakan spesies T. tonggol.
Ucapan terimakasih
Penulis mengucapkan terimakasih kepada United States Agency for International Development (USAID) melalui Enhanced Engagement in Research (PEER) Science Program (AID-OAA-A-11-00012) yang telah mendanai penelitian ini, serta Biodiversitas Indonesia (BIONESIA) atas segala fasilitas dan laboratorium dalam penelitian ini. Selain itu, terima kasih juga kepada serta semua pihak yang telah membantu dan membimbing dalam proses penyelesaian penelitian ini.
Daftar Pustaka
Akbar, N., Aris, M., Irfan, M., Tahir, I., Baksir, A., Surahman., Madduppa, H. H., & Kotta, R. (2018).
Filogenetik ikan tuna (Thunnus spp.) di perairan Maluku Utara, Indonesia. Jurnal Iktiologi Indonesia, 18(1), 1-11.
Akbar, N., Irfan, M., & Aris, M. (2018). Population
genetics and phylogeography of bigeye tuna in Moluccas Seas, Indonesia. Indonesian Journal of Marine Sciences/Ilmu Kelautan, 23(4), 145-155
Alfajri, S. I., & Pahlawan, I. (2017). Kepentingan
Indonesia mengekspor ikan tuna ke Amerika Serikat tahun 2012-2015. Jurnal Online Mahasiswa Fakultas Ilmu Sosial dan Ilmu Politik Universitas Riau, 4(2), 1-12.
Boustany, A. M., Reeb, C. A., Block, B. A. (2008). Mitochondrial DNA and electronic tracking reveal population structure of Atlantic bluefin tuna
(Thunnus thynnus). Marine Biology 156(1), 13-24.
Carlsson, J., Mcdowell, J. R., Díaz‐Jaimes, P., Carlsson, J. E. L., Boles, S. B., Gold, J. R., & Graves, J. E. (2004). Microsatellite and mitochondrial DNA analyses of Atlantic bluefin tuna (Thunnus thynnus thynnus)
population structure in the Mediterranean Sea. Molecular Ecology, 13(11), 3345-3356.
Chiang, H. C., Hsu, C. C., Wu, G. C. C., Chang, S. K., & Yang, H. Y. (2008). Population structure of bigeye tuna (Thunnus obesus) in the Indian Ocean inferred from mitochondrial DNA. Fisheries Research, 90(1-3), 305-312.
Fahmi, M. F. I., Budiharjo, A., & Suprihadi, A. (2014). Potensi rhizobakteri dari tanaman kubis (Brassica oleracea var. capitata L.) daerah Getasan Semarang sebagai agen biobakterisida terhadap patogen Xanthomonas campestris. Jurnal Biologi, 3(3), 53-64.
Fakhri, F., Narayani, I., & Mahardika, I. G. N. K. (2015). Keragaman genetik ikan cakalang (Katsuwonus pelamis) dari Kabupaten Jembrana dan Karangasem, Bali. Jurnal Biologi, 19(1), 11-14.
Hernawan, R. A. (2017). Analisis dayasaing dan faktor-faktor yang memengaruhi ekspor longtail tuna Indonesia di pasar Jepang. Skripsi. Bogor, Indonesia: Program Studi Agribisnis, Fakultas Ekonomi dan Manajemen Institut Pertanian Bogor.
Jatmiko, I., Rochman, F., & Agustina, M. (2018). Variasi genetik madidihang (Thunnus albacares; Bonnaterre, 1788) dengan analisis mikrosatelit di perairan Indonesia. Jurnal Penelitian Perikanan Indonesia, 24(3), 157-164.
Jefri, E., Zamani, N. P., Subhan, B., & Madduppa, H. H. (2015). Molecular phylogeny inferred from mitochondrial DNA of the grouper Epinephelus spp. in Indonesia collected from local fish market. Biodiversitas Journal of Biological Diversity, 16(2), 254263.
Kunal, S. P., Kumar, G., Menezes, M. R., & Meena, R. M. (2013). Mitochondrial DNA analysis reveals three stocks of yellowfin tuna Thunnus albacares (Bonnaterre, 1788) in Indian waters. Conservation genetics, 14(1), 205-213.
Kunal, S. P., Kumar, G., Menezes, M. R., & Meena, R. M. (2014). Genetic homogeneity in longtail tuna Thunnus tonggol (Bleeker, 1851) from the northwest coast of India inferred from direct sequencing analysis of the mitochondrial DNA D-loop region. Marine Biology Research, 10(7), 738-743.
Lee, W. J., Conroy, J., Howell, W. H., & Kocher, T. D. (1995). Structure and evolution of teleost mitochondrial control regions. Journal of Molecular Evolution, 41(1), 54-66.
Moria, S. B., Permana, G. N., & Hutapea, J. H. (2009). Karakterisasi tiga lokus mikrosatelit pada telur dan larva tuna sirip kuning, Thunnus albacares. Jurnal Perikanan Universitas Gadjah Mada, 11(2), 144-149.
NCBI. (2013). The NCBI Handbook. (2nd Edition). Bethesda, US: National Center for Biotechnology Information.
Sahari, J., Rimper, J., & Wullur, S. (2017). Identifikasi molekuler rotifer Brachionus sp. asal perairan
Tumpaan, Minahasa Selatan. Jurnal Pesisir dan Laut Tropis, 1(1), 56-61.
Saleky, D., Setyobudiandi, I., Toha, H. A., Takdir, M., & Madduppa, H. H. (2016). Length-weight relationship and population genetic of two marine gastropods species (Turbinidae: Turbo sparverius and Turbo bruneus) in the Bird Seascape Papua, Indonesia.
Biodiversitas Journal of Biological Diversity, 17(1), 208217.
Santos, M. D., Lopez, G. V., & Barut, N. C. (2010). A pilot study on the genetic variation of eastern little tuna (Euthynnus affinis) in Southeast Asia. Philippine Journal of Science, 139(1), 43-50.
Satiyarti, R. B., Nurmilah., & Rosahdi, T. D. (2017). Identifikasi fragmen DNA mitokondria pada satu garis keturunan ibu dari sel epitel rongga mulut dan sel folikel akar rambut. Biosfer: Jurnal Tadris Pendidikan Biologi, 8(1), 13-27.
Satiyarti, R. B., & Anggita, R. (2018). Endogamy
marriage mitochondrial DNA variation of north Cigintung Garut isolates. Biosfer: Jurnal Tadris
Pendidikan Biologi, 9(1), 72-83.
Sulistyaningsih, R. K., Wujdi, A., & Nugraha, B. (2016). Distribusi panjang dan estimasi total tangkapan tuna sirip biru selatan (Thunnus maccoyii) pada musim pemijahan di Samudera Hindia. Jurnal Penelitian Perikanan Indonesia, 20(4), 215-224.
Tangke, U., Karuwal, J. W. C., Mallawa, A., &
Zainuddin, M. (2017). Analisis hubungan suhu
permukaan laut, salinitas, dan arus dengan hasil tangkapan ikan tuna di perairan bagian barat Pulau Halmahera. Jurnal IPTEKS Pemanfaatan Sumberdaya Perikanan, 3(5), 368-382.
Tamura, K., Peterson, D., Peterson, N., Stecher, G., Nei, M., & Kumar, S. (2011). MEGA5: molecular
evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods. Molecular biology and evolution, 28(10), 2731-2739.
Wardani, M. T., Kusdiyantini, E., & Budiharjo, A. (2017). Identifikasi isolat Monascus sp. hasil isolasi angkak berdasarkan gen internal transcribed spacer (ITS) dan pengukuran kandungan pigmen. Jurnal Biologi, 6(2), 34-40.
Wirdateti,. & Semiadi, G. (2017). Variasi genetik trenggiling sitaan di Sumatra, Jawa, dan Kalimantan berdasarkan control region DNA mitokndria. Jurnal Veteriner, 18(2), 181-191.
Yahya, E., Rosyid, A., & Suherman, A. (2013). Tingkat pemanfaatan fasilitas dasar dan fungsional dalam strategi peningkatan produksi di Pelabuhan Perikanan Pantai Tegalsari Kota Tegal Jawa Tengah. Journal of Fisheries Resources Utilization Management and Technology, 2(1), 56-65.
Yaskun, M., & Sugiarto, E. (2017). Potensi hasil
perikanan laut terhadap kesejahteraan nelayan dan masyarakat di Kabupaten Lamongan. Juirnal Studi Manajemen dan Bisnis. 4(1), 257-264.
© 2021 by the authors; licensee Udayana University, Indonesia. This article is an open access article distributed under the terms and conditions of the Creative Commons Attribution license (http://creativecommons.org/licenses/by/3.0/).
J. Mar. Aquat. Sci. 7: 176-183 (2021)
Discussion and feedback