PERBANDINGAN KUALITAS DNA DENGAN MENGGUNAKAN DUA METODE BOOM MODIFIKASI PADA ISOLAT Mycobacterium tuberculosisP10 DI BALI
on
Perbandingan Kualitas DNA Dengan Menggunakan Dua Metode Boom Modifikasi Pada Isolat
Mycobacterium tuberculosis P10 Di Bali
(Mirawati, N. K. W., Wirajana, I.N., Yowani, S.C.)
PERBANDINGAN KUALITAS DNA DENGAN MENGGUNAKAN DUA METODE BOOM
MODIFIKASI PADA ISOLAT Mycobacterium tuberculosisP10 DI BALI
Mirawati, N.K.W.1), Wirajana, I.N.2,3), Yowani, S.C. 1,3)
1)Jurusan FarmasiFakultas Matematika dan Ilmu Pengetahuan AlamUniversitas Udayana
2)Jurusan Kimia, Fakultas Matematika dan Ilmu Pengetahuan Alam Universitas Udayana 3)Kelompok Studi MDR & XDR-TB Fakultas Matematika dan Ilmu Pengetahuan AlamUniversitas
Udayana
Korespondensi: Mirawati, N.K.W.
Jurusan Farmasi-Fakultas Matematika dan Ilmu Pengetahuan Alam-Universitas Udayana
Jalan Kampus Unud-Jimbaran, Jimbaran-Bali, Indonesia 80364 Telp/Fax: 0361-703837
Email: wikamira@ymail.com
Abstrak
Isolasi DNA bakteri merupakan salah satu metode penunjang yang dapat digunakan untuk mendeteksi bakteri penyebab infeksi. Metode yang umum digunakan dalam mengisolasi DNA Mycobacterium tuberculosis adalah metode Boom. Metode ini masih perlu mengalami modifikasi agar pelepasan DNA menjadi lebih optimal serta mengurangi inhibitor pada proses PCR. Tujuan dari penelitian ini adalah untuk membandingkan kualitas DNA yang dihasilkan dari masing-masing metode Boom modifikasi dalam mengisolasi DNA M. tuberculosis. Hasil isolasi ini nantinya akan diamplifikasi dengan teknik PCR sehingga bakteri dapat terdeteksi dengan cepat.Visualisasi produk PCR akan dianalisis dengan menggunakan 1,5 % gel agarosa. Dalam penelitian ini, dibandingkan 2 metode Boom yang telah dimodifikasi. Hasil yang diperoleh diketahui bahwa pita DNA produk PCR terlihat tebal pada masing-masing metode. Kemudian dilanjutkan analisis untuk mengetahui ketebalan pita yang dihasilkan dengan menghitung area pita. Hasil yang diperoleh diketahui bahwa metode Boom modifikasi yang dikerjakan di Laboratorium Biomolekular FK Unud lebih baik dibandingkan dengan metode Boom modifikasi yang dikerjakan oleh Traore.
Kata Kunci: Tuberkulosis, Isolasi DNA, Metode Boom, Kualitas DNA
-
1. Latar Belakang
Tuberkulosis (TB) adalah penyakit menular yang disebabkan oleh bakteri Mycobacterium tuberculosis.Pada tahun 2012,diperkirakan8,6 jutaorangterinfeksi TBdan1,3 jutameninggal akibat penyakit ini (termasuk 320.000 kematian dengan HIV-positif) (WHO, 2013).
Metode konvensional yang biasa digunakan dalam membantu penegakan diagnosis pada pasien adalah uji mikroskopik BTA (Bakteri Tahan Asam) terhadap apusan dahak suspek TB. Metode ini merupakan gold standard untuk deteksi awal TB selain uji biakan kultur bakteri pada media sediaan yang sesuai (Murrayet al., 2005).
Kelemahan uji mikroskopik BTA terletak pada jumlah bakteri yang dibutuhkan untuk dapat teridentifikasi positif, yaitu minimal 104bakteri per ml dahak. Sehingga metode ini umumnya hanya efektif terhadap pasien yang sudah memiliki manifestasi klinis (Boehmeet al., 2007). Diketahui bahwa manisfestasi klinis TB perlu waktu hampir satu bulan atau bahkan lebih sebelum dapat menimbulkan respon imunitas selular dan jumlah bakteri dapat ditemukan dalam jumlah cukup banyak di dalam dahak (Nester et al., 2007; Gantzet al., 2006). Hal ini menjadi keterbatasan uji mikroskopis BTA di daerah endemik yang sering menjadi penghambat terapi dini penangan kasus tuberkulosis (Forbeset al., 2007).
Sejak berkembangnya teknik molekular, berbagai cara dilakukanuntuk mendeteksi M.tuberculosis. Salah satu teknik yang dilakukan adalah dengan menggunakan PCR (Polymerase Chains Reactions). Namun sebelum melakukan identifikasi dengan PCR, DNA genom harus diisolasi terlebih dahulu.Isolasi DNA pada umumnya cukup melelahkan karena membutuhkan waktu hingga beberapa jam bahkan beberapa hari tergantung metode yang digunakan. Selain itu biaya yang harus dikeluarkan untuk pembelian reagen maupun kit komersial isolasi DNA juga tidak murah (Sunarno,2013).
Metode Boom adalah metode pilihan yang telah lama digunakan dalam beberapa diagnosis laboratorium karena memberi hasil yang cepat dan sederhana. Namun demikian metode ini masih memerlukan beberapa modifikasi pada beberapa langkah kerjanya dalam mengisolasi DNA M.tuberculosis. Modifikasi dilakukan dengan tujuan mengoptimalkan pelepasan DNA bakteri dan mengurangi inhibitor dalam PCR serta hilangnya DNA target pada proses isolasi (Traore et al, 2006).
Berdasarkan latar belakang tersebut, maka perlu dilakukan perbandingan terhadap metode Boom yang telah dimodifikasi, sehingga dapat diketahui metode Boom modifikasi mana yang memberi kualitas terbaik.
-
2. BAHAN DAN METODE
-
2.1 Bahan Penelitian
-
Bahan yang digunakan dalam penelitian ini adalah isolat P10 MDR-TB, larutan pelisis L6 (GuSCN, Tris-HCl, EDTA dan Triton-X),buffer L2 (GuSCN, Tris-HCl),suspensi diatom, etanol 70%, aseton, akuadest, buffer TE(Tris-EDTA) pH 8. Gel agarosa 1,5% (Promega), marker 100 bp DNA ladder,TBE (Tris-Boric Acid-EDTA) (Invitrogen) dan etidium bromide (Promega).
-
2.2 Alat Penelitian
Alat yang digunakan dalam penelitian ini adalah pipet mikro (BioRad), tip pipet mikro, microtube, biological safety cabinet class II
(BSC ESCO® COS II Type 2A), vortex (Maxi Mix II®),shakerrotator (HEALTH® tipe H-SR-200), sentrifuse (Biofuge Primo R), microwave,inkubator (BINDER®),lemari pendingin, thermalcycler (Applied Biosystem Veriti 96®), alat elektroforesis (comb, tray dan chamber elektroforesis) dan UV transiluminator Gel Doc XR System (BioRad®).
-
2.3 Prosedur Penelitian
-
2.3.1 Isolasi DNA
-
-
a. Isolasi DNA MTB dengan Metode Boom Modifikasi Laboratorium Biomolekuler FK Unud (Unpublish)
Metode ini merupakan metode yang telah dikerjakan oleh Laboratorium Biomolekuler Fakultas Kedokteran Universitas Udayana dan dalam pengerjaannya telah dilakukan modifikasi pada beberapa tahapan kerja meliputi penggunaan jumlah isolat, jumlah buffer, kecepatan dan waktu sentrifugasi serta cara dan waktu pengeringan pelet dari metode Boom original. Pada penelitian ini dilakukan pengulangan sebanyak 3 kali pada isolat P10.
Isolasi dilakukan dengan memipet sebanyak 100 µL sampel dan 40 µL suspensi diatom dimasukkan ke dalam tabung effendorf yang berisi 1 mL buffer L6. Dihomogenkan dengan vorteks selama beberapa detik kemudian dikocok menggunakan shaker dengan kecepatan 100 rpm selama 15 menit. Larutan ini disentrifugasi selama 1 menit dengan kecepatan 12.000 rpm dan supernatan yang diperoleh dibuang sehingga diperoleh pelet diatom DNA. Pelet diatom DNA kemudian ditambahkan dengan washing buffer L2 sebanyak 0,5 mL dan disentrifugasi selama 1 menit dengan kecepatan 12.000 rpm. Supernatan yang diperoleh dibuang kembali (langkah tersebut diulangi 1x). Pelet yang diperoleh kemudian dicuci dengan etanol 70% sebanyak 1 mL, disentrifugasi selama 1 menit dengan kecepatan 12.000 rpm, kemudian supernatan dibuang kembali. Pelet diatom dicuci kembali dengan aseton sebanyak 1 mL, disentrifugasi selama 1 menit dengan kecepatan 12.000 rpm, kemudian supernatan dibuang. Pelet yang diperoleh dikeringkan dalam waterbath suhu 56ºC selama 15 menit. Aquadest steril
ditambahkan sebanyak 50 µL, dihomogenkan dengan vortex, kemudian diinkubasi selama 10 menit pada suhu 56ºC. Divorteks kembali dan kemudian disentrifugasi selama 5 menit dengan kecepatan 12.000 rpm. Supernatan dipipet dan dipindahkan ke dalam tabung mikro steril baru sehingga diperoleh DNA kromosomal. Proses ini dilakukan di dalam Biological safety cabinet class II.
-
b. Isolasi DNA MTB dengan Metode Boom
Modifikasi (Traore et al, 2006).
Pada penelitian yang telah dikerjakan oleh Traore et al (2006), dilakukan modifikasi metode Boom original yaitu modifikasi pada jumlah larutan dan buffer yang digunakan, lama sentrifugasi serta perlakuan lainnya seperti penghomogenan dengan shaker serta jumlah pencucian menggunakan etanol. Pada penelitian ini dilakukan pengulangan sebanyak 3 kali pada masing-masing isolat.
Proses ekstraksi dilakukan dengan menambahkan 50 μL spesimen kedalam 1,5 mL tabung eppendorf yang mengandung 900 μL buffer L6 dan 40 μL dari suspensi diatom. Tabung kemudian dihomogenkan dengan divorteks selama 5 detik, dicampur perlahan selama 10 menit pada temperatur ruangan pada horizontal shaker dan kemudian disentrifugasi pada 12,000 x g selama 8 menit. Supernatan yang diperoleh kemudian dipisahkan, dan pelet (endapan) yang didapatkan dicuci dengan menambahkan 1 mL buffer L2, kemudian spin dan sentrifugasi pada 12.000 xg selama 15 detik. Buang supernatant dan sisakan pellet (langkah tersebut diulangi 1x). Pellet lalu ditambahkan dengan 1 mL etanol 70%, vortex dan sentrifugasi pada 12.000 xg selama 15 detik. Buang kembali supernatant dan sisakan pellet. Pellet kemudian ditambahkan dengan 1 mL aseton, vortex dan sentrifugasi pada 12.000 xg selama 15 detik. Supernatan yang diperoleh kemudian dibuang. Pelet diambil dan dikeringkan selama 15 menit pada temperatur ruangan dengan cara membuka tutup tabung. DNA yang didapat kemudian dilarutkan dengan mensuspensikan kembali pelet dalam 125 μL TE buffer (10μM Tris, 1 mM EDTA pH 8,0) diikuti dengan inkubasi pada 560C selama 10 menit dan disentrifugasi selama 2
menit pada 12,000 xg. Supernatandipipet dan dipindahkan ke dalam tabung mikro steril baru sehingga diperoleh DNA kromosomal. Proses ini dilakukan di dalam Biological safety cabinet class II.
-
2.3.2 Amplifikasi DNA Hasil Isolasi dengan Polymerase Chains Reactions (PCR) PCR dilakukan dengan alat thermalcycle dengan menggunakan sepasang primer
oligonukleotida yaitu terdiri atas primer forward (mabA-inhA-promoter-FS) dengan urutan
5’ACATACCTGCTGCGCAAT3’ dan primer reverse (mabA-inhA-promoter-R) dengan urutan yaitu 5’CTCCGGTAACCAGGACTGAA3’ (Chen et al., 2011).Proses ini dilakukan dengan tahapan yaitu predenaturasi 950 C selama 15 menit, 45 siklus amplifikasi (940 C selama 1 menit, 540 C selama 1 menit 20 detik ,720 C selama 1 menit 10 detik) dan elongasi akhir pada 720 C selama 10 menit.
-
2.3.3 Deteksi Hasil Isolasi dan Produk PCR Dengan Elektroforesis
DNA hasil isolasi dan produk PCR dielektroforesis pada 1.5% agarosa yang dilarutkan dalam 1.0X TBE (Tris-borat-EDTA). Sebanyak 3 µL sampel hasil PCR dan hasil isolasi serta 3 µL DNA ladder 100 bp dimasukkan ke dalam sumur yang terbentuk dari sisir. Kemudian dielektroforesis dengan running buffer TBE (Tris-Boric Acid-EDTA) 1X pada tegangan 65 volt selama 35 menit. Hasil elektroforesis kemudian divisualisasi pada alat UV Transiluminator (Gel Doc).
-
3. HASIL
Sampel yang digunakan dalam penelitian ini adalah isolat M.tuberculosis P10. Isolat P10 ini diperoleh dari Instalasi Mikrobiologi Klinik Rumah Sakit Umum Pusat (RSUP) Sanglah Denpasar. Berikut adalah elektroforegram dari produk PCR masing-masing metode dapat dilihat pada gambar 1
2072 bp
1500 bp
600 bp
100 bp
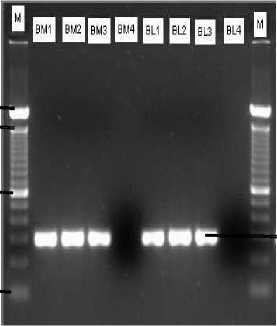
300 bp
Gambar 1.Elektroforegram produk PCR isolat M.tuberculosis P10 menggunakan modifikasi masing-masing metode Boom, M: Marker DNA Ladder 100 bp; BM1-BM3 (pita produk PCR Boom Modifikasi Traore); BL1-BL3 (pita produk PCR Boom modifikasi Laboratorium Biomolekular FK Unud).
-
4. PEMBAHASAN
Isolasi DNA adalah serangkaian proses untuk memisahkan DNA dari komponen-komponen lainnya. Hasil isolasi tersebut merupakan tahapan penting untuk langkah berikutnya dan harus dilakukan dengan baik dan bebas kontaminasi.
Untuk mengetahui kualitas DNA yang dihasilkan, maka dilakukan amplifikasi DNA hasil isolasi dengan menggunakan teknik PCR. Keberhasilan amplifikasi DNA dengan PCR ditentukan oleh beberapa faktor yaitu : kemurnian dan konsentrasi komponen dalam larutan premix PCR, primer oligonukleotida gen, konsentrasi [α-32P]dCTP, jumlah dan kemurnian DNA sampel sputum, faktor teknis dan non teknis misalnya kontaminasi (Purnami,2009). Dari hasil pada gambar 1, dapat dilihat pita-pita yang tebal pada kedua metode tersebut. Hal ini dikarenakan adanya proses amplifikasi DNA sehingga jumlah DNA akan berlipat-lipat dari jumlah templat yang ditambahkan. Pita ini kemudian akan
tervisualisasii dengan jelas karena adanya penambahan etidhium bromide (EtBr) pada gel elektroforesis. EtBR akan berinterkalasi diantara basa-basa nitrogen dari nukleotida sehingga akan membuat basa merenggang dan basa-basa nukleotida akan memberi serapan pada sinar UV. Proses ini akan membuat DNA tampak berpendar dan tervisualisasi pada gel elektroforesis.
Kualitas dari kedua metode Boom modifikasi yang terlihat pada gambar 1, menunjukkan kemampuan masing-masing metode modifikasi dalam mengisolasi DNA. Proses lisis dari metode Boomdidasarkan pada kemampuan lisis dari sel serta proses inaktivasi nuklease dengan menggunakan ”chaotropicagent” yaitu guanidium thiocyanate (GuSCN) yang terkandung dalam buffer lisis L6.GuSCN merupakan reagensia yang kuat untuk memurnikan dan mendeteksi DNA dan RNA karena potensinya dalam melisis sel dan menonaktifkan nuklease (Lina et al., 2007).
Untuk memastikan kualitas DNA yang dihasilkan dari masing-masing metode, maka dilakukan pengukuran tebal pita yang tervisualisasi. Pita-pita ini diukur dengan menggunakan aplikasi Image-J. Hasil pengukuran menunjukkan area atau ketebalan pita serta persentase yang dihasilkan masing-masing pita seperti yang terangkum pada Tabel 1.
Tabel 1. Hasil Pengukuran Area dan Persentase Ketebalan Pita Produk PCR
|
No |
Pita* |
Area |
Persentase |
|
1 |
M |
30236,567 |
37,588 |
|
2 |
BM 1 |
7958,669 |
9,894 |
|
3 |
BM 2 |
8989,569 |
11,175 |
|
4 |
BM 3 |
8056,619 |
10,016 |
|
5 |
BL1 |
8127,326 |
10,103 |
|
6 |
BL2 |
8990,690 |
11,177 |
|
7 |
BL3 |
8081,962 |
10,047 |
Keterangan*:
M : Marker DNA Ladder 100 bp ; BM1, BM2 dan BM3 (pita produk PCR Boom Modifikasi Traore pada 3 kali pengulangan); BL1, BL2 dan BL3 (pita produk PCR Boom modifikasi Laboratorium Biomolekular FK Unud pada 3 kali pengulangan).
Dari tabel diatas, diketahui bahwa pita yang lebih tebal akan memberi luas area yang lebih besar maka persentase yang dihasilkan juga akan semakin besar. Berdasarkan data tersebut, dapat diketahui bahwa metode Boom modifikasi yang dikerjakan di Laboratorium Biomolekular FK Unud lebih tebal pada ketiga hasil pengulangan dibandingkan dengan metode Boom modifikasi yang dikerjakan oleh Traore et al.
-
5. KESIMPULAN
Metode Boom modifikasi yang dikerjakan oleh Laboratorium Biomolekular FK Unud (unpublish) lebih baik dari segi kualitas DNA yang dihasilkan dibandingkan dengan metode Boom modifikasi yang dikerjakan oleh Traore et al (2006).
UCAPAN TERIMAKASIH
Terimakasih kami ucapkan kepadaKepala Laboratorium Mikrobiologi Klinik RSUP Sanglah yang telah menyediakan isolat MDR-TB serta kepada seluruh staff Laboratorium Biomolekular Fakultas Kedokteran Universitas Udayana.
DAFTAR PUSTAKA
Boehme, C.C., et al. 2007. Operational Feasibility of UsingLoop-Mediated Isothermal Amplificatin for Diagnosis ofPulmonary Tuberculosis in Microscopy Centers of Developing Countries. Journal of Clinical. Microbiology. 45: 1936 -1940.
Chen, X.,Kong, F.,Wang., Li, C.,Zhang.,and Gilbert, G.L. 2011. Rapid Detection of Isoniazid, Rifampin, and Ofloxacin Resistance inMycobacterium tuberculosis Clinical Isolates UsingHigh-Resolution Melting Analysis. Journal Of Clinical Microbiology, 49(10):3450–3457.
Forbes, B., Sahm, D.F., and Weissfeld, A.S. 2007. Bailey &Scott's: Diagnostic
Microbiology, twelfth edition. Philadelphia : MOSBYElsevier.p: 509478.
Gantz, N.M., Brown, R.B., Berk, S.L., and Myers, J.W. 2006.Manual of Clinical Problems in Infectious Disease: Role ofTuberculin Test. 5t' ed. Philadelphia: Lippincott Williams &Wilkins. pp407-411.
Lina, R., Budiman, B., dan Mukh, S.. 2007. Deteksi Gen Target INH pada DNA Sputum Basil Tahan Asam Positif dengan Teknik Polymerase Chain Reaction. Majalah Kedokteran Indonesia, 57(8): 245-250.
Murray, P.R., Rosenthal, K.S., Pfaller, M.A. 2005. Medical Microbiology: Mycobacterium. 5th ed. Philadelphia: Elsevier Mosby. 297-301
Nester, E.W., Anderson, D.G., Roberts, Jr., C.E. 2007.Microbiology: A Human
Perspective. 5th ed. New York: McGraw Hill. 245-263.
Purnami, S., Syaifudin, M., Dan Giyatmi. 2009. Penandaan Dna Dengan 32pUntuk Deteksi Resistensi MycobacteriumTuberculosis Terhadap Isoniazid. Jfn, 3(1): 12-18.
Sunarno, Rizki, A.,Sariadji, K., Malik, Amarila., Karuniawati, Anis., dan Soebandrio, A. 2013. Direct Polymerase Chain Reaction: Sebuah Alternatif Metode Diagnostik Difteri Secara Cepat, Mudah dan Hemat. Makara Seri Kesehatan, 17(2):88-94.
Traore, H., Armand, V.D., Isdore, C.S., Leen, R., dan Prancoise, P. 2006. Direct Detection of Mycobacterium tuberculosis Complex DNA and Rimfampin Resistance In Clinical Specimens From Tuberculosis Patients by Line Probe Assay. Journal Of Clinical Microbiologi, 2004:4384-4388.
WHO. 2013. Global Tuberculosis Report. Switzerland: WHO Press. p 1-6.
49
Discussion and feedback