MOLECULAR DOCKING SENYAWA XANTON, BENZOFENON, DAN TRITERPENOID SEBAGAI ANTIDIABETES DARI EKSTRAK TUMBUHAN GARCINIA COWA
on
JURNAL KIMIA (JOURNAL OF CHEMISTRY) 16 (1), JANUARI 2022 DOI: https://doi.org/10.24843/JCHEM.2022.v16.i01.p10
p-ISSN 1907-9850 e-ISSN 2599-2740
MOLECULAR DOCKING SENYAWA XANTON, BENZOFENON, DAN TRITERPENOID SEBAGAI ANTIDIABETES DARI EKSTRAK TUMBUHAN GARCINIA COWA
I. R. Hartanti1*, A. A. Putri1, N. N. Auliya AS1, A. L. Triadenda1, E. Laelasari1, C. Suhandi1 dan M. Muchtaridi2
1Program Studi Farmasi, Fakultas Farmasi, Universitas Padjadjaran, Sumedang, Indonesia 2Departemen Analisis Farmasi dan Kimia Medisinal, Fakultas Farmasi, Universitas Padjadjaran, Sumedang, Indonesia
*Email: intan19007@mail.unpad.ac.id
ABSTRAK
Diabetes mellitus tipe 2 disebabkan oleh ekspresi PTP1B yang tinggi dan mempengaruhi aktivitas PTKs, yang menyebabkan insulin gagal berikatan dengan reseptor insulin dan menginduksi resistensi insulin. Senyawa xanton dan benzofenon merupakan senyawa yang telah diketahui memiliki aktivitas farmakologi sebagai antidiabetes. Salah satu tanaman dengan kandungan senyawa tersebut adalah Garcinia cowa. Penelitian ini dilakukan dengan tujuan untuk mengetahui afinitas dan mekanisme inhibisi PTP1B oleh senyawa turunan xanton, benzofeno, dan triterpenoid dalam Garcinia cowa antara lain garcinia cowone K, guttiferone I, 1,7-dihydroxyxanthone, 1-hydroxyl-7-methoxyxanthone, mangostinone, α-mangostin, cowanol, gacibiphenyl C, friedelin, β-friedelinol, dan oleanane-12-ol secara in silico dengan molecular docking serta melakukan studi pre-ADMET terhadap senyawa tersebut. Molecular docking dilakukan melalui beberapa tahap diantaranya preparasi dan optimasi struktur 3D senyawa uji, preparasi struktur 3D reseptor PTP1B, validasi metode, dan docking senyawa uji dengan PTP1B. Hasil yang diperoleh dari docking senyawa uji dengan reseptor PTP1B berupa energi ikatan, konstanta inhibisi (KI), dan ikatan hidrogen. Semakin rendah nilai energi ikatan menunjukkan ikatan antara protein dan ligan yang dihasilkan semakin stabil. Hasil penelitian menunjukkan energi ikatan dan KI PTP1B dengan native ligand berturut-turut sebesar -10,07 kkal/mol dan 0,0417 µM. Sementara dengan senyawa α-mangostin berturut-turut sebesar -8,91 kkal/mol dan 0.29317 µM. Hal tersebut menunjukkan bahwa senyawa α-mangostin memiliki potensi sebagai antidiabetes mellitus tipe 2 dengan menghambat PTP1B. Selain itu, senyawa α-mangostin juga memiliki profil ADMET yang baik.
Kata kunci: antidiabetes, diabetes mellitus tipe 2, penambatan molekuler, PTP1B, Xanton
ABSTRACT
Type 2 diabetes mellitus is caused by high PTP1B expression and affects the activity of PTKs, which causes insulin to fail to bind to insulin receptors, and induces insulin resistance. Xanthones and benzophenones are compounds that have been known to have pharmacological activity as antidiabetic. One of the plants containing these compounds is Garcinia cowa. This study aims to know the affinity and inhibition mechanism of PTP1B by xanthones, benzophenones, and triterpenoid in Garcinia cowa, including garcinia cowone K, guttiferone I, 1,7-dihydroxyxanthone, 1-hydroxyl-7 methoxyxanthone, mangostinone, α-mangostin, cowanol, gacibiphenyl C, friedelin, β-friedelinol, and oleanane-12-ol in silico by molecular docking and conducted a pre-ADMET study of these compounds. Molecular docking is carried out in several steps including preparation and optimization of the 3D structure of the compound, preparation of the PTP1B receptor 3D structure, method validation, and docking of the compound with PTP1B. The results obtained from the docking of the compound with the PTP1B receptor appears in the form of bond energies, inhibition constant (IC), and hydrogen bonds. The lower the bond energy value, the more stable the bond between the protein and the resulting ligand is. The results showed that the bond energy and IC value of PTP1B with the native ligand is -10,07 kcal/mol and 0,0417 µM. Meanwhile the α-mangostin compound is -8,91 kcal/mol and 0,29317 µM. It shows that the α-mangostin has potential as an antidiabetic mellitus type 2 agent by inhibiting PTP1B. In addition, α-mangostin also shows a good ADMET profile.
Keywords: antidiabetic, molecular docking, PTP1B, type 2 diabetes mellitus, xanthones
PENDAHULUAN
Diabetes mellitus tipe 2 merupakan suatu kelainan dari metabolisme kronik yang disebabkan oleh kerusakan sekresi insulin dan aksi insulin. Jika telah berkembang sepenuhnya secara klinis maka akan ditandai dengan hiperglikemia puasa dan postprandial, aterosklerosis dan penyakit vaskular mikroangiopati (Fatimah, 2015; Olokoba et al., 2021).
Diabetes mellitus tipe 2 merupakan penyakit hiperglikemia yang diakibatkan oleh insensitivitas terhadap insulin, terjadi ketika kondisi kadar insulin mungkin sedikit menurun atau bisa dikatakan dalam keadaan rentang normal. Diabetes mellitus tipe 2 dianggap sebagai non insulin dependent diabetes mellitus dikarenakan insulin tetap dihasilkan oleh sel-sel beta pankreas. Umumnya faktor diabetes mellitus tipe 2 yaitu karena peningkatan prevalensi gaya hidup yang tidak banyak bergerak dan obesitas (Olokoba et al., 2021).
Penyakit diabetes mellitus tipe 2 terjadi lebih tinggi pada wanita dibandingkan dengan pria, dikarenakan indeks massa tubuh wanita peluang peningkatannya lebih besar. Pada hasil riset kesehatan dasar pada tahun 2008, menunjukan prevalensi penyakit diabetes mellitus tipe 2 di Indonesia meningkat hingga mencapai 57%, dilihat pada tahun 2012 angka kejadian diabetes mellitus di dunia sebanyak 371 juta jiwa dimana keseimbangan diabetes mellitus tipe 2 adalah 95% dari populasi dunia yang menderita penyakit diabetes mellitus dan yang menderita diabetes mellitus tipe 1 hanya 5% (Fatimah, 2015).
Disfungsi dari sel β-pankreas merupakan proses dasar dari patogenesis diabetes melitus tipe 2. Insulin disekresikan oleh sel β-pankreas dan berfungsi dalam regulator utama homeostasis glukosa melalui jalur pensinyalan yang kompleks. Insulin reseptor merupakan sejenis glikoprotein transmembran yang terdiri dari 2 alpha dan beta subunits yang dihubungkan oleh ikatan 3 sulfida. Alpha subunits terletak di sel lateral sedangkan beta subunits terletak di seberang sel membran. Insulin yang terikat pada reseptor alpha subunit menginduksi fosforilasi dari residu tirosin dan protein tyrosine kinase (PTK) dari beta subunits dan menghasilkan serangkaian reaksi fosforilasi dan defosforilasi kaskade termasuk
mitogen-activated protein kinases (MAPK) dan phosphoinositide 3-kinase (PI3K) untuk mengatur metabolisme. Ketika konsentrasi insulin melebihi konsentrasi fisiologi atau bisa disebut juga hiperinsulinemia, insulin akan mendorong sel untuk berproliferasi dan berkembang. Ketika proses penggabungan dari insulin dan reseptornya, protein tyrosine phosphatase 1B (PTP1B) mengkatalisis reseptor insulin dan substrat reseptor insulin untuk berfosforilasi, mengkoordinasikan keseimbangan antara fosforilasi dan defosforilasi oleh residu tirosin. Di samping itu, PTP1B dapat melakukan defosforilasi janus kinase 2 (JAK2) yang sudah teraktivasi dan signal transducer and activator of transcription 3 (STAT3) dan mencegah transduksi sinyal leptin. Ekspresi yang tinggi dari PTP1B memengaruhi aktivitas PTKs, yang menyebabkan insulin gagal berikatan dengan reseptor insulin, dan menginduksi resistensi insulin dan mengakibatkan diabetes melitus tipe 2 (Sun et al., 2016).
Salah satu pengobatan untuk diabetes tipe 2 adalah menggunakan trodusquemine. Beberapa dosis trodusquemine dapat meningkatkan toleransi glukosa pada orang yang mengidap diabetes melitus tipe 2. Trodusquemine adalah senyawa yang menekan nafsu makan dan merupakan glukoregulator yang bekerja secara sentral/perifer. Trodusquemine merupakan inhibitor PTP1B yang sangat selektif, yang berperan penting dalam mengontrol fungsi leptin dan jalur insulin. Dengan menghambat PTP1B, trodusquemine dapat menurunkan nafsu makan dan menormalkan gula darah, menghasilkan penurunan berat badan yang konsisten dan berkelanjutan serta meningkatkan sensitivitas insulin (American Diabetes Association, 2009).
Senyawa xanton, benzofenon, dan triterpenoid merupakan senyawa yang telah diketahui memiliki aktivitas farmakologi sebagai antidiabetes. Salah satu tanaman dengan kandungan senyawa tersebut adalah Garcinia cowa. Tanaman ini merupakan bagian dari famili Guttiferae dan berasal dari Thailand. Namun, tanaman ini juga dapat ditemukan di Indonesia yang dikenal sebagai asam kandis (Heyne 1987). Berdasarkan studi yang telah dilakukan, ekstrak kasar tanaman Garcinia cowa ini memiliki aktivitas antidiabetes dengan kandungan xanton,
benzofenon, dan triterpenoid yaitu garcinia cowone K, guttiferone I, 1,7-dihydroxyxanthone, 1-hydroxyl-7 methoxyxanthone, mangostinone, α-mangostin, cowanol, gacibiphenyl C, serta dengan kandungan triterpenoid yaitu friedelin, β-friedelinol, dan oleanane-12-ol (Phukhatmuen et al., 2020). Salah satu senyawa yang terkandung dalam Garcinia cowa, yaitu α-mangostin telah diketahui memiliki khasiat sebagai antidiabetes pada penelitian secara in vivo (Ratwita et. al., 2019). Namun, belum ada penelitian yang menunjukkan adanya aktivitas spesifik terhadap protein PTP1B.
Dengan demikian, penelitian ini bertujuan untuk menganalisis potensi aktivitas dari senyawa turunan xanton, benzofenon, dan triterpenoid yang terkandung dalam Garcinia cowa sebagai antidiabetes yang spesifik terhadap protein PTP1B melalui studi in silico (virtual screening). Studi in silico (virtual screening) ini umum digunakan untuk memprediksi pengikatan ligan dari database dalam jumlah besar pada target/reseptor tertentu yang bertujuan untuk mengidentifikasi senyawa yang paling berpotensi dari database tersebut yang dapat digunakan untuk studi selanjutnya. Oleh karena itu, studi secara in silico ini sangat diperlukan.
MATERI DAN METODE
Bahan
Struktur 2D dari senyawa garciniacowone K (1), guttiferone I (2), 1,7-dihydroxyxanthone (3), 1-hydroxyl-7 methoxyxanthone (4), mangostinone (5), α-mangostin (6), cowanol (7), gacibiphenyl C (8), friedelin (9), β-friedelinol (10), oleanane-12-ol (11), trodusquemine (12) sebagai standar dan p-nitrophenyl phosphate (13) sebagai substrat yang disiapkan menggunakan aplikasi ChemDraw kemudian diubah ke bentuk 3D dan dioptimasi menggunakan aplikasi Chem 3D, serta struktur 3D dari protein PTP1B (PDB ID: 1ECV) diunduh pada website Protein Data Bank yaitu https://www.rcsb.org/structure/1ECV.
Peralatan
Hardware yang digunakan berupa komputer dengan spesifikasi AMD A9-9420 RADEON R5 3.00 GHz RAM 4.00 GB
dengan software Windows 10 64-bit, ChemDraw Pro 12.0, Chem3D Pro 12.0, Autodock 4.2.6., Autodock Tools 1.5.6. dan BIOVIA Discovery Studio 2020.
Cara Kerja
Prediksi Farmakokinetik dan Toksisitas
Prediksi farmakokinetik (absorbsi, distribusi, metabolisme) dan toksisitas meliputi Human Intestinal Absorption (HIA), Caco2, Plasma Protein Binding (PPB), Blood-Brain Barrier (BBB), potensi mutagen, dan karsinogenik yang dilakukan melalui situs PreADMET (https://preadmet.bmdrc.kr/) dengan menggambarkan masing-masing senyawa uji.
Preparasi Ligan Uji
Sebanyak tiga belas ligan yang akan digunakan masing-masing dibuat struktur 2D-nya terlebih dahulu menggunakan software ChemDraw Pro 12.0. Kemudian dibangun struktur 3D-nya dan dilakukan optimasi geometri mekanika molekul (MM) menggunakan software Chem3D Pro 12.0. Struktur ligan uji yang telah dioptimasi kemudian disimpan dengan format file PDB. Masing-masing ligan uji kemudian dipreparasi menggunakan AutoDockTools 1.5.6. dengan cara mengoreksi struktur senyawa dan menambahkan muatan Gasteiger ke dalamnya. Hasil preparasi kemudian disimpan sebagai berkas dengan format *pdbqt.
Preparasi Reseptor
Struktur 3D protein PTP1B yang diunduh dari Protein Data Bank ditampilkan dengan software BIOVIA Discovery Studio. Molekul air di sekitar protein dihapus kemudian rantai protein dipisahkan dari native ligand dengan menghapus bagian tersebut dan juga molekul air kemudian disimpan dengan format *pdb sebagai reseptor. Struktur native ligand juga diekstrak dengan menghapus bagian rantai protein dan disimpan dengan format *pdb sebagai native ligand. Protein dan native ligand kemudian masing-masing dipreparasi menggunakan software AutoDockTools 1.5.6. dengan cara menambahkan atom Hidrogen pada sisi polar struktur dan menambahkan muatan Kollman ke dalamnya. Hasil preparasi kemudian disimpan sebagai berkas dengan format *pdbqt.
Validasi Metode Molecular Docking
Validasi metode dilakukan untuk memastikan parameter docking sudah valid untuk dilakukan pada proses docking ligan uji terhadap protein PTP1B. Validasi ini dilakukan dengan re-docking atau menambatkan kembali native ligand yang sebelumnya telah dipisahkan ke dalam protein PTP1B. Proses redocking dilakukan dengan menggunakan grid box dengan ukuran 40 x 40 x 40 dengan koordinat x = 10,265; y= 45,877; z = 19,734.
Parameter docking, diantaranya nilai Genetic Algorithm (GA) diatur pada nilai sebesar 50 dan digunakan Lamarckian GA 4.2 sebagai algoritma luaran hasil docking. Parameter docking lainnya diatur ke dalam parameter default
Parameter yang harus diperhatikan dari hasil re-docking ini adalah Root Mean Square Deviation (RMSD). Nilai RMSD yang dapat diterima adalah ≤ 3,0 A (Jain dan Nicholls, 2008).
Docking Senyawa Uji terhadap PTP1B
Masing-masing dari dua belas senyawa uji yang telah dioptimasi dan dipreparasi didocking-kan terhadap reseptor PTP1B yang telah dipisahkan dari native ligand-nya dan juga telah dipreparasi menggunakan AutodockTools 1.5.6. dengan tahapan docking yang sama ketika melakukan validasi metode. Parameter yang harus diperhatikan pada hasil docking senyawa uji pada PTP1B adalah energi ikatan dan ikatan hidrogen. Visualisasi hasil docking dilakukan menggunakan software BIOVIA Discovery Studio untuk dilihat bentuk interaksi antara senyawa uji dengan residu asam amino pada reseptor dalam bentuk 3D maupun 2D.
HASIL DAN PEMBAHASAN
Prediksi Farmakokinetik dan Toksisitas
Proses uji prediksi farmakokinetik dan toksisitas bertujuan untuk memprediksi proses absorpsi, distribusi, metabolisme, dan eliminasi serta toksisitas yang terjadi di dalam tubuh manusia. Pengujian ini dilakukan secara in silico dengan parameter yaitu Human Intestinal Absorption (HIA), Caco2, Plasma
Protein Binding (PPB), Blood-Brain Barrier (BBB), dan potensi mutagen.
Hasil prediksi profil ADMET senyawa xanton dan benzofenon dari ekstrak Garcinia Cowa dapat dilihat pada Tabel 1. Prediksi absorpsi obat terdiri atas parameter HIA (Human Intestinal Absorption) dan Sel Caco2. Parameter HIA (Human Intestinal Absorption) bertujuan untuk memprediksi proses absorpsi yang terjadi di dalam usus. Hasil parameter HIA merupakan hasil dari penjumlahan bioavailabilitas dengan absorpsi yang dievaluasi dari hasil rasio ekskresi melalui urine, empedu, dan feses (Nursamsiar dkk, 2016). Kategori dari parameter HIA ini yaitu 70-100% (kategori baik); 20-70% (kategori sedang); 0-20% (kategori rendah). Sel Caco2 merupakan parameter kemampuan permeabilitas yang digunakan untuk mengetahui perpindahan obat melalui sel epitel di usus yang berasal dari adenocarcinoma usus besar manusia dengan jalur transportasi ganda dengan model secara in vitro (Nursamsiar dkk, 2016). Kategori parameter sel Caco2 yaitu >70 nm/detik (kemampuan permeabilitas senyawa tinggi; 470 nm/detik (kemampuan permeabilitas sedang); <4 nm/detik (kemampuan permeabilitas yang rendah) (Cheng et al., 2013).
Semua senyawa uji menunjukkan nilai HIA dengan kategori baik dengan hasil parameter Caco2 kategori sedang. Hasil tersebut menunjukkan bahwa 11 senyawa uji tersebut memiliki kemampuan absorpsi di dalam usus dengan baik dan memiliki kemampuan permeabilitas sedang.
Prediksi distribusi obat terdiri atas parameter PPB (Plasma Protein Binding) dan BBB (Blood Brain Barrier). Nilai %PPB lebih dari 90% menunjukkan bahwa molekul terikat kuat pada protein plasma. Apabila %PPB berada di bawah 90% menunjukkan bahwa molekul berikatan dengan lemah pada protein plasma (Kumar et al., 2018; Purwaniati, 2020). Dari 11 senyawa uji, semua senyawa memiliki nilai %PPB di atas 90%. Hal tersebut menunjukkan bahwa molekul-molekul yang ada pada senyawa tersebut berikatan dengan kuat pada protein plasma.
Tabel 1. Prediksi profil ADMET senyawa xanton dan benzofenon ekstrak Garcinia Cowa
Parameter
|
Senyawa Uji |
Absorpsi |
Distribusi |
Metabolisme dan Ekskresi |
Toksis-itas Ames Test | |||||
|
HIA (%) |
Caco2 (nm/detik) | ||||||||
|
PPB (%) |
BBB |
CYP2-C19 Inhibition |
CYP2-C9 Inhibition |
CYP2- D6 Inhibit ion |
CYP3-A4 - Inhibition | ||||
|
C1 |
98,16 |
54,1576 |
99,95 |
11,6979 |
Non |
Inhibitor |
Non |
Inhibitor |
Non Mutagen |
|
C2 |
95,76 |
26,41 |
100 |
9,27696 |
Non |
Inhibitor |
Non |
Inhibitor |
Non Mutagen |
|
C3 |
91,87 |
0,216 |
100 |
0,823 |
Inhibitor |
Inhibitor |
Non |
Inhibitor |
Mutagen |
|
C4 |
95,45 |
4,23 |
97,75 |
0,911 |
Inhibitor |
Inhibitor |
Non |
Inhibitor |
Mutagen |
|
C5 |
100 |
22,2007 |
100 |
23,1985 |
Non |
Inhibitor |
Non |
Inhibitor |
Mutagen |
|
C6 |
91,81 |
20,694 |
96,62 |
3,93794 |
Inhibitor |
Inhibitor |
Non |
Inhibitor |
Non Mutagen |
|
C7 |
90,396 |
19,1975 |
96,11 |
3,87424 |
Inhibitor |
Inhibitor |
Non |
Inhibitor |
Non Mutagen |
|
C8 |
86,508 |
21,1779 |
94,29 |
1,6839 |
Inhibitor |
Inhibitor |
Non |
Inhibitor |
Non Mutagen |
|
C9 |
100 |
46,4784 |
100 |
22,1 |
Non |
Inhibitor |
Non |
Inhibitor |
NonMutagen |
|
C10 |
100 |
46,8445 |
100 |
22,3207 |
Non |
Inhibitor |
Non |
Inhibitor |
NonMutagen |
|
C11 |
100 |
46,79 |
92,76 |
15,2747 |
Non |
Inhibitor |
Non |
Inhibitor |
NonMutagen |
|
Keterangan: C1: garciniacowone K C2: guttiferone I C3: 1,7-dihydroxyxanthone C4: 1-hydroxyl-7 methoxyxanthone C5: mangostinone |
C6: α-mangostin C7: cowanol C8: gacibiphenyl C C9: friedelin C10: β-friedelinol C11: oleanane-12-ol | ||||||||
|
Nilai prediksi BBB menunjukkan prediksi konsentrasi senyawa dapat menembus sawar darah otak. Klasifikasi penetrasi BBB berdasarkan Pre-ADMET |
yaitu nilai BBB >2.0 (high absorption to CNS); nilai BBB antara 0,1-2,0 (middle absorption to CNS); nilai BBB <0,1 (low absorption to CNS) (Ma et al, 2005). Hasil | ||||||||
nilai prediksi BBB dari 11 senyawa uji menunjukkan senyawa C1, C2, C5, C6, C7, C9, C10, dan C11 memiliki kemampuan menembus sawar darah otak yang tinggi (high absorption to CNS). Sedangkan, senyawa C3, C4, dan C8 memiliki kemampuan menembus sawar darah otak menengah (middle absorption to CNS).
Berdasarkan data prediksi metabolisme, senyawa C3, C4, C6, C7, C8 merupakan inhibitor CYP2C19. Sedangkan, senyawa C1, C2, C5, C9, C10, dan C11 bukan merupakan inhibitor CYP2C19. Semua senyawa merupakan inhibitor CYP2C9 dan CYP3A4 serta bukan merupakan inhibitor CYP2D6. Obat-obat yang merupakan inhibitor CYP2C19 dan CYP2C9 dapat meningkatkan konsentrasi protein plasma dan terkadang menimbulkan efek samping (Foti dan Wahlstrom, 2008; Van Booven et al., 2010). CYP2D6 bertanggung jawab terhadap metabolisme dari kebanyakan obat dan senyawa-senyawa kimia (Bertilsson et al., 2002). CYP2D6 ini tersebar luas di beberapa jaringan dan paling besar terdapat di organ hati (Ali et al., 2013). CYP3A4 merupakan
enzim yang paling berperan dalam metabolisme di hati yang bertanggung jawab atas proses oksidasi dari molekul organik kecil (xenobiotics), seperti racun dan obat, sehingga dapat dikeluarkan dari dalam tubuh (Dai et al., 2001; Oyesakin et al., 2018).
Prediksi toksisitas senyawa uji terdiri atas parameter Ames Test. Uji Ames bertujuan untuk mengetahui sifat mutagenik dari suatu senyawa (Prasetiawati dkk, 2021). Berdasarkan data prediksi, hanya senyawa C3, C4, dan C5 yang memiliki sifat mutagenik. Senyawa lainnya memiliki sifat non-mutagenik.
Preparasi Ligan Uji
Preparasi ligan uji dilakukan dengan menggunakan software ChemDraw Pro 12.0 untuk membuat struktur 2D dari tiga belas ligan uji. Kemudian dibangun struktur 3D-nya dan dilakukan optimasi geometri mekanika molekul (MM) menggunakan software Chem3D Pro 12.0 untuk mendapatkan struktur senyawa uji yang paling stabil. Besar energi total hasil optimasi geometri yang diperoleh ditunjukkan pada Tabel 2.
Tabel 2. Hasil optimasi geometri ligan uji
|
Senyawa Uji |
Optimasi Geometri (kkal/mol) |
|
Trodusquemine (standar) |
266,2671 |
|
p-Nitrophenyl phosphate (substrat) |
91,7946 |
|
garciniacowone K |
169,9900 |
|
guttiferone I |
89,9485 |
|
1,7-dihydroxyxanthone |
16,2849 |
|
1-hydroxyl-7 methoxyxanthone |
23,7363 |
|
mangostinone |
58,7016 |
|
α-mangostin |
27,8396 |
|
cowanol |
35,8925 |
|
gacibiphenyl C |
0,9740 |
|
friedelin |
114,6079 |
|
β-friedelinol |
117,6584 |
|
oleanane-12-ol |
84,9465 |
Pada proses optimasi geometri, energi total molekul diminimalisasi sehingga didapatkan struktur senyawa yang paling stabil dan semirip mungkin dengan struktur senyawa yang tersedia di alam (Fitriasari dkk., 2008). Berdasarkan hasil tersebut, diperoleh struktur senyawa uji yang telah teroptimasi.
Preparasi Reseptor
Preparasi reseptor PTP1B dilakukan dengan menggunakan software BIOVIA Discovery Studio untuk memisahkan protein dari native ligand-nya agar terdapat ruang/pocket yang akan digunakan pada saat simulasi docking. Selain didapat struktur protein dengan pocket sebagai tempat protein
akan berikatan dengan ligan uji, didapat pula struktur native ligand tanpa proteinnya.
Pada proses ini juga perlu dilakukan penghapusan molekul air (H2O). Tujuan penghapusan molekul air adalah agar hanya tersisa molekul-molekul asam amino pada protein PTP1B sehingga dapat dipastikan hanya molekul asam amino tersebutlah yang nantinya akan berinteraksi dengan ligan uji. Selain itu, penghapusan molekul air juga dapat membuat interaksi protein dengan ligan uji lebih maksimal (Huey et al., 2012; Kitchen et al., 2004). Struktur 3D dari PTP1B tanpa native ligand-nya dan native ligand yang telah dipisahkan dapat dilihat pada Gambar 1.
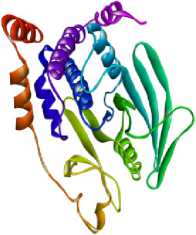
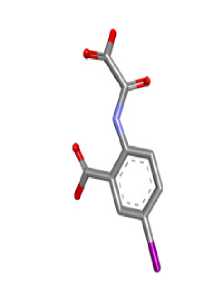
(b)
(a)
Gambar 1. Struktur 3 Dimensi Protein PTP1B tanpa ligan (a) dan native ligand PTP1B 2-(Oxalylamino)-Benzoic Acid (b)
Validasi Metode Molecular Docking
Validasi metode Molecular Docking dilakukan dengan tujuan untuk memastikan metode yang digunakan telah memenuhi syarat dan dapat digunakan untuk tahap pengujian selanjutnya. Parameter yang digunakan adalah nilai Root Mean Square Deviation (RMSD). RMSD adalah suatu parameter yang menggambarkan seberapa besar perubahan interaksi protein-ligan pada struktur kristal sebelum dan setelah dilakukannya docking. Metode penambatan dapat dikatakan valid
apabila nilai RMSD nya sebesar ≤ 2 Å (Istyastono, 2018). Nilai RMSD yang didapatkan pada validasi metode pada percobaan ini adalah 0,474 Å, sehingga dapat dikatakan metode docking yang digunakan telah valid. Data dapat dilihat pada Tabel 3. Visualisasi 2D dan 3D interaksi antara ligan alami dan enzim PTP1B hasil re-docking dapat dilihat pada Gambar 2, di mana ligan berinteraksi dengan PTP1B melalui ikatan hidrogen konvensional dan ikatan alkil .
Tabel 3. Hasil validasi metode dengan re-docking dengan 2-(Oxalylamino)-Benzoic Acid
Kode Grid Box
PDB (x, y, z)
Validasi
Energi Ikatan (kkal/mol)
RMSD Cluster (Å)
RMSD reference (Å)
1ECV
10,265
45,877
19,734
0,474
< 2 -10,07
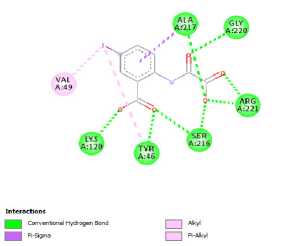
(a)
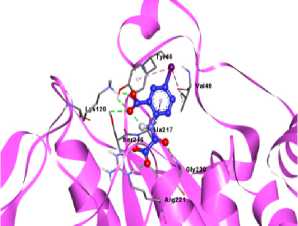
(b)
Gambar 2. Visualisasi 2D interaksi antara enzim PTP1B dengan ligan alami (a) dan Visualisasi 3D interaksi antara enzim PTP1B dengan ligan alami (b)
Docking Senyawa Uji terhadap PTP1B
Melihat hasil validasi metode docking yang telah memenuhi syarat, maka prosedur dilanjutkan dengan docking senyawa uji pada PTP1B menggunakan software yang sama yaitu AutodockTools 1.5.6. Koordinat/grid box docking senyawa uji pada PTP1B ini disesuaikan dengan grid box pada saat validasi metode karena koordinat tersebut merupakan tempat native ligand berinteraksi dengan PTP1B.
Hasil yang diperoleh dari docking senyawa uji dengan reseptor PTP1B yaitu berupa energi ikatan, konstanta inhibisi, dan ikatan hidrogen. Energi ikatan adalah hasil kalkulasi atau scoring function dari konformasi ligan (I) pada makromolekul/reseptor dalam kondisi ekuilibrium (conformational search). Dari variabel tersebut maka energi ikatan atau energi Gibbs (∆G) dapat dimuat dalam persamaan [E+I] = [EI] (Kitchen et al., 2004). Energi ikatan menunjukkan besarnya afinitas antara senyawa uji dengan reseptor PTP1B. Semakin rendah nilai energi ikatan menunjukkan ikatan antara protein dan ligan yang dihasilkan semakin stabil (Manna et al., 2017). Sementara itu, konstanta inhibisi menunjukkan kekuatan suatu senyawa dalam menghambat kerja reseptornya. Semakin kecil
nilai konstanta inhibisi, maka kekuatan hambatannya akan semakin besar (Umamaheswari et al., 2013)
Berdasarkan hasil docking (Tabel 4), tidak terdapat senyawa uji yang memiliki nilai energi ikatan maupun konstanta inhibisi yang lebih rendah dibandingkan native ligand/ligan alami. Namun, senyawa α-mangostin (senyawa uji 6) memiliki nilai energi ikatan dan konstanta inhibisi lebih rendah dibandingkan senyawa uji lainnya dengan nilai berturut-turut sebesar -8,91 kkal/mol dan 0,29317 µM. Hasil tersebut menunjukkan kemampuan senyawa α-mangostin untuk berikatan dengan sisi aktif reseptor PTP1B lebih lemah dibandingkan native ligand. Namun, senyawa ini masih memiliki kemungkinan untuk berikatan dengan sisi aktif reseptor PTP1B serta memiliki potensi hambatan yang cukup baik. Selain itu, nilai energi ikatan dan konstanta inhibisi senyawa α-mangostin menunjukkan hasil yang lebih rendah dibandingkan dengan substrat PTP1B, yaitu p-nitrophenyl phosphate dan standar berupa senyawa obat trodusquemin yang menunjukkan bahwa senyawa α-mangostin memiliki afinitas yang lebih baik dibandingkan substrat dan trodusquemin pada reseptor PTP1B.
Tabel 4. Hasil docking senyawa ekstrak Garcinia cowa terhadap reseptor PTP1B
|
No. |
Senyawa |
Energi Ikatan (kkal/mol) |
KI (µM) |
Interaksi dengan asam amino | |
|
Ikatan hidrogen |
Ikatan lain | ||||
|
1 |
Ligan Alami (2-(Oxalylamino)-Benzoic Acid) |
-10,07 |
0,0417 |
ALA217, GLY 220, ARG221, SER216, TYR46, LYS120 |
VAL49**, ALA217*, TYR46** |
|
2 |
Standar (Trodusquemine) Substrat |
-5,02 |
209,92 |
LYS120 |
LYS11**6, TYR46^, PHE18**2, ASP48^^ |
|
3 |
(p-nitrophenyl phospate) |
-8,14 |
1,08 |
ARG221, SER216, ALA217 |
CYS215**, ILE219**, LYS120^^^, TYR46^^^ |
|
4 |
Senyawa Uji 1 (garciniacowone K) Senyawa Uji 2 |
-8,01 |
1,34 |
ASP48 |
VAL49**, ALA217**, ILE219**, PHE182**, TYR46** |
|
5 |
(guttiferone I) Senyawa Uji 3 |
-8,12 |
1,11 |
LYS120, CYS215, GLY220, ASP181, SER216, ARG221 |
ARG47**, PHE182**, LYS116**, ALA217*, TYR46• |
|
6 |
(1,7-dihydroxyxanthone) Senyawa Uji 4 |
-7,64 |
2,53 |
SER126, ARG221, LYS120 |
CYS215**, ALA217*, ALA217**, PHE182••, TYR46•• |
|
7 |
(1-hydroxyl-7 methoxyxanthone) Senyawa Uji 5 |
-7,26 |
4,79 |
ARG221, SER216, ASP216 |
CYS215^^, ILE219**, ALA217*, ALA217**, VAL49**, TYR46** |
|
8 |
(mangostinone) Senyawa Uji 6 |
-7,65 |
81,87 |
LYS120, ASP181, SER216, ARG221 |
VAL49**, TYR46••, PHE182••, ALA217*, CYS215^ |
|
9 |
(α-mangostin) Senyawa Uji 7 |
-8,91 |
0,29317 |
GLN262, SER216, ASP181, LYS120 |
CYS215^, PHE182••, ARG221°, ALA217* |
|
10 |
(Cowanol) Senyawa Uji 8 |
-8,27 |
0,86933 |
GLN262, LYS120 |
LYS116**, PHE182**, TYR46•• |
|
11 |
(gacibiphenyl C) |
-7,54 |
2,96 |
LYS120, TYR46, ASP181, SER216 |
PHE182**, ALA217**, VAL49** |
|
13 |
Senyawa Uji 10 (β-friedelinol) |
-5,58 |
0,08187 - |
PHE182*, TYR46**, ARG47** | |
|
Senyawa Uji 11 | |||||
|
14 |
(oleanane-12-ol) |
-5,4 |
86,41 |
GLN262 |
PHE182**, ARG47** TYR46** |
Keterangan:
|
1. 2. 3. 4. |
Ikatan pi-sigma: * 5. Ikatan pi-anion/attractive charge: ^^^ Ikatan alkil/pi-alkil: ** 6. Pi-lone pair: • Ikatan pi-sulfur: ^ 7. Pi-pi stacked/T-shaped: •• Ikatan karbon hidrogen/pi-donor 8. Unfavorable donor-donor: ° hidrogen: ^^ |
Senyawa Uji 9
12 (Friedelin)
-6,08 34,73 ASP48, ARG47 PHE182*, PHE182**
Pada Tabel 4 juga dapat dilihat bahwa senyawa α-mangostin berinteraksi dengan PTP1B melalui ikatan hidrogen diantaranya pada asam amino SER216 dan LYS120. Ikatan hidrogen tersebut sama dengan interaksi yang terjadi antara native ligand dengan PTP1B. Hal tersebut menandakan kantung/sisi aktif tempat native ligand dan senyawa α-mangostin berikatan dengan PTP1B juga sudah sama dan diharapkan akan memberikan afinitas yang sama dalam menghambat kerja PTP1B. Interaksi α-mangostin dengan PTP1B dapat dilihat pada Gambar 3.
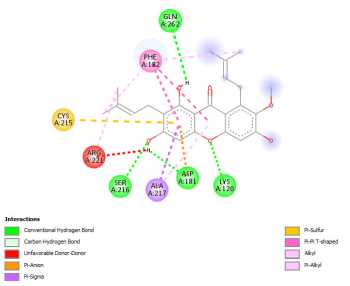
(a)
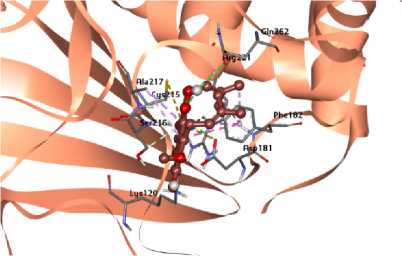
(b)
Gambar 3. Visualisasi 2D interaksi antara enzim PTP1B dengan α-mangostin (a) dan
Visualisasi 3D interaksi antara enzim PTP1B dengan α-mangostin (b)
SIMPULAN
Senyawa α-mangostin memiliki potensi sebagai antidiabetes melalui ikatan pada reseptor PTP1B karena memiliki afinitas dengan reseptor PTP1B tersebut dengan nilai ikatan sebesar -8,91 kkal/mol, memiliki aktivitas hambatan yang cukup baik dengan nilai konstanta inhibisi sebesar 0,29317 µM
serta berinteraksi melalui ikatan hidrogen diantaranya pada asam amino SER216 dan LYS120. Disamping itu, hasil uji prediksi farmakokinetik dan toksisitas α-mangostin menunjukkan bahwa senyawa tersebut memiliki profil farmakokinetika yang baik yaitu HIA (91,810986%), Sel Caco2 (20,694 nm/detik), dan PPB (96,619656%). Pada pengujian toksisitasnya, α-mangostin
menunjukkan hasil non-mutagenik dan negatif karsinogenik pada tikus. Namun, memiliki sifat karsinogenik pada mencit sehingga dapat dijadikan bahan pertimbangan pada pengujian lebih lanjut secara in-vivo. Dari hasil tersebut, menunjukkan bahwa α-mangostin memiliki potensi sebagai antidiabetes mellitus tipe 2 dengan menghambat PTP1B.
DAFTAR PUSTAKA
Ali, S., Mouton, C. D., Jabeen, S., Zeng, Q., Galloway, G., dan Mendelson, J. 2013. Suicide, depression, and CYP2D6: How are they linked? Current Psychiatry. 12(5).
American Diabetes Association. 2009. Multiple Doses of Trodusquemine Improve Glucose Tolerance in Type 2 Diabetic Subjects. Tersedia secara online
https://professional.diabetes.org/abstra ct/multiple-doses-trodusquemine-improve-glucose-tolerance-type-2-diabetic-subjects [Diakses pada 28 Maret 2021].
Bertilsson, L., Dahl, M.-L., Dal´en, P., dan Al-Shurbaji, A. 2002. Molecular genetics of CYP2D6: clinical relevance with focus on psychotropic drugs. Br. J. Clin. Pharmacol. 53: 111–122.
Cheng, J., Palva, A.M., de Vos, W.M., dan Satokari, R. 2013. Contribution of the intestinal microbiota to human health: from birth to 100 years of age. Curr Top Microbiol Immunol. 358: 323-46
Dai, D., Tang, J., Rose, R., Hodgson, E., Bienstock, R.J., Mohrenweiser, H.W., dan Goldstein, J.A. 2001. Identification of variants of CYP3A4 and characterization of their abilities to metabolize testosterone and chlorpyrifos. J. Pharmacol. Exp. Ther. 299: 825–831.
Fatimah, R.N. 2015. Diabetes Melitus Tipe 2. J Majority. 4(5): 93-99.
Fitriasari, A., Wijayanti, N. K., Ismiyati, N., Dewi, D., Kundarto, W., A. Sudarmanto, B. S., dan Meiyanto, E. 2008. Studi Potensi Kurkumin dan Analognya sebagai Selective Estrogen Receptor Modulator (SERMs): Docking pada Reseptor Estrogen ß. Pharmacon. 9(1).
Foti, R.S dan Wahlstrom, J.L. 2008. CYP2C19 inhibition: the impact of substrate probe selection on in vitro inhibition profiles. Drug Metab. Dispos. 36: 523–528.
Heyne, K. 1987. Tumbuhan Berguna Indonesia Jilid III. Jakarta: Yayasan Sarana Wana Jaya.
Huey, R., Morris, G. M., dan Forli, S. 2012. Using AutoDock 4 and AutoDock Vina with AutoDockTools: A Tutorial, California: The
Scripps Research Institute.
Istyastono, E. P. 2018. Rancangan Obat dan Penapisan Virtual Berbasis Struktur. Yogyakarta: Sanata Dharma
University Press.
Jain, A. J., dan Nicholls, A. 2008. Recommendations for evaluational methods. J. Comput.
Aided Mol. 22: 133-139.
Kitchen, D. B., Decornez, H., Furr, J. R., dan Bajorath, J. 2004. Docking
and Scoring in Virtual Screening in Drug Discovery, Methods and
Applications. Nat. Rev. Drug Discov. 3: 935-949
Kumar, R., Giri, A., Nadendla, R.R. 2018. In Silico ADME Profiling of CDK9 Inhibitors. Journal of Scientific Research in Pharmacy. 7(3): 30-4.
Ma, XI., Chen, C., dan Yang, J. 2005. Predictive model of blood-brain barrier penetration of organic compounds. Acta Pharmacologica Sinica. 26: 500–512.
Manna, A., Laksitorini, M. D., Hudiyanti, D., dan Siahaan, P. 2017. Molecular Docking of Interaction between E-Cadherin Protein and Conformational Structure of Cyclic Peptide ADTC3 (Ac-CADTPC-NH2) Simulated on 20 ns. Journal of Scientific and Applied Chemistry. 20(1): 30-36.
Nursamsiar, N., Toding, A. T., dan Awaluddin, A. 2016. Studi In Silico Senyawa Turunan Analog Kalkon dan Pirimidin Sebagai Antiinflamasi: Prediksi Absorpsi, Distribusi, dan Toksisitas. PHARMACY. 13(1): 92-100.
Olokoba, A.B, Obateru, O.A, dan Olokoba, L.B. 2012. Type 2 Diabetes Mellitus: A Review. of Current Trends. Oman Medical Journal. 27(4).
Oyesakin Y.M., George D.E., Fadare R.Y., Idris A.Y., dan Fadare O.A. 2018. Molecular Docking and In-Silico ADME Prediction of Substituted (E)-4-Styryl-7,8-dihydroquinazolin-5(6H)-ones and 5-((E)-Styryl)pyrimidine[4,5-d]pyrimidine-2,4(1H,3H)-diones as Potential SERT Inhibitors and Antidepressants. American Journal of Pharmacological Sciences. 6(1): 2532.
Phukatmuen, P., Raksat, A., Lapookhieo, S., Charoensup, R., Duangyod, T., dan Maneerat, W. 2020. Bioassay-guided isolation and identification of antidiabetic compounds from Garcinia cowa leaf extract. Heliyon. 6(4).
Prasetiawati, R., Suherman, M., Permana, B., dan Rahmawati. 2021. Molecular Docking Study of Anthocyanidin Compounds Against Epidermal Growth Factor Receptor (EGFR) as Anti-Lung Cancer. Indonesian Journal of Pharmaceutical Science and Technology. 8(1): 8-20.
Purwaniati. 2020. Studi Docking Molekul Aktivitas Obat COVID-19 dari Derivat N-(2-phenylethyl)methanesulfonamide sebagai Inhibitor Main Protease. Ad-Dawaa’ J. Pharm. Sci. 3(1): 1 – 11.
Ratwita, W., Sukandar E. Y., Asnyana, K., dan Kurniawati, N. F. Alpha Mangostin and Xanthone Activity on Fasting Blood Glucose, Insulin and Langerhans Islet of Langerhans in Alloxan Induced Diabetic Mice. Pharmacognosy Journal. Vol. 11(1): 64-68.
Sun, J., Qu, C., Wang, Y., Huang, H., Zhang, M., dan Zou, W. 2016. Type 2 Diabetes Mellitus and ProteinTyrosine Phosphatase 1b. Journal of Diabetes, Metabolic Disorders & Control. 3(8): 180-183.
Umamaheswari, M., Madeswaran, A., and Asokkumar, K., 2013. Virtual
Screening Analysis and In-vitro Xanthine Oxidase Inhibitory Activity of Some Commercially Available Flavonoids. Iranian Journal of Pharmaceutical Research: IJPR. 12(3): 317–323.
Van Booven, D., Marsh, S., McLeod, H., Carrillo, M.W., Sangkuhl, K., Klein, T.E., dan Altman, R.B. 2010. Cytochrome P450 2C9-CYP2C9.
Pharmacogenet. Genomics. 20: 277– 281.
83
Discussion and feedback