MOLECULAR DOCKING LIKOPEN SEBAGAI ANTIOSTEOPOROSIS SECARA IN SILICO
on
p-ISSN 1907-9850 e-ISSN 2599-2740
MOLECULAR DOCKING LIKOPEN SEBAGAI ANTIOSTEOPOROSIS SECARA IN SILICO
N. M. P. Susanti1*, D. P. D. Saputra1, P. L. Hendrayati1, I. P. D. N. Parahyangan1, G. A. K.
Amarawati1
1Program Studi Farmasi, Fakultas Matematika dan Ilmu Pengetahuan Alam, Universitas Udayana
*Email: dekpitsusanti@unud.ac.id
ABSTRAK
Osteoporosis merupakan penyakit degeneratif akibat pengurangan massa atau jaringan tulang sehingga tulang menjadi rapuh. Salah satu penyebab semakin rapuhnya tulang adalah terbentuknya radikal bebas akibat stress oksidatif oleh reactive oxygen species (ROS). ROS dapat merangsang terjadinya resorpsi tulang. Likopen merupakan senyawa yang terdapat dalam buah tomat yang ditemukan dalam jumlah besar. Likopen diketahui memiliki aktivitas antioksidan kuat. Tujuan dari penelitian ini adalah untuk mengetahui aktivitas antiosteopotosis likopen dari buah tomat melalui mekanisme antioksidan menggunakan metode molecular docking secara in silico. Tahapan penelitian yang dilakukan adalah penyiapan database struktur 3D likopen serta protein Super oxide Dismutase (SOD) dan Gluthation Peroxidase (GPx), preparasi protein menggunakan aplikasi Chimera 1.10.1, optimasi struktur 3D likopen menggunakan aplikasi HyperChem 8, serta validasi metode molecular docking dan docking likopen pada protein-protein tersebut menggunakan aplikasi Autodock 4.2. Hasil penelitian menunjukkan bahwa likopen mampu berinteraksi dengan protein SOD dan GPx yang ditunjukkan oleh energi ikatan yang bernilai negatif, yaitu berturut-turut -0,75 dan -1,61 kkal/mol. Interaksi tersebut menunjukkan bahwa likopen mampu menetralkan radikal bebas dengan cara menginduksi protein SOD dan GPx sehingga stress oksidatif pemicu terjadinya resorpsi tulang yang merupakan salah satu penyebab terjadinya osteoporosis dapat dicegah.
Kata Kunci : Antiosteoporosis, In Silico, Likopen, Molecular Docking
ABSTRACT
Osteoporosis is a degenerative disease due to the reduction of mass or bone tissue so that the bones become brittle. One of the causes of the fragility of bone is the formation of free radicals due to oxidative stress by reactive oxygen species (ROS). ROS can stimulate bone resorption. Lycopene is a compound contained in tomatoes found in large quantities. Lycopene is known to have strong antioxidant activity. The purpose of this research is to know the activity of lycopene as antiosteopotosis from tomato fruit through antioxidant mechanism using molecular docking method in silico. The stages of this research are preparation of 3D lycopene structure database and SOD and GPx protein, protein preparation using Chimera 1.10.1 application, optimization of lycopene 3D structure using HyperChem 8 application, and validation of molecular docking and lycopene docking methods on the proteins using Autodock application 4.2. The results showed that lycopene was able to interact with SOD and GPx proteins shown by negative bond energy, ie -0.75 and -1,61 kcal mol respectively. These interactions show that lycopene can neutralize free radicals by inducing SOD and GPx proteins so that oxidative stress triggering the bone resorption which as one of the causes of osteoporosis can be prevented.
Keywords : Antiosteoporosis, In Silico, Lycopene, Molecular Docking
PENDAHULUAN
Osteoporosis merupakan kondisi pengurangan substansi tulang dibandingkan dengan keadaan normal sehingga tulang menjadi rapuh (Hurwitz, 2016; Ramadani, 2010). Kondisi ini lebih besar dialami oleh wanita terutama ketika pasca menopause. Di Indonesia, 8,5 juta dari 222 juta penduduk Indonesia mengalami osteoporosis dan seiring
dengan peningkatan jumlah penduduk, maka diperkirakan jumlah penderita ostoeporosis meningkat hingga 11,5 juta pada tahun 2050 (Nuhonni, 2000).
Pengobatan osteoporosis saat ini banyak mengacu pada peningkatan kepadatan tulang (Junaidi, 2009). Selain itu, beberapa obat seperti golongan bifosfonat, raloxifen, kalsitonin, dan tibolone sering digunakan untuk membangun tulang, memadatkan tulang,
serta menghambat proses pengeroposan (Tandra, 2009). Namun, semakin rapuh dan tipisnya tulang juga disebabkan oleh produksi hormon, proses penuaan, serta terbentuknya radikal bebas akibat stress oksidatif oleh Reactive Oxygen Species (ROS) yang mampu merangsang resorpsi tulang (Monroe et al., 2003; Yoon et al., 2016). Stress oksidatif
merupakan hasil dari kelebihan produksi ROS dalam tubuh. ROS mengandung satu atau lebih elektron yang tidak berpasangan sehingga sangat reaktif untuk menstabilkan keseimbangan elektronnya. Produksi ROS meningkat seiring dengan pertambahan usia serta berhubungan dengan beberapa penyakit kronis termasuk osteoporosis.
Buah tomat (Solanum lycopersicum L.) merupakan salah satu tanaman yang banyak tumbuh di Indonesia. Salah satu kandungan fitokimia dalam buah tomat adalah likopen. Buah tomat mensintesis likopen dalam jumlah banyak selama pemasakan, yaitu mencapai 90% dari fraksi karotenoid total (Salunkhe et al., 1991 dalam Novita dkk., 2015). Beberapa penelitian menunjukkan potensi likopen dalam bidang kesehatan. Salah satu penelitian menunjukkan adanya korelasi langsung antara likopen serum dengan penurunan risiko osteoporosis antara perempuan pasca menopause (Rao et al., 2007).
Untuk mengetahui aktivitas likopen sebagai antiosteoporosis, maka perlu dilakukan uji pendahuluan menggunakan metode molecular docking secara in silico. Teknik molecular docking secara in silico dapat digunakan untuk memprediksi proses interaksi suatu protein dengan suatu ligan, sehingga mekanisme molekularnya dapat diketahui. Metode ini dapat meningkatkan efektivitas dan efisiensi dalam penelitian untuk menemukan obat baru. Oleh karena itu, penelitian ini sangat penting dilakukan untuk mengetahui mekanisme likopen dalam mencegah osteoporosis melalui penetralan radikal bebas dengan induksi perubahan oksigen reaktif menjadi molekul air oleh antioksidan endogen (protein SOD dan GPx) secara in silico.
MATERI DAN METODE
Bahan
Struktur 2D likopen diunduh dari https://pubchem.ncbi.nlm.nih.gov/compound/4 46925 yang diubah ke dalam bentuk 3D dan
dioptimasi menggunakan aplikasi HyperChem 8, serta struktur 3D protein SOD (PDB ID: 1MFM) dan GPx (PDB ID: 2F8A) diunduh dari http://www.rcsb.org/pdb/home.do.
Peralatan
Seperangkat komputer dengan spesifikasi Windows 8 64 bit dan program Autodock 4.2, HyperChem 8, Chimera 1.10.1, dan Open Babel.
Cara Kerja
Preparasi Protein
Preparasi protein SOD dan GPx dilakukan dengan memisahkan protein dengan native ligand-nya menggunakan aplikasi Chimera 1.10.1.
Optimasi Struktur 3D Likopen
Struktur 2D likopen yang telah diunduh diubah ke dalam bentuk 3D dan dioptimasi menggunakan aplikasi HyperChem 8. Digunakan metode komputasi semi empirik AM1 dengan kalkulasi single point dan optimasi geometri.
Validasi Metode Molecular Docking
Validasi metode molecular docking dilakukan dengan men-docking-kan kembali native ligand pada protein yang telah dihilangkan native ligand-nya menggunakan aplikasi Autodock 4.2 dengan parameter RMSD. Semakin kecil nilai RMSD yang diperoleh (mendekati 0) maka pose native ligand setelah redocking semakin mendekati pose native ligand sebelum dipisahkan dengan proteinnya.
Docking Sianidin dan Peonidin pada Protein SOD dan GPx
Senyawa likopen hasil optimasi didocking-kan pada protein SOD dan GPx yang telah dihilangkan native ligand-nya menggunakan aplikasi Autodock 4.2. Hasil analisis menunjukkan konformasi energi ikatan terendah untuk berikatan dengan protein target.
Analisis Data
Hasil docking molecular adalah energi ikatan. Nilai energi ikatan menunjukkan kekuatan ikatan antara senyawa dan reseptor. Semakin rendah harga energi ikatan, maka ikatannya semakin kuat dan stabil.
HASIL DAN PEMBAHASAN
Preparasi Protein SOD dan GPx
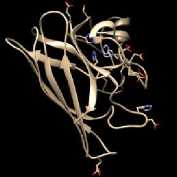
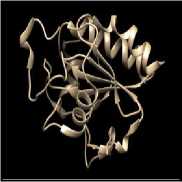
Preparasi protein dilakukan dengan memisahkan native ligand dari struktur protein menggunakan program Chimera 1.10.1 sehingga diperoleh struktur protein SOD dan GPx tanpa native ligand dan tersedia ruang (pocket/cavity) yang nantinya digunakan dalam proses docking. Selain diperoleh struktur protein tanpa native ligand, diperoleh pula struktur native ligand yang terpisah dari protein. Struktur protein tanpa native ligand dan struktur native ligand yang telah terpisah dari proteinnya dapat dilihat pada gambar 1 dan 2.
(a)

(b)
Gambar 1. Struktur Protein SOD (a) dan native
ligand-nya (b)
(a)
(b)
Gambar 2. Struktur Protein GPx (a) dan native ligand-nya (b)
Optimasi Struktur 3D Likopen
Struktur 2D likopen yang telah diunduh diubah ke dalam bentuk 3D dan dioptimasi menggunakan aplikasi HyperChem 8 dengan
metode semi empirik AM1 serta kalkulasi single point dan optimasi geometri untuk memperoleh struktur 3D likopen yang paling stabil dengan nilai energi struktur terendah. Besar energi saat dilakukan kalkulasi single point adalah -11310,758 kkal/mol, kemudian dilanjutkan dengan optimasi geometri dan terjadi penurunan energi struktur menjadi -11359,031 kkal/mol. Saat dilakukan optimasi geometri, energi struktur dapat diminimalisir untuk memperoleh struktur yang paling stabil. Struktur 3D likopen hasil optimasi geometri ditunjukkan oleh gambar 3.
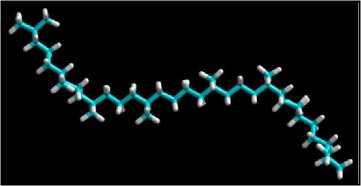
Gambar 3. Hasil Optimasi Struktur 3D Senyawa Likopen
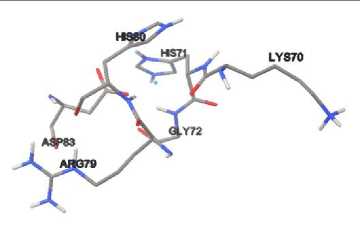
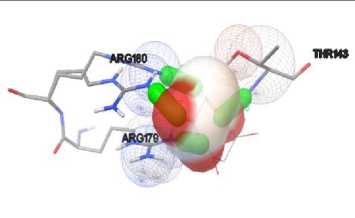
Validasi Metode
Validasi metode molecular docking dilakukan dengan men-docking-kan kembali native ligand pada protein SOD dan GPx menggunakan program Autodock 4.2. Pada tahap ini, dilakukan penyesuaian koordinat tempat interaksi pada aplikasi Autodock 4.2 dengan pengaturan grid center dan grid size. Parameter validasi metode yang digunakan adalah nilai RMSD. RMSD merupakan pengukuran dua pose dengan membandingkan posisi atom antara struktur eksperimental dengan struktur yang di-docking-kan pada protein (Lestari, 2015). Semakin kecil nilai RMSD yang diperoleh maka pose ligand yang diprediksi semakin baik karena semakin mendekati konformasi native ligand,
sedangkan semakin besar nilai RMSD maka semakin besar kesalahan prediksi interaksi antara ligand dengan protein (Agistia dkk., 2013). Nilai RMSD yang diperoleh pada penelitian ini adalah sebesar 3,54 Å untuk protein SOD dengan native ligand-nya dan 2,12 untuk protein GPx dengan native ligand-nya. Berdasarkan hasil yang didapat, maka metode molecular docking yang digunakan pada penelitian ini dikatakan valid dan dapat digunakan untuk docking senyawa likopen pada protein SOD dan GPx. Visualisasi interaksi yang terjadi antara protein SOD dan
GPx dengan native ligand-nya ditunjukkan pada gambar 4.
(a)
(b)
Gambar 4. Visualisasi interaksi antara Protein SOD dengan Native Ligand-nya (a) dan GPx dengan Native Ligand-nya (b)
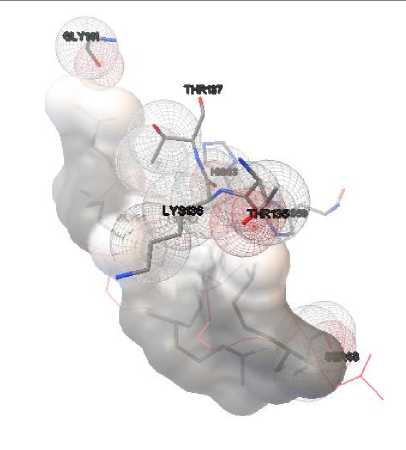
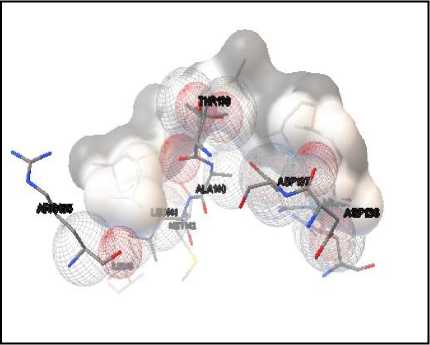
Docking Likopen pada Protein SOD dan GPx
Senyawa likopen teroptimasi didocking-kan pada protein SOD dan GPx menggunakan program Autodock 4.2 pada koordinat yang sama dengan koordinat yang digunakan saat melakukan validasi metode. Proses docking menghasilkan sepuluh konformasi ikatan likopen dengan protein SOD dan GPx. Dari sepuluh konformasi tersebut dipilih satu konformasi dengan nilai energi ikatan terendah. Energi ikatan menunjukkan afinitas likopen pada protein. Semakin kecil nilai energi ikatan yang diperoleh maka semakin stabil ikatan yang terjadi antara likopen dengan protein SOD dan GPx. Konfomasi struktur hasil docking yang diharapkan adalah konformasi dengan nilai energi ikatan terendah yang berada pada sisi aktif protein atau koordinat tempat ikatan yang sama dengan native ligand yang telah diatur sebelumnya.
Nilai energi ikatan terendah yang diperoleh antara likopen dengan protein SOD dan GPx berturut-turut -0,75 dan -1,61 kkal/mol. Hal ini menunjukkan bahwa likopen mampu berinteraksi pada sisi aktif kedua 32
protein tersebut. Maka dari itu, berdasarkan hasil penelitian ini dapat diprediksi bahwa likopen memiliki aktivitas sebagai antiosteoporosis karena memiliki afinitas dengan protein SOD dan GPx. Interaksi antara likopen dengan kedua protein tersebut mampu menginduksi penetralan radikal bebas sehingga resorpsi tulang dapat dicegah. Visualisasi interaksi antara likopen dengan protein SOD dan GPX ditunjukkan oleh gambar 5.
(a)
(b)
Gambar 5. Visualisasi Interaksi Likopen dengan Protein SOD (a) dan GPx (b).
SIMPULAN DAN SARAN
Simpulan
Likopen memiliki potensi sebagai antiosteoporosis secara molekular karena
memiliki afinitas dengan protein SOD dan GPx dengan nilai energi ikatan berturut-turut -0,75 dan -1,61 kkal/mol, sehingga dapat menginduksi protein SOD dan GPx dalam menetralkan radikal bebas.
Saran
Perlu dilakukan uji secara in vitro dan in vitro untuk melihat kemampuan serta efektifitas dari likopen sebagai
antiosteoporosis.
UCAPAN TERIMA KASIH
Penulis mengucapkan terimakasih kepada dosen pembimbing, keluarga, teman-teman dan semua pihak yang telah sangat membantu dalam penelitian ini.
DAFTAR PUSTAKA
Hurwitz, S., 2016, Osteoporosis and Fracture, Journal of Osteoporosis and Physical Activity, 4 (4): 1-2
Junaidi, L., 2009, Osteoporosis: Pengenalan, Pencegahan serta Pengobatan Penyakit Osteoporosis dan Penyakit Tulang Lain yang Mirip, PT Bhuana Ilmu Populer, Jakarta
Monroe, D. G., F. J. Secreto, T. C. Spelsberg, 2003, Overview of Estrogen Acton in Osteoblasts: Role of The Ligand The Receptor and The Co-Regulation,
Journal Musculosceletal Neuron
Novita, M., Satriana, dan E. Hsmarita, 2015, Kandungan Likopen dan Karotenoid Buah Tomat (Lycopercium pyriforme) pada Berbagai Tingkat Kematangan: Pengaruh Pelapisan dengan Kitosan dan Penyimpanan, Jurnal Teknologi Industri Pertanian Indonesia, 7 (1): 35-39
Nuhonni, S. A., 2000, Osteoporosis dan
Pencegahannya, Fakultas Kedokteran Universitas Indonesia, Jakarta
Ramadani, M., 2010, Faktor-Faktor Resiko
Osteoporosis dan Upaya
Pencegahannya, Jurnal Kesehatan Masyarakat, 4 (2): 111-115
Rao, L. G., E. S. Mackinnon, R. G. Josse, T. M. Murray, A. Strauss, and A. V. Rao, 2007, Lycopene Consumption Decrease Oxidative Stress and Bone Resorptionmarkers in Postmenopausal Women, Osteoporosis International, 18 (1): 109-115
Tandra, H., 2009, Sesuatu yang Harus Anda Ketahui Tentang Osteoporosis: Mengenal, Mengatasi, dan Mencegah Tulang Keropos, PT Gramedia Pustaka Utama, Jakarta
Yoon, J., D. Kim, E. Kim, B. Park, J. Yoon, H. Kim, and J. Park, 2016, Protective Effects of Remifentanil Agains H2O2-Induced Oxidative Stress in Human Osteoblas, Journal of Dental Anesthesia and Pain Medicine, 16 (4): 263-271
33
Discussion and feedback