ANALISIS PRIMER UNTUK AMPLIFIKASI PROMOTER inhA MULTIDRUG RESISTANCE TUBERCULOSIS (MDR-TB) DENGAN METODE POLYMERASE CHAIN REACTION (PCR)
on
ISSN 1907-9850
ANALISIS PRIMER UNTUK AMPLIFIKASI PROMOTER inhA MULTIDRUG RESISTANCE TUBERCULOSIS (MDR-TB) DENGAN METODE POLYMERASE CHAIN REACTION (PCR)
I Gusti Ayu Agung Septiari 1*, Putu Sanna Yustiantara 1,2, dan Sagung Chandra Yowani 1,2
1Jurusan Farmasi FMIPA Universitas Udayana, Bukit Jimbaran, Bali
2Kelompok Studi MDR-TB & XDR-TB, FMIPA, Universitas Udayana, Bukit Jimbaran, Bali *Email : gung.ari71@gmail.com
ABSTRAK
Penelitian ini bertujuan untuk menganalisis beberapa pasangan primer dalam mengamplifikasi daerah promoter inhA secara in silico dan in vitro. Analisis primer secara in silico dilakukan menggunakan program Clone Manger Suite 6. Sekuen DNA templat yang digunakan dalam analisis primer diunduh dari situs www.ncbi.nlm.nih.gov dengan kode genbank : U66801.1 yang merupakan sekuens DNA gen fabG M. tuberculosis H37Rv. Deteksi primer secara in vitro dilakukan dengan teknik PCR menggunakan isolat P16 dan 86 M. tuberculosis MDR sebagai templat. Proses amplifikasi dilakukan dengan kondisi sebagai berikut: denaturasi awal pada suhu 95°C selama 15 menit , amplifikasi sebanyak 45 siklus (denaturasi pada suhu 94°C selama 1 menit, annealing pada suhu 55°C selama 1 menit 20 detik dan ekstensi pada suhu 72°C selama 1 menit 10 detik) serta ekstensi akhir pada suhu 72°C selama 10 menit. Selanjutnya, deteksi produk PCR dilakukan dengan gel elektroforesis 1,5%. Kesimpulan yang diperoleh dari penelitian ini adalah, diperoleh pasangan primer forward (mabA-inhA-promoter-FS) dengan urutan sekuen 5’-ACATACCTGCTGCGCAAT-3’ (18 nukleotida) dan primer reverse (mabA-inhA-promoter-R) dengan urutan sekuens 5’-CTCCGGTAACCAGGACTGAA-3’ (20 nukleotida) (Chen et al., 2011) yang memenuhi kriteria pasangan primer yang baik dilihat dari panjang primer, nilai Tm, %GC, stabilitas, jumlah hairpins, dimers dan runs. Dari deteksi secara in vitro sepasang primer tersebut juga telah mampu mengamplifikasi daerah promoter inhA sebesar 284 pb.
Kata kunci : Analisis primer, PCR, promoter inhA, M. tuberculosis, in silico, in vitro, Clone Manager Suite 6
ABSTRACT
The aim of this study was to analyze several primary combinations for amplifying inhA promoter region by in silico and in vitro ways. Primary in silico’s analysis was done by Clone Manager Suite 6 program. fabG gene sequence of M. tuberculosis was downloaded from www.ncbi.nlm.nih.gov (genbank: U66801.1) and used as DNA template. In vitro detection was done by PCR technique using P16 and 86 M. tuberculosis MDR isolates as DNA template. Amplification was done in described conditions: predenaturation at 95°C for 15 minutes, 45 cycle of amplication (denaturation on 94°C for 1 minute, annealing on 54°C for 1 minute 20 seconds dan extension on 72°C for 1 minute 10 seconds) and also post extension on 72°C for 10 minutes. PCR product was detected by agarose gel elektroforesis (1,5%). In conclusion, combination of primary forward (mabA-inhA-promoter-FS) 5’-ACATACCTGCTGCGCAAT-3’ (18 nucleotide) and primary reverse (mabA-inhA-promoter-R) 5’-CTCCGGTAACCAGGACTGAA-3’ (20 nucleotide) (Chen et al., 2011) have met the good criteria of primary combination which was seen from several aspects such as: primary length, Tm value, %GC, stability, number of hairpins, dimers and runs. In vitro detection showed that the primary combination also amplified inhA promoter region with the length of 284 pb.
Keywords : Primer analysis, PCR, inhA promoter region, M. tuberculosis, in silico, in vitro, Clone Manager Suite 6
PENDAHULUAN
Salah satu masalah kesehatan utama yang masih dihadapi dunia adalah penyakit tuberkulosis (TB). TB merupakan penyakit infeksi dengan tingkat mortalitas yang tinggi. Berdasarkan laporan World Health Organization (WHO), pada tahun 2011 terjadi 1,4 juta kematian karena TB di seluruh dunia (WHO, 2012).
Usaha dalam menurunkan angka kejadian TB menghadapi tantangan baru dengan munculnya fenomena Multi Drug Resistance-Tuberculosis (MDR-TB). MDR-TB merupakan TB yang disebabkan oleh bakteri yang resisten terhadap setidaknya isoniazid (INH) dan rifampisin (RIF) (WHO, 2012). Isoniazid (INH) merupakan OAT lini pertama yang termasuk dalam regimen standar pengobatan TB (Dipiro et al., 2008). Terdapat beberapa penelitian sebelumnya yang telah mendeteksi adanya mutasi pada daerah promoter inhA penyebab resistensi terhadap INH dengan teknik PCR.
Salah satu komponen yang berperan penting dalam proses PCR adalah primer (Innis dan Gelfand, 1990). Primer akan membatasi fragmen DNA target yang akan diamplifikasi (Handoyo dan Ari, 2001). Sebelum digunakan dalam proses amplifikasi, perlu diperhatikan beberapa hal sehingga suatu primer dapat menempel secara efisien dan spesifik pada fragmen DNA target yang nantinya akan berpengaruh terhadap keberhasilan proses PCR. Pertimbangan tersebut meliputi, panjang primer, %GC, Tm, dimer pada ujung 3’, stabilitas, jumlah runs, repeats, hairpins dan false priming (Bartlett dan Stirling, 2003; Borah, 2011). Perkembangan ilmu bioinformatika memungkinkan dilakukannya desain dan analisis primer. Penggunaan software merupakan salah satu hal yang identik dengan bioinformatika. Penggunaan software hanya merupakan suatu proses analisis secara in silico yang perlu diikuti dengan deteksi secara in vitro untuk mengukuhkan kemampuan suatu primer dalam menghasilkan produk melalui proses PCR dan visualisasi dengan gel elektroforesis. Penelitian ini menjadi bagian dari proses untuk mendeteksi adanya mutasi pada daerah promoter inhA. Tujuan khusus dari penelitian ini adalah untuk menganalisis kombinasi pasangan primer
yang digunakan dalam proses PCR untuk mengamplifikasi daerah promoter inhA.
MATERI DAN METODE
Gen fabG (mabA) M. tuberculosis H37Rv
Gen mabA (fabG) M. tuberculosis H37Rv digunakan sebagai templat untuk menganalisa pasangan primer (kode genbank U66801.1) yang dapat diunduh pada situs www.ncbi.nlm.nih.gov.
Primer
Terdapat beberapa pasangan primer yang diperoleh dari penelitian-penelitian sebelumnya yang selanjutnya akan dianalisis secara in silico menggunakan program Clone Manager Suite 6 (University of Groningen).
Software dalam Analisis Primer
Pada penelitian ini dilakukan analisis primer secara in silico dengan menggunakan software Clone Manager Suite 6 (University of Groningen).
Cara Kerja
Prosedur Kerja Software Clone Manager Suite 6 (University of Groningen)
Untuk melakukan analisis pasangan primer, langkah-langkah yang dilakukan adalah mengunduh sekuens gen mabA (fabG) pada situs www.ncbi.nlm.nih.gov kemudian sekuens tersebut di-copy. Langkah selanjutnya adalah memasukkan sekuens gen mabA (fabG) dengan cara membuka program Clone Manager Suite 6. Pada menu bar, dipilih menu File klik New kemudian akan muncul kotak Create New Molecule Wizard, ditandai bagian Paste Sequence. Kemudian pada menu bar dipilih menu Primer, diklik submenu Direct Entry masukkan urutan nukleotida primer forward dan primer reverse. Setelah memasukkan urutan sekuens masing-masing primer selanjutnya diklik toolbar Link to Molecule untuk mengetahui daerah penempelan primer pada sekuens gen mabA (fabG). Untuk memperoleh hasil analisis pasangan primer tersebut, pilih kembali menu Primer, diklik submenu Analyze dan Analyze Mix Wizard.
Amplifikasi Daerah Promoter inhA dengan Teknik PCR
DNA templat yang digunakan untuk amplifikasi berasal dari DNA kromosomal yang diisolasi dari isolat P16 dan 86 M. tuberculosis Multi Drug Resistance. Kondisi PCR yang diaplikasikan adalah, denaturasi awal pada suhu 95°C selama 15 menit , amplifikasi sebanyak 45 siklus (denaturasi pada suhu 94°C selama 1 menit, annealing pada suhu 55°C selama 1 menit 20 detik dan ekstensi pada suhu 72°C selama 1 menit 10 detik) serta ekstensi akhir pada suhu 72°C selama 10 menit.
Deteksi Produk PCR
Produk PCR dideteksi dengan teknik elektroforesis gel agarosa 1,5% b/v.
HASIL DAN PEMBAHASAN
Data sekuens gen mabA (fabG) M. tuberculosis H37Rv yang diperoleh dari database
NCBI pada URL http://www.ncbi.nlm.nih.gov dengan kode genbank : U66801.1 dimasukkan ke dalam program Clone Manager Suite 6. Setelah itu dimasukkan beberapa sekuens primer yang akan dibandingkan dari hasil penelusuran pustaka (Morlock et al., 2003; Leung et al.,2006; Chen et al., 2011). Kemudian dilakukan analisis terhadap beberapa pasangan primer tersebut. Analisis hasil perbandingan dari beberapa primer dapat dilihat pada Tabel 1. Primer yang relatif paling memenuhi kriteria dilihat dari panjang primer, nilai Tm, %GC, stabilitas, jumlah hairpins, dimers dan runs adalah primer forward (mabA-inhA-promoter-FS) dengan urutan sekuen 5’-ACATACCTGCTGCGC AAT-3’ (18 nukleotida) dan primer reverse (mabA-inhA-promoter-R) dengan urutan sekuens 5’-CTCCGGTAACCAGGACTGAA-3’ (20 nuk-leotida) (Chen et al., 2011).
Tabel 1. Analisis Perbandingan Primer dengan Program Clone Manager Suite 6 (University of Groningen)
|
Kriteria |
Primer 1* |
Primer 2* |
Primer 3* |
Acuan Clone Manager Suite 6 | |||
|
Forward |
Reverse |
Forward |
Reverse |
Forward |
Reverse | ||
|
Panjang Primer (pb) |
18 |
20 |
20 |
20 |
21 |
21 |
Tidak ada |
|
Persen GC (%GC) |
50 |
55 |
65 |
65 |
52 |
52 |
50-60 |
|
Tm(°C) |
61 |
62 |
68 |
69 |
65 |
63 |
55-80 |
|
Dimer pada ujung 3’ |
2 |
1 |
2 |
0 |
1 |
2 |
< 3 |
|
Stabilitas (kcals) |
1,5 |
2,2 |
1 |
0,4 |
2 |
2,7 |
≥ 1,2 |
|
Runs |
2 |
2 |
3 |
5 |
3 |
2 |
< 3 |
|
Repeats |
2 |
Tidak ada |
Tidak ada |
2 |
Tidak ada |
2 |
<3 |
|
Hairpins |
Tidak ada |
Tidak ada |
Tidak ada |
Tidak ada |
Tidak ada |
Tidak ada |
Tidak ada |
|
False Priming (°C) |
Tidak ada |
Tidak ada |
Tidak ada |
17 |
Tidak ada |
Tidak ada |
Tidak ada |
|
Pustaka |
(Chen et al., 2011) |
(Morlock et al., 2003) |
(Leung et al., 2006) | ||||
Keterangan:
Primer 1*: Forward (mabA-inhA-promoter-FS): 5’-ACATACCTGCTGCGCAAT-3’
Reverse (mabA-inhA-promoter-R): 5’-CTCCGGTAACCAGGACTGAA-3’
Primer 2*: Forward (inhA-1): 5’-CCTCGCTGCCCAGAAAGGGA-3’
Reverse (inhA-2): 5’-ATCCCCCGGTTTCCTCCGGT-3’
Primer 3*: Forward (MabA-115F): 5’-ACAAACGTCACGAGCGTAACC-3’
Reverse (MabA+336R): 5’-GTTGGCGTTGATGACCTTCTC -3’
Hal ini terlihat pada Gambar 1 dan Gambar 2.
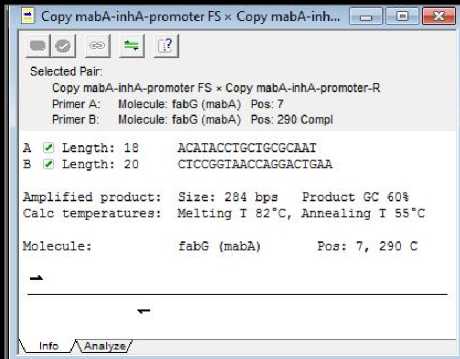
Gambar 1. Hasil Analisis Sekuens Pasangan Primer pada Program Clone Manager Suite 6
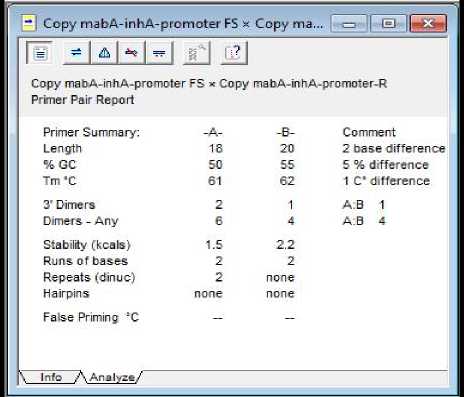
Gambar 2. Hasil Analisis Kriteria Pasangan Primer pada Program Clone Manager Suite 6
Dari hasil analisis yang telah dilakukan terlihat bahwa kedua primer telah memenuhi kriteria primer yang baik dilihat dari panjang primer seperti hasil yang ditunjukkan pada Gambar 1. Dimana secara berturut-turut primer forward (mabA-inhA-promoter-FS) dan primer reverse
(mabA-inhA-promoter-R) memiliki panjang 18 dan 20 nukleotida.
Menurut Innis dan Gelfand (1990), primer yang baik tersusun atas 18-28 nukleotida. Panjang minimal suatu primer adalah 18 nukleotida, jika lebih pendek akan mengakibatkan berkurangnya spesifisitas primer. Pada primer dengan ukuran yang pendek akan memungkinkan terjadinya mispriming (penempelan primer pada tempat lain yang tidak diinginkan) yang tinggi. Hal ini akan mempengaruhi efektifitas dan efisiensi proses PCR. Sedangkan, primer dengan panjang lebih dari 30 nukleotida tidak akan meningkatkan spesifisitas primer secara bermakna (Handoyo dan Ari, 2000).
Gambar 2 menunjukkan bahwa primer mabA-inhA-promoter-FS (forward) dan mabA-inhA-promoter-R (reverse) masing-masing memiliki nilai %GC 50% dan 55% dengan nilai melting temperature (Tm) masing-masing 61°C dan 62°C. Besarnya %GC suatu primer akan mempengaruhi nilai Tm dan Ta primer tersebut. Primer dengan panjang 18-28 nukleotida seharusnya memiliki %GC antara 50-60 % (Innis dan Gelfand, 1990).
Primer dengan nilai %GC yang tinggi akan memiliki nilai Tm yang tinggi pula karena terdapat lebih banyak ikatan hidrogen di dalamnya (3 ikatan hidrogen). Terdapat hubungan yang linear antara nilai %GC dengan nilai Tm dan Ta primer (Chen dan Janes, 2002). Pada kedua primer tersebut terdapat perbedaan nilai Tm sebesar 1°C, nilai ini masih dapat diterima didasarkan pada penelitian Barlett dan Stirling (2003) yang menyatakan nilai perbedaan Tm di antara dua primer yang diperbolehkan adalah 2-5 °C. Hal ini akan menjamin diperolehnya nilai Ta yang tepat untuk kedua primer (Bartlett dan Stirling, 2003).
Nilai Tm yang terlalu tinggi akan mengakibatkan terjadinya hibridisasi yang tidak adekuat antara primer dan templat dimana akan terbentuk ikatan yang terlalu kuat antara primer dengan DNA target sehingga produk PCR yang dihasilkan rendah namun lebih spesifik. Sedangkan nilai Tm yang terlalu rendah akan menyebabkan terbentuknya produk yang tidak spesifik karena tingginya penempelan primer pada daerah yang tidak tepat (mismatches) (Lee et al., 1997; Borah, 2011). Menurut Burden dan Whitney (1995) untuk primer dengan panjang 18-22 nukleotida dan %GC 45-55°C umumnya memiliki rentang nilai Tm 55-
80 °C. Sehingga dapat disimpulkan bahwa primer yang akan digunakan telah memenuhi kriteria %GC dan Tm yang baik.
Dimers yang terjadi pada ujung 3’ harus dihindari karena akan menyebabkan terjadinya pemanjangan primer PCR dengan menggunakan primer kedua sebagai templat yang nantinya membentuk struktur sekunder yang disebut dengan primer-dimers. Hal ini akan mengakibatkan primer kehilangan fungsinya sehingga amplikon yang dihasilkan akan berkurang jumlahnya (Clone Manager Suite 6). Dari hasil analisis in silico nilai dimers masing-masing primer telah memenuhi kriteria primer yang baik.
Kriteria lain yang harus dipenuhi oleh primer adalah dalam hal stabilitas. Parameter ini berkaitan dengan perbedaan nilai stabilitas antara ujung 5’ dan 3’ primer. Afinitas maksimal primer terhadap templatnya terdapat pada ujung 5’ primer. Karenanya, ujung 3’ primer hanya perlu berikatan secukupnya sehingga DNA polimerase dapat memulai replikasi.
Namun, jika ujung 3’ primer mempunyai stabilitas termal yang tinggi, hal tersebut akan mengakibatkan terjadinya penempelan ujung 3’ primer pada templat dan selanjutnya memulai replikasi, walaupun ujung 5’ tidak cocok. Hal ini akan menyebabkan penurunan spesifisitas hibridisasi. Sehingga nilai stabilitas ujung 5’ harus lebih besar dibandingkan ujung 3’ (Clone Manager Suite 6). Dari hasil analisis in silico terhadap primer mabA-inhA-promoter-FS dan mabA-inhA-promoter-R dengan nilai stabilitas masing-masing sebesar 1,5 kcals dan 2,2 kcals. Sehingga dapat disimpulkan bahwa kedua primer tersebut telah memenuhi kriteria stabilitas yang ditetapkan.
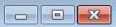
Hasil analisis terhadap kualitas masing-masing primer secara terpisah ditunjukkan pada Gambar 3 dan 4.
Gambar 3 dan 4 secara terpisah menunjukkan bahwa primer forward dan reverse tersebut telah memenuhi kriteria runs, repeats dan hairpins sesuai yang dipersyaratkan program Clone Manager Suite 6 (University of Groningen) Runs, repeats dan hairpins merupakan struktur sekunder yang terbentuk akibat adanya interaksi intramolekular dan intermolekular. Struktur sekunder yang terbentuk ini akan mengurangi ketersediaan primer untuk berinteraksi dengan
templat target. Jumlah maksimal runs dan repeats yang diperbolehkan adalah 4 pb (Borah, 2011).
"* Copy mabA-inhA-promoter FS

PCR Primer: Copy mabA-inhA-promoter FS Pos: 7 fabG (mabA)
One Primer Report
Criteria Used: PCR Primer
%GC Tm'C
-
3 , Dimers Dimers-Any
Stability Runs
Repeats
Hairpins
Range 50-60
Range 55-80
-
< 3 matches 3 end
-
< 7 adj homol bases
>= 1.2 kcals 5 vs3-
-
< 3 base runs
-
< 3 dinuc repeats
Annealing 55*C
This Meets
Primer Criteria
50 Yes
61 Yes
2 Yes
6 Yes
-
1.5 Yes
-
2 Yes
-
2 Yes
none Yes
Worst-case False Priming sC
∖ Info ∕∖ Edit 7∖Aπalyze/
Gambar 3. Hasil Analisis Kualitas Primer Forward pada Program Clone Manager Suite 6
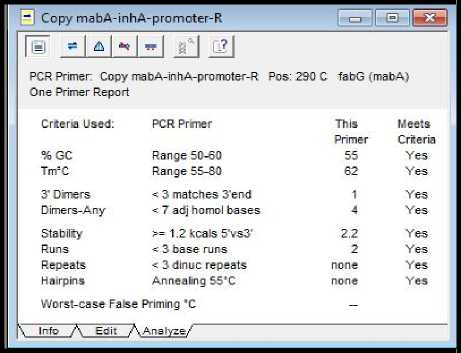
Gambar 4. Hasil Analisis Kualitas Primer Reverse pada Program Clone Manager Suite 6
Kemudian dilakukan simulasi pada sepasang primer tersebut secara in silico dengan menggunakan analyze mix wizard dengan menggunakan gen fabG (mabA) M. tuberculosis sebagai DNA templat. Hasil analisis yang diperoleh terdapat pada Gambar 5.
Hasil analisis tersebut menunjukkan bahwa produk PCR (amplikon) yang akan dihasilkan memiliki ukuran 284 pb yang terletak
pada posisi nukleotida 7 sampai dengan 290. Berdasarkan studi pustaka dan acuan kriteria dari Clone Manager Suite 6, diperoleh kesimpulan bahwa secara keseluruhan primer yang akan digunakan telah memenuhi kriteria primer yang baik dan daerah target yang diharapkan.
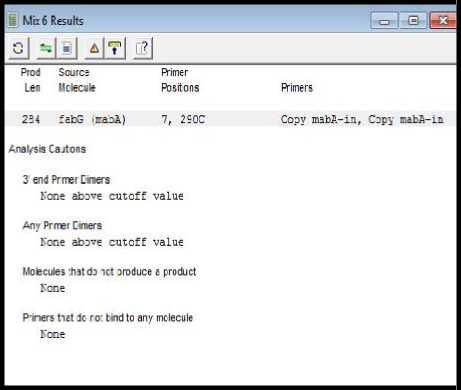
Gambar 5. Hasil Analisis Sepasang Primer dengan gen fabG (mabA) sebagai DNA templat
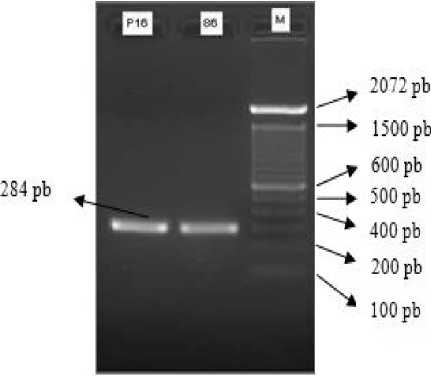
Keterangan :
Marker DNA ladder 100 pb (M); isolat P16 (P16); isolat 86 (86).
Gambar 6. Elektroforegram amplifikasi fragmen promoter inhA isolat P16 dan 86 pada suhu annealing 54°C
Selain analisis in silico, dilakukan juga deteksi secara in vitro terhadap pasangan primer tersebut melalui proses PCR dan deteksi produk PCR dengan teknik elektroforesis gel agarosa 1,5%. Kondisi PCR yang diaplikasikan adalah, denaturasi awal pada suhu 95°C selama 15 menit , amplifikasi sebanyak 45 siklus (denaturasi pada suhu 94°C selama 1 menit, annealing pada suhu 55°C selama 1 menit 20 detik dan ekstensi pada suhu 72°C selama 1 menit 10 detik) serta ekstensi akhir pada suhu 72°C selama 10 menit. DNA templat yang digunakan berasal dari DNA kromosomal yang diisolasi dari isolat P16 dan 86 M. tuberculosis Multi Drug Resistance. Hasil yang diperoleh berupa elektroforegram yang diperlihatkan pada Gambar 6.
Hasil elektroforegram pada Gambar 6 menunjukkan bahwa primer mabA-inhA-promoter-FS (forward) dan mabA-inhA-promoter-R (reverse) telah berhasil mengamplifikasi daerah promoter inhA dengan ukuran produk 284 pb sesuai dengan hasil analisis secara in silico.
SIMPULAN DAN SARAN
Simpulan
-
1. Primer mabA-inhA-promoter-FS (forward) dengan urutan nukleotida 5’-ACATACC TGCTGCGCAAT-3’ dan primer mabA-inhA-promoter-R (reverse) 5’- CTCCGGT
AACCAGGACTGAA-3’ (Chen et al., 2011) memenuhi kriteria primer yang baik dilihat dari panjang primer, %GC, Tm, dimer pada ujung 3’, stabilitas, jumlah runs, repeats, hairpins dan false priming.
-
2. Pasangan primer berhasil mengamplifikasi daerah promoter inhA dan menghasilkan produk PCR (amplikon) dengan ukuran 284 pb sesuai dengan hasil analisis secara in silico pada program Clone Manager Suite 6.
Saran
Perlu dilakukan analisis lebih lanjut terhadap produk PCR (amplikon) dengan teknik sekuensing untuk mengetahui urutan nukleotida amplikon.
UCAPAN TERIMA KASIH
Penulis mengucapkan terimakasih kepada semua pihak yang telah membantu sehingga penelitian ini dapat terlaksana dengan baik.
DAFTAR PUSTAKA
Bartlett, J. M. S. dan Stirling, D., 2003, Methods in Molecular Biology, Vol. 226 : PCR Protocols 2nd Editions, Human Press Inc., Totowa, NJ, p. 81-604
Borah, P., 2011, Primer Designing for PCR, Science Vision, 11 (3) : 134-136
Burden, D.W. dan Whitney, D.B., 1995, Biotechnology Proteins to PCR: Proteins to PCR : A Course in Strategies and Lab Technique, Birkhauser, USA, p. 304
Chen, B.Y. dan Janes, H. W. 2002. Methods in Molecular Biology Vol. 192: PCR Cloning Protocols 2nd Edition, Humana Press Inc., Totowa, NJ, p. 19-21
Chen, X., Kong, F., Wang, Q., Li, C., Zhang, J., dan Gilbert, G.L., 2011, Rapid Detection of Isoniazid, Rifampin, and Ofloxacin Resistance in Mycobacterium tuberculosis Clinical Isolates Using High-Resolution Melting Analysis, Journal of Clinical Microbiology, 49 (10) : 3450–3457
Dipiro J.T., Talbert, R.T., Yee, G.C., Matzke, G.R., Wells, B.G., dan Posey, L.M. Michael P., 2008, Pharmacotherapy: A Pathophysiologic Approach, 7th Edition, The McGraw Hill Companies Inc., United States of America, p. 1839-1854
Handoyo, D. dan Ari, R., 2000, Prinsip Umum dan Pelaksanaan Polymerase Chain Reaction (PCR), Unitas, 9(1) : 17-29
Innis, M.A. dan Gelfand, D.H., 1990, PCR Protocols: A Guide to Methods and Applications, Academic Press Inc., London, UK, p. 3-11
Lee, H. H., Morse, S.A., and Olsvik, Ø., 1997, Nucleic Acid Amplification Technologies: Application to Disease Diagnosis, Eaton Publishing, USA
Leung, E. T. Y., Ho, P. L., Yuen, K. Y., Woo, W. L., Lam, T. H., Kao, R. Y., Seto, W. H dan Yam, W. C., 2006, Molecular Characterization of Isoniazid Resistance in Mycobacterium tuberculosis: Identification of a Novel Mutation in inhA, Journal of Clinical Microbiology, 50 (3) : 10751078
Machado, D., Perdigao, J., Ramos, J., Couto, I., Portugal, I., Ritter, C., Boettger E.C., dan Viveiros, M., 2013, High-level Resistance to Isoniazid and Ethionamide in Multidrug-resistant Mycobacterium tuberculosis of the Lisboa Family is Associated with inhA Double Mutations, Journal of Clinical Microbiology, p. 1-5
Morlock, G.P., Metchock, B., Sikes, D., Crawford, J.T., and Cooksey, R.C., 2003, ethA, inhA, and katG Loci of EthionamideResistant Clinical Mycobacterium tuberculosis Isolates, Journal of Clinical Microbiology, 47 (12) : 3799-3805
World Health Organization, 2012, Global Tuberculosis Report 2012, World Health Organization, Geneva, Switzerland, p. 166
123
Discussion and feedback