POLYMERASE CHAIN REACTION (PCR) UNTUK IDENTIFIKASI GEN bfpA, stx1, dan stx2 DARI BAKTERI Escherichia coli YANG TERISOLASI DARI SPESIMEN USAP DUBUR PENJAMAH MAKANAN DI KOTA DENPASAR TAHUN 2015
on

JMU
ISSN: 2597-8012 JURNAL MEDIKA UDAYANA, VOL. 9 NO.9,SEPTEMBER, 2020

Diterima:06-08-2020 Revisi:12-08-2020 Accepted: 14-09-2020
POLYMERASE CHAIN REACTION (PCR) UNTUK IDENTIFIKASI GEN bfpA, stx1, dan stx2 DARI BAKTERI Escherichia coli YANG TERISOLASI DARI SPESIMEN USAP DUBUR PENJAMAH MAKANAN DI KOTA DENPASAR TAHUN 2015
I Gusti Agung Indah Pradnyani R.S1, Ni Nyoman Sri Budayanti2 `1Program Studi Pendidikan Dokter, Fakultas Kedokteran Universitas Udayana 2Bagian/SMF Mikrobiologi Klinik RSUP Sanglah
Email: indah.pradnyani@gmail.com
ABSTRAK
Diarrheagenic Escherichia coli (DEC) merupakan bakteri penyebab tersering pada diare yang terkait dengan foodborne disease. Penjamah makanan, sebagai seseorang yang terlibat dalam proses penangangan makanan, berperan besar dalam transmisi diare, salah satunya adalah sebagai pembawa (carrier) DEC. Penelitian ini bertujuan untuk mengidentifikasi gen bfpA, stx1, dan stx2 dari E.coli yang terisolasi dari spesimen usap dubur penjamah makanan di Denpasar, Bali. Penelitian deskriptif observasional potong lintang ini menggunakan sampel sebanyak 17 isolat bakteri Escherichia coli yang didapat dari usap dubur penjamah makanan di Denpasar, Bali pada tahun 2015. Setelah dilakukan ekstraksi DNA, Polymerase Chain Reaction (PCR) digunakan untuk mengamplifikasi DNA tersebut dengan tujuan mengidentifikasi keberadaan gen bfpA, stx1, dan stx2. Hasil amplifikasi kemudian divisualisasi dengan bantuan elektroforesis gel agarose. Dalam penelitian ini, tidak terdeteksi adanya penjamah makanan pembawa gen bfpA yang mewakili patotipe EPEC, sementara lima penjamah makanan terdeteksi sebagai pembawa EHEC dengan rincian satu penjamah makanan terdeteksi keberadaan gen stx1 saja dan pada empat penjamah makanan lainnya terdeteksi keberadaan gen stx2 saja. Tidak ada penjamah makanan yang terdeteksi keberadaan gen stx1 dan stx2 sekaligus. Gen bfpA dan stx1 lebih optimal jika diamplifikasi dengan suhu dan durasi denaturasi yang lebih tinggi, serta dengan suhu annealing yang lebih rendah jika dibandingkan dengan stx2.
Kata kunci: PCR, EPEC, EHEC, Penjamah Makanan.
ABSTRACT
Diarrheagenic Escherichia coli (DEC) is known as the most common etiology of foodborne diarrhea. Food handlers play important role in the transmission of this disease, namely as the asymptomatic carriers of DEC. This study aimed to identify the presence of bfpA, stx1, and stx2 gene isolated from rectal swab specimens of food handlers, which would determine whether they asymptomatically carry these genes. This cross-sectional descriptive study used 17 Escherichia coli isolates collected as rectal swab specimens from food handlers residing in Denpasar, Bali in 2015. DNA extraction was then performed, followed by amplification by means of Polymerase Chain Reaction (PCR) to detect the presence of bfpA, stx1, and stx2 gene. Agarose gel electrophoresis was used to visualize the amplicons. In this study, no food handlers were detected as the carrier of bfpA which represented EPEC pathotype. Five food handlers were positive for carrying
EHEC, where one carried only stx1 and four others carried stx2 only. There were no food handlers in which stx1 and stx2 were simultaneously detected. Both bfpA and stx1 gene achieved optimal amplification in higher denaturation temperature, longer denaturation period, and lower annealing temperature compared to stx2.
Keywords: PCR, EPEC, EHEC, Food Handlers.
PENDAHULUAN
Selain mengandung gizi yang cukup, makanan yang baik dikonsumsi juga harus terjaga keamanan dan kebersihannya. Menurut data yang dirilis World Health Organization (WHO), ada lebih dari 200 jenis penyakit yang diketahui dihantarkan melalui makanan. Diare merupakan salah satu dari penyakit-penyakit tersebut yang menyebabkan kematian pada kurang lebih 1.800.000 orang dan diduga disebabkan oleh air dan makanan yang terkontaminasi oleh mikroorganisme penyebab diare.1
Di dunia, sebagian besar kasus diare yang dihantarkan melalui makanan disebabkan oleh kontaminasi bakteri. Escherichia coli (E. coli) merupakan bakteri penyebab diare terkait makanan yang paling umum. Bakteri ini diketahui memiliki beberapa patotipe yaitu Enteropathogenic E. coli (EPEC), Enterotoxigenic E. coli (ETEC),
Enteroaggregative E. coli (EAEC), Enteroinvasive E. coli (EIEC), dan Shiga toxin-producing E. coli (STEC) yang juga dikenal sebagai Enterohaemorrhagic E. coli (EHEC). Diantara kelima patotipe tersebut, ETEC adalah patotipe yang paling banyak ditemukan, diikuti oleh EPEC dan STEC/EHEC.2
Sebagai penyakit yang ditransmisikan melalui jalur fekal-oral, penyebaran diare sebagai foodborne disease sangat dipengaruhi oleh kebersihan dalam proses pengolahan makanan. Penjamah makanan (food handlers), yang didefinisikan sebagai orang yang secara langsung berhubungan dengan makanan dan peralatan mulai dari tahap persiapan, pembersihan, pengolahan, pengangkutan, sampai dengan penyajiannya, berpotensi menyebabkan kontaminasi pada makanan yang dijamahnya apabila penjamah makanan tersebut tidak menerapkan higiene dan sanitasi yang baik selama ia bekerja. Penjamah makanan dapat pula berperan sebagai pembawa (carrier) beberapa
patogen, salah satunya adalah Escherichia coli. Keadaan ini dapat memperbesar risiko kontaminasi pada makanan yang dijamah.3,4 Untuk mengetahui apakah seorang penjamah makanan merupakan pembawa patogen atau
bukan, diperlukan suatu metode identifikasi yang akurat. Hal ini penting agar langkah-langkah higiene dan sanitasi khusus bisa diterapkan pada penjamah makanan tersebut sebagai bentuk pencegahan kontaminasi pada makanan yang dijamah, juga dalam penatalaksanaan misalnya dalam pemilihan antibiotik yang sesuai seandainya ia terbukti sebagai pembawa patogen. Saat ini, Polymerase Chain Reaction (PCR) merupakan salah satu metode yang umum digunakan untuk mengidentifikasi patogen dari spesimen yang tersedia. Metode ini menguntungkan dalam identifikasi patogen seperti bakteri E. coli yang sulit dibedakan antara patotipe yang satu dengan yang lain jika menggunakan metode konvensional, misalnya dengan mengamati koloni bakterinya saja, serta lebih efisien seperti pada PCR multipleks yang memungkinkan identifikasi cepat terhadap lebih dari satu patotipe sekaligus. Target PCR adalah gengen virulensi yang khas untuk suatu patotipe tertentu, misalnya gen eaeA dan bfpA untuk identifikasi EPEC, serta gen stx1 dan/atau stx2 untuk identifikasi EHEC.5 Identifikasi keberadaan patotipe ETEC dan EAEC dari E. coli pada penjamah makanan menggunakan metode PCR telah dilakukan,6 namun belum ada penelitian terhadap keberadaan patotipe lainnya seperti EPEC atau EHEC.
BAHAN DAN METODE
Penelitian ini merupakan penelitian deskriptif observasional potong-lintang yang dilakukan di Laboratorium Mikrobiologi dan Laboratorium Biologi Molekuler Fakultas Kedokteran Universitas Udayana, mulai bulan Maret sampai Oktober 2016. Total sampel yang digunakan dalam penelitian ini adalah sebanyak 17 sampel. Sampel tersebut merupakan isolat bakteri E. coli yang didapat dari usap dubur penjamah makanan yang dipilih secara acak di restoran dan rumah makan di Denpasar, Bali pada tahun 2015. Isolat E. coli yang digunakan dalam penelitian ini didapat dari penjamah makanan yang berusia diatas 18 tahun, tidak memiliki riwayat diare, serta tidak sedang
dalam pengobatan diare atau konsumsi antibiotik. Isolat bakteri E. coli yang tidak tumbuh homogen pada proses subkultur dieksklusi dari penelitian ini.
Stok isolat bakteri E. coli yang telah diisolasi dari sampel usap dubur kemudian ditumbuhkan dalam media agar Mueller Hinton (MH) selama 18-24 jam pada suhu 37oC. Hasil kultur kemudian diidentifikasi ulang menggunakan metode pengecatan Gram dan uji biokimia. Dari hasil kultur, diambil koloni kersangka E. coli dan dimasukkan dalam tabung ukuran 1,5 mL berisi 200 μL Tris EDTA (TE) dengan pH 8. Tabung tersebut kemudian dimasukkan dalam water bath 100oC selama 10 menit. Selanjutnya, tabung dimasukkan dalam es dan disentrifugasi dengan kecepatan 8000 rpm selam 1 menit. Supernatan diambil sebanyak 100 μL dan disimpan pada suhu -80oC hingga siap digunakan.
Dalam penelitian ini, gen yang dijadikan target untuk identifikasi E. coli pada sampel adalah bfpA untuk EPEC, serta stx1 dan/atau stx2 untuk EHEC. Primer-primer yang digunakan dalam penelitian ini merupakan sekuens primer yang telah digunakan dalam penelitian sebelumnya5 yang disajikan dalam Tabel 1.
|
Tabel 1. Daftar Primer, Sekuens, dan | ||||
|
Ukurannya Penelitian. |
yang Digunakan dalam | |||
|
Target gen |
Primer |
Sekuens (5'-3,) |
Ukuran W |
Referensi |
|
EPJOOF |
Ggaagtcaaattc atggggg | |||
|
⅛Λ |
EPJOORbt EH132F |
ggaatcagacgca gactggt Acttctcgactgca aagacgtatg |
301 |
Taniuchi |
|
stxl |
EH132Rbt EH255F |
acaaattatcccct gagccactatc ggcactgtctgaa actgctcc |
132 |
dkk (2012)i |
|
stx2 |
EH255Rbt |
tcgccagttatctg acattctg |
255 | |
Amplifikasi dilakukan dengan prosedur sesuai seperti pada penelitian sebelumnya5 (Tabel 2). Hasil amplifikasi kemudian divisualisasi dengan bantuan elektroforesis gel agarose di bawah sinar UV. Setelah sampel diamplifikasi dengan PCR dan divisualisasi dengan gel agarose, data
diambil dan diolah dengan program pengolahan data untuk mengetahui prevalensi EPEC dan EHEC yang terdeteksi pada sampel. Data kemudian ditampilkan dalam bentuk narasi, tabel, dan gambar. Penelitian ini telah dinyatakan laik etik oleh Komisi Etika Penelitian kedokteran Universitas Udayana/Rumah Sakit Umum Pusat Sanglah Denpasar dengan nomor 1314/UN.14.2/Litbang/2016.
HASIL
Identifikasi gen bfpA dilakukan pertama kali dengan program PCR seperti yang tersaji dalam Tabel 2. Dari 17 sampel yang diidentifikasi, terdapat satu sampel (sampel nomor 12) yang menunjukkan gambaran pita spesifik untuk bfpA (301 pasang basa) pada gel elektroforesis.
Namun, karena pita yang nampak pada elektroforesis pertama ini masih sangat tipis, maka dilakukan re-PCR dengan program PCR yang sama namun dengan template DNA berupa hasil PCR pertama ini. Hasil re-PCR menunjukkan adanya pita yang spesifik untuk bfpA pada tiga sampel (sampel nomor 14, 15, dan 16), namun didapatkan pula pita nonspesifik dalam jumlah yang cukup banyak. Oleh karena banyaknya pita nonspesifik yang muncul pada hasil elektroforesis re-PCR, maka dilakukan optimasi dengan menurunkan suhu annealing sebanyak 2oC, menaikkan suhu denaturasi sebanyak 1oC, dan menambah durasi denaturasi sebanyak 30 detik. Program PCR kedua ini ditampilkan dalam Tabel 3.
|
Tabel 2. Program PCR Pertama untuk Identifikasi Gen bfpA, stx1, dan stx2. | ||
|
Program PCR |
Durasi |
Suhu |
|
Pre-denaturasi |
3 menit |
9?C |
|
Denaturasi |
30 detik |
94cC |
|
Annealing |
30 detik |
50°C |
|
Ekstensi |
60 detik |
7?C |
|
Ekstensi final |
5 menit |
72°C |
|
Putarari |
40 kali | |
Tabel 3. Program PCR Kedua untuk Identifikasi Gen bfpA dan stx1.
|
Program PCR |
Durasi |
Suhu |
|
Pre-denaturasi |
3 memt |
9?‘C |
|
Denaturasi |
60 detik |
95C |
|
Annealing |
30 detik |
48“C |
|
Ekstensi |
60 detik |
72°C |
|
Ekstensi final |
5 memt |
72“C |
|
Putaran |
40 kali |
Sampel yang digunakan adalah tiga sampel yang sebelumnya telah terdeteksi keberadaan bfpA (sampel nomor 14, 15, dan
16). Pada hasil elektroforesis kedua ini, tampak ada pita yang muncul pada semua sampel kecuali kontrol negatif, namun tidak ada yang spesifik untuk bfpA. Hasil elektroforesis ditampilkan pada Gambar 1.
Proses identifikasi gen stx1 mengalami kendala serupa dengan identifikasi gen bfpA sehingga dilakukan langkah-langkah yang sama untuk mengatasinya. Identifikasi pertama untuk gen stx1 dilakukan dengan program PCR yang sama seperti Tabel 2. Dari 17 sampel yang diidentifikasi, terdapat dua sampel (sampel nomor 15 dan 17) yang menunjukkan gambaran pita tipis yang spesifik untuk stx1. Untuk mendapatkan hasil elektroforesis yang lebih jelas, maka dilakukan re-PCR dengan tetap menggunakan program yang sama seperti pada Tabel 2. Hasil re-PCR menunjukkan adanya pita spesifik pada dua sampel (sampel nomor 14 dan 15), disertai beberapa pita nonspesifik. Oleh karena itu, maka dilakukan optimasi dengan program PCR seperti yang ditampilkan pada Tabel 3. Dari hasil elektroforesis kedua ini, tampak ada satu sampel yang mengandung pita spesifik untuk stx1 yaitu pada sampel S1TE16R (sampel nomor 14). Hasil elektroforesis ditampilkan pada Gambar 2.
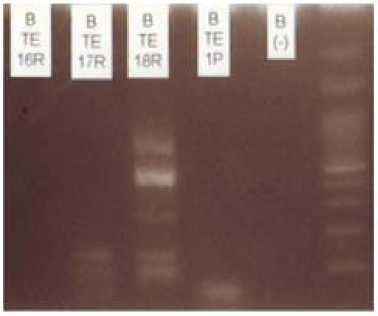
Gambar 1. Hasil Elektroforesis Kedua untuk Identifikasi Gen bfpA (sampel nomor 14,15, dan 16).
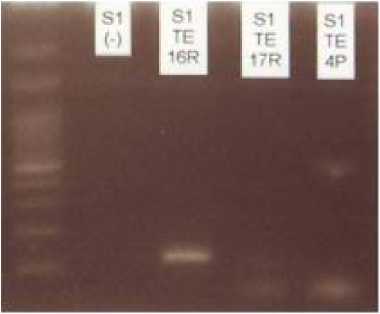
Gambar 2. Hasil Elektroforesis Kedua untuk Identifikasi Gen stx1 (sampel nomor 14 dan 15).
Identifikasi pertama untuk gen stx2 dilakukan dengan program PCR yang sama seperti pada Tabel 2. Identifikasi gen ini tidak mengalami kendala seperti pada identifikasi gen bfpA dan stx1. Dari 17 sampel yang diidentifikasi, terdapat empat sampel yang menunjukkan gambaran pita yang spesifik untuk stx2 yaitu pada sampel S2TE14R (sampel nomor 12), S2TE15R (sampel nomor 13), S2TE17R (sampel nomor 15), dan S2TE18R (sampel nomor 16). Pita yang terdeteksi sudah cukup jelas dan spesifik, sehingga tidak dilakukan optimasi untuk gen stx2. Hasil elektroforesis ditampilkan pada Gambar 3.
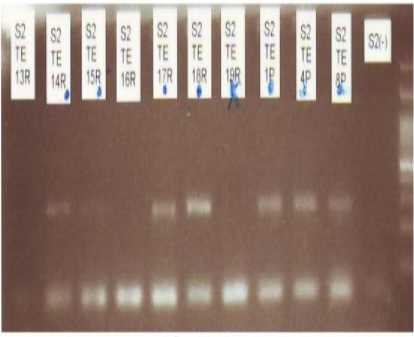
Gambar 3. Hasil Elektroforesis Pertama untuk Identifikasi Gen stx2 (sampel 11-17).
Dari sampel akhir sejumlah 17 sampel, tidak terdeteksi adanya gen bfpA (yang mewakili patotipe EPEC), sementara lima sampel positif terdeteksi sebagai EHEC dengan rincian sebagai berikut: satu sampel terdeteksi keberadaan gen stx1 saja dan pada empat sampel lainnya terdeteksi keberadaan gen stx2 saja. Tidak ada sampel yang terdeteksi keberadaan gen stx1 dan stx2 sekaligus.
PEMBAHASAN
Proses amplifikasi DNA template akan menjadi lebih efisien jika annealing dilakukan pada suhu yang relatif rendah, namun dengan konsekuensi berupa meningkatnya kemungkinan terjadinya mispriming secara signifikan.7 Dalam penelitian ini, suhu annealing diturunkan dengan tujuan membantu proses terbentuknya ikatan antara primer dan target sekuens dari DNA template dengan harapan amplifikasi template DNA mengalami peningkatan dan pita yang tampak pada hasil elektroforesis menjadi lebih terang dan jelas.8 Berdasarkan hasil penelitian ini, dapat dilihat bahwa program PCR pada Tabel 2 bersifat lebih optimal jika digunakan untuk mengamplifikasi gen stx2, sementara gen bfpA dan stx1 lebih optimal jika diamplifikasi dengan suhu dan durasi denaturasi yang lebih tinggi, serta dengan suhu annealing yang lebih rendah. Walaupun penelitian ini menggunakan set primer yang sama dengan penelitian sebelumnya5, terdapat perbedaan program PCR dalam penelitian ini yang kemungkinan disebabkan oleh perbedaan alat-alat serta bahan yang digunakan dalam proses amplifikasi.
Patotipe EPEC yang memiliki gen bfpA disebut EPEC tipikal, sementara yang tidak memilikinya disebut EPEC atipikal. EPEC atipikal merupakan galur penyebab diare terbanyak dan ditemukan sedikit lebih banyak pada orang sehat dibandingkan orang dengan diare, yang menunjukkan kecenderungan adanya asymptomatic carrier.9 Oleh karena seluruh sampel yang digunakan dalam penelitian ini berasal dari orang sehat, maka kemungkinan hal ini menjadi penyebab tidak terdeteksinya gen bfpA dalam penelitian ini.
Dahulu, EPEC tipikal diyakini sebagai penyebab utama diare di negara berkembang sementara EPEC atipikal lebih umum ditemukan di negara maju. Namun, data penelitian terbaru menunjukkan bahwa prevalensi EPEC atipikal cenderung meningkat baik di negara berkembang maupun negara maju.10 Fakta tersebut kemungkinan juga menjadi alasan tidak terdeteksinya gen bfpA dalam penelitian ini. Hasil penelitian ini memiliki dua implikasi. Pertama, kemungkinan bahwa galur EPEC yang lebih umum ditemukan di Indonesia adalah galur atipikal. Implikasi kedua adalah bahwa kemungkinan Indonesia telah mengalami pergeseran pola prevalensi galur EPEC dari pola prevalensi negara berkembang (EPEC tipikal lebih umum) menuju pola prevalensi negara maju (EPEC atipikal lebih umum). Berkurangnya prevalensi EPEC tipikal bisa jadi merupakan tanda keberhasilan dalam upaya perbaikan aspek kesehatan publik.11 Namun, kedua implikasi ini perlu dibuktikan lebih lanjut kebenarannya sebab tidak tersedia data mengenai prevalensi kedua galur EPEC di Indonesia.
Patotipe EHEC/STEC dapat diidentifikasi melalui keberadaan gen stx1 dan/atau stx2. Penelitian sebelumnya menunjukkan bahwa EHEC/STEC lebih umum ditemukan pada orang sehat.12 Pada penelitian ini, proporsi gen stx1 yang terdeteksi lebih sedikit jumlahnya dibandingkan dengan gen stx2. Gen stx2 merupakan gen penyandi faktor virulensi yang dikaitkan dengan penyakit yang lebih berat pada manusia.13 Oleh karena itu, fakta bahwa seluruh sampel penelitian ini didapat dari penjamah
makanan dan empat dari lima sampel tersebut terdeteksi membawa gen stx2
mengimplikasikan bahwa perlu
dipertimbangkan untuk melakukan tindakan pencegahan kontaminasi yang lebih lanjut
pada orang-orang yang menjadi sumber keempat sampel tersebut.
Asymptomatic carrier merupakan salah satu 3.
sumber kontaminasi DEC terbesar.14 Seseorang bisa menjadi pembawa DEC setelah melalui proses interaksi yang kompleks antara kerentanan pejamu (contohnya umur, status gizi, serta imunitas) dan juga faktor lingkungan (contohnya higiene yang buruk serta kontaminasi 4.
feses).9 Oleh karena itu, hasil penelitian ini mengimplikasikan bahwa para pejamu makanan harus menjaga kondisi kesehatan dirinya serta kebersihan lingkungannya 5.
untuk mengurangi peluang menjadi pembawa DEC yang akhirnya berpotensi mengontaminasi makanan yang ditangani.
SIMPULAN
Dalam penelitian ini, tidak terdeteksi adanya penjamah makanan pembawa gen bfpA yang mewakili patotipe EPEC, sementara lima penjamah makanan terdeteksi sebagai pembawa EHEC dengan rincian satu penjamah makanan terdeteksi keberadaan 6.
gen stx1 saja dan pada empat penjamah makanan lainnya terdeteksi keberadaan gen stx2 saja. Tidak ada penjamah makanan yang terdeteksi keberadaan gen stx1 dan stx2 sekaligus. Gen bfpA dan stx1 lebih optimal jika diamplifikasi dengan suhu dan durasi denaturasi yang lebih tinggi, serta dengan suhu annealing yang lebih rendah jika 7.
dibandingkan dengan stx2.
SARAN
Diharapkan ada penelitian lebih lanjut 8.
dengan jumlah sampel yang lebih besar agar hasil penelitian dapat digeneralisasi ke populasi yang lebih luas.
DAFTAR PUSTAKA
-
1. World Health Organization. Five Keys to Safer Food Manual. France: WHO Press, 2006. Diunduh dari:
http://www.who.int/foodsafety/publica tions/5keysmanual/en/. (diakses 18 Januari 2016). 9.
-
2. Kirk M., Pires S., Black R., Caipo M., Crump J., Devleesschauwer B., Dofer, D., Fazil, A., Fischer-Walker, C., Hald, T., Hall, A., Keddy, K., Lake R., Lanata C., Torgerson, P., Havelaar, A., dan Angulo, F. World Health Organization Estimates of the Global and Regional Disease Burden of 22 Foodborne Bacterial, Protozoal, and 10.
Viral Diseases, 2010: A Data
Synthesis. PLoS Med. 2015; 12(12):7-11.
Kementerian Kesehatan Republik Indonesia. Keputusan Menteri
Kesehatan Republik Indonesia Nomor 715 Tahun 2003 tentang Persyaratan Hygiene Sanitasi Jasaboga. Jakarta: Kementerian Kesehatan Republik Indonesia: 2003.
Rane, S. Street Vended Food in Developing World: Hazard Analyses. Indian J Microbiol. 2011; 51(1):100-106.
Taniuchi, M., Walters, C., Gratz, J., Maro, A., Kumburu, H.,
Serichantalergs, O., Sethabutr, O., Bodhidatta, L., Kibiki, G., Toney, D., Berkeley, L., Nataro, J., Houpt, E. R. Develoment of a Multiplex Polymerase Chain Reaction Assay for
Diarrheagenic Escherichia coli and Shigella spp. and Its Evaluation on Colonies, Culture Broths, and Stool. J Diag Microbio. 2012; 73:121-128.
Gitaswari, D. A. I. Identifikasi Subtipe Enterotoxigenic Escherichia coli dan Enteroaggregative Escherichia coli dari Spesimen Usap Dubur Penjamah Makanan di Denpasar Menggunakan Polymerase Chain Reaction.
Denpasar: Fakultas Kedokteran
Universitas Udayana: 2015.
Green, M. dan Sambrook, J. Molecular Cloning: A Laboratory Manual. 4th ed. Cold Spring Harbor, N.Y.: Cold Spring Harbor Laboratory Press: 2012.
ThermoFisher Scientific Inc. PCR Troubleshooting Guide|Thermo Fisher Scientific. ThermoFisher Scientific Inc., 2017. Tersedia di:
https://www.thermofisher.com/id/en/h ome/life-science/cloning/cloning-learning-center/invitrogen-school-of-molecular-biology/pcr-education/pcr-reagents-enzymes/pcr-troubleshooting.html. (Diakses 17 Januari 2017).
Contreras, C., Ochoa, T., Lacher, D., DebRoy, C., Navarro, A., Talledo, M., Donnenberg, M., Ecker, L., Gil, A., Lanata, C. dan Cleary, T. Allelic variability of critical virulence genes (eae, bfpA and perA) in typical and atypical enteropathogenic Escherichia coli in Peruvian children. J Med Microbio. 2010; 59(1):25-31.
Ochoa, T., Barletta, F., Contreras, C. dan Mercado, E. New insights into the
epidemiology of enteropathogenic Escherichia coli infection. J Trans Trop Med H. 2008; 102(9):852-856.
-
11. Araujo, J., Tabarelli, G., Aranda, K., Fabbricotti, S., Fagundes-Neto, U., Mendes, C. dan Scaletsky, I. Typical Enteroaggregative and Atypical Enteropathogenic Types of Escherichia coli Are the Most Prevalent Diarrhea-Associated Pathotypes among
Brazilian Children. J Clin Microbio. 2007; 45(10):3396-3399.
-
12. Moura, C., Fregolente, M., Martini, I., Domingos, D., Silva, E., Ferraz, M., Gatti, M. dan Leite, D. Prevalence of enteropathogens in normal feces from healthy children at an infant day care in Brazil. J Infect Dev C. 2012; 6(02):177-179.
-
13. Pradel, N., Bertin, Y., Martin, C. dan Livrelli, V. Molecular Analysis of Shiga Toxin-Producing Escherichia coli Strains Isolated from Hemolytic-Uremic Syndrome Patients and Dairy Samples in France. Appl Environ Microbio. 2008; 74(7):2118-2128.
-
14. Bardiau, M., Szalo, M. dan Mainil, J. Initial adherence of EPEC, EHEC and VTEC to host cells. J Vet R. 2010; 41(5):57.
https://ojs.unud.ac.id/index.php/eum
doi:10.24843.MU.2020.V9.i9.P06
36
Discussion and feedback