DETEKSI MOLEKULER GEN ampC PADA ISOLAT KLINIS Klebsiella pneumoniae PENGHASIL ESBL DI RSUP SANGLAH DENPASAR
on
ISSN: 2597-8012
JURNAL MEDIKA UDAYANA, VOL. 8 NO.11,NOPEMBER, 2019
i—∖f^ λ Idirectoryof
OPEN ACCESS L>O∕-∖^J JOURNALS
C^* ≡ι∏tsx
DETEKSI MOLEKULER GEN ampC PADA ISOLAT KLINIS Klebsiella pneumoniae PENGHASIL ESBL DI RSUP SANGLAH DENPASAR
I Gusti Ngurah Krishna Priyaka1*, Ni Made Adi Tarini2, Ni Nengah Dwi Fatmawati2
1Program Studi Pendidikan Dokter Fakultas Kedokteran Universitas Udayana 2Departemen/SMF Mikrobiologi Klnik RSUP Sanglah
*email: krishna.priyaka@yahoo.com
ABSTRAK
Sifat resistensi yang dimiliki oleh bakteri merupakan salah satu masalah pada kasus -kasus Healthcare Associated Infections (HAIs). Klebsiella pneumoniae dapat bersifat resisten terhadap antibiotik jenis cephalosporin akibat diproduksinya enzim AmpC yang dikode oleh gen ampC. Penelitian ini bertujuan untuk mengetahui prevalensi gen ampC dari isolat klinis K. pneumoniae penghasil ESBL di RSUP Sanglah tahun 2013. Penelitian ini menggunakan metode deskriptif cross-sectional. K. pneumoniae diisolasi dari spesimen klinis di Instalasi Mikrobiologi Rumah Sakit Umum Pusat (RSUP) Sanglah selama tahun 2013. Uji fenotif DDST dilakukan pada sampel untuk mendeteksi enzim ESBL. Selanjutnya uji genotif PCR dan sequencing dilakukan untuk mendeteksi gen ampC. Dalam penelitian ini ditemukan hasil PCR 4 sampel positif dengan panjang pita yang tidak sesuai. Salah satu sampel lalu di-sequencing untuk mengidentifikasi gen yang terdeteksi tersebut. Uji sequencing menemukan gen yang terdeteksi adalah fragmen gen yang mengkode enzim alpha—amylase pada K. pneumoniae. Penelitian ini menunjukan gen ampC pada K. pneumoniae belum berhasil teramplifikasi melalui uji genotif PCR. Empat sampel terdeteksi memiliki fragmen gen yang mengkode enzim alpha-amylase dari hasil sequencing dan analisis. Perlu dilakukan penelitian lanjutan dengan menggunakan primer dengan target gen pengkode AmpC lainnya.
Kata Kunci: Klebsiella pneumoniae, HAIs, AmpC, ESBL, PCR
ABSTRACT
Resistance characteristic of bacteria is one of the many problems in Healthcare Associated Infections (HAIs). Klebsiella pneumoniae can become resistant to cephalosporines by producing AmpC enzyme which is decoded by ampC genes. This study aims to determine the prevalence of ampC genes from clinical isolates of ESBL-producing K. pneumoniae in Sanglah General Hospital in 2013. This study used descriptive cross-sectional method. K. pneumoniae were isolated from clinical speciments in Clinical Microbiology Laboratory of Sanglah General Hospital during 2013. DDST phenotypic test then conducted to the samples in order to detect presence of ESBL enzymes. PCR and sequencing were then conducted to detect ampC genes. The results were analyzed descriptively. This study found 4 samples to be positive, but with bands which were inappropriate in size. One of the samples was then sequenced to identify and confirm the detected gene. The sequencing results showed that the detected gene was a fragment of gene that encodes alpha-amylase enzyme in K. pneumoniae. This study showed ampC gene in K. pneumoniae could not be amplified by PCR genotypic test yet. Four samples were detected to have a fragment of gene that encodes alpha-amylase enzyme in K. pneumoniae. Further study using other targeted genes encoding AmpC enzymes is needed to be performed.
Keywords: Klebsiella pneumoniae, HAIs, AmpC, ESBL, PCR
i—∖f^ λ Idirectoryof
OPEN ACCESS L>O∕-∖^J JOURNALS
C^* ≡ι∏tsx
PENDAHULUAN
Kemampuan bakteri untuk mengembangkan sifat resistensi terhadap antibiotik merupakan suatu permaslahan. Sifat resistensi yang dimiliki bakteri ini berdampak signifikan pada penanganan penyakit yang akan menjadi lebih sulit dan lama, sehingga menyebabkan meningkatnya biaya perawatan.1 Sifat resistensi antibiotik ini juga berdampak pada berbagai penyakit infeksi di rumah sakit atau Healthcare Associated Infections (HAIs).1,2
Salah satu bakteri dengan sifat resistensi yang dikhawatirkan adalah Klebsiella pneumoniae. K. pneumoniae merupakan salah satu bakteri gram negatif komponen flora normal di lapisan mukosa saluran pencernaan manusia.1 K. pneumoniae bisa menjadi resisten terhadap antibiotik dengan cara memproduksi enzim β-lactamase, yang akan menghidrolisis cincin β-lactam pada antibiotik – antibiotik jenis β-lactam, seperti cephalosporin dan penicillin. Pada tahun 1995, Bush et al. mengklasifikasikan enzim -enzim β-lactamase menjadi 3 grup. Salah satu diantaranya adalah grup dengan sifat resistensi terhadap antibiotik golongan cephalosporin.3 Antibiotik golongan cephalosporin masih merupakan obat pilihan untuk penanganan penyakit infeksi Klebsiella.3,4
Enzim yang menyebabkan terjadinya resistensi terhadap antibiotik jenis cephalosporin ini adalah enzim β-lactamase golongan AmpC. Enzim AmpC ini dapat berasal dari deregulasi gen kromosomal ampC atau didapatkan dari gen yang ditransfer antar populasi bakteri melalui plasmid yang sering disebu t dengan plasmid-mediated AmpC β-lactamases. K. pneumoniae merupakan salah satu bakteri yang mendapatkan gen ampC tersebut melalui transfer gen antar plasmid.4
Enzim β-lactamase jenis AmpC ini juga dapat diproduksi bersamaan dengan enzim β-lactamase jenis Extended-Spectrum-β-Lactamase (ESBL) pada satu organisme, tapi pada umumnya efek dari AmpC β-lactamase ini akan menutupi efek dari ESBL tersebut, akibatnya hasil pada tes identifikasi β-lactamase nanti akan disalahinterpretasikan menjadi ESBL negatif. Hal ini akan membuat identifikasi dari β-lactamase secara fenotipik menjadi sulit, sehingga identifikasi dan isolasi dengan menggunakan teknik Polymerase Chain Reaction atau PCR masih merupakan pilihan.3,4
Perlu dilakukan penelitian untuk bisa lebih memahami mekanisme resisten yang melibatkan gen ampC tersebut untuk kepentingan penatalaksanaan terapi nantinya pada penyakit infeksi K. pneumoniae. Membedakan antara organisme – organisme yang mengekspresikan
ESBL dari organisme – organisme yang mengekspresikan plasmid-mediated AmpC β-lactamase juga diperlukan untuk kepentingan surveilans, epidemiologi, dan kontrol infeksi di rumah sakit, ditambah lagi dengan kemampuan gen ampC ini untuk ditransfer antar populasi bakteri.4 Belum tersedianya data mengenai prevalensi gen ampC pada bakteri K. pneumoniae di RSUP Sanglah, Denpasar membuat peneliti tertarik untuk melakukan penelitian epidemiologi molekuler yang bertujuan untuk mendeteksi dan mengidentifikasi gen ampC bakteri K. pneumoniae penghasil ESBL dari isolat klinik kasus - kasus infeksi nosokomial di RSUP Sanglah, Denpasar.
BAHAN DAN METODE
Penelitian ini menggunakan rancangan penelitian potong lintang (cross-sectional). Penelitian dilakukan selama 10 (sepuluh) bulan dimulai pada bulan Februari 2015 - November 2015 di Instalasi Mikrobiologi Klinik RSUP Sanglah dan Laboratorium Mikrobiologi FK Unud.
Sampel penelitian adalah semua isolat klinis K. pneumoniae yang terisolasi periode 2013, dan menunjukkan hasil positif ESBL yang diuji dengan metode Double Disk Synergy Test (DDST). Sampel ditentukan secara convenient purposive sampling, dan ditemukan 5 isolat dengan hasil ESBL positif.
Kelima isolat tersebut lalu diisolasi menggunakan High Pure Purification Kit (Roche®), lalu dilakukan PCR Go Green Taq (Promega®) untuk mendeteksi gen resistensi ampC. Primer yang digunakan adalah blaFOX dengan panjang 189 base pair, dengan konsentrasi sebanyak 0,4 µl.5 PCR dijalankan sebanyak 35 siklus, dengan suhu annealing optimal pada 58OC. Elektroforesis dilakukan dengan menggunakan gel agarosa 2%, pada tegangan 50 volt.
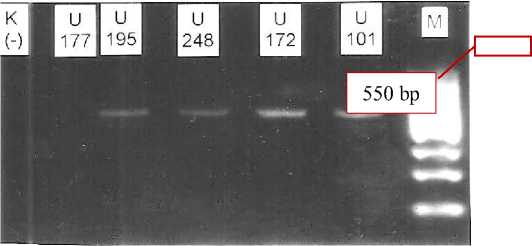
Gambar 1. Produk PCR gen ampC dari lima isolat klinis yang menunjukkan panjang pita sekitar 550 bp.
i—∖f^ λ Idirectoryof OPEN ACCESS L>O∕-V^J JOURNALS
C^* ≡ι∏tsx
Klebsiella pneumoniae strain XH209, complete genome
Sequence ID: qblCP009461.11 Length: 5118878 Number of Matches: 1
Range 1: 5080496 to 5080938 UenBank Graphics
Score Expect Identities Gaps Strand
811 blts(439)0.0 442/443(99%)1/443(0%)Plus/Minus
Features: alpha-amylase
|
Query |
3 |
Tctcggcgagcgctggacggactgga-Gccgggcgcgggacagacctggcacagctttaa |
61 |
|
Iiiiiiiiiiiiiiiiiiiiiiiiii Iiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiii | |||
|
Sbjct |
5080938 |
Tctcggcgagcgctggacggactggaagccgggcgcgggacagacctggcacagctttaa |
5080879 |
|
Query |
62 |
Cgactatatcaacttcagcgacaaagccgggtgggagaaatggtggggtaagaagtggat |
121 |
|
Iiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiii | |||
|
Sbjct |
5080878 |
Cgactatatcaacttcagcgacaaagccgggtgggagaaatggtggggtaagaagtggat |
5080819 |
|
Query |
122 |
Ccgcaccgatatcggcgattacgacaatccgggctatgacgatctcaccatgtcgctggc |
181 |
|
Illllllllllllllllllligilllllllllllllllllllllllllllllllllllll | |||
|
Sbjct |
5080818 |
Ccgcaccgatatcggcgattacgacaatccgggctatgacgatctcaccatgtcgctggc |
5080759 |
|
Query |
182 |
Cttcctgccggacctgaaaaccgaatcgaaggaggtctcaggcctgccgaacttctataa |
241 |
|
Sbjct |
5080758 |
Cttcctgccggacctgaaaaccgaatcgaaggaggtctcaggcctgccgaacttctataa |
5080699 |
|
Query |
242 |
Ccataaaccagacaccgcagcgaaagcgatcccgggctatacgccgcgcgactatctgac |
301 |
|
Iiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiii | |||
|
Sbjct |
5080698 |
Ccataaaccagacaccgcagcgaaagcgatcccgggctatacgccgcgcgactatctgac |
5080639 |
|
Query |
302 |
Acactggctgagccagtgggtacgtgactacggcatcgatggcttccgcgtcgataccgc |
361 |
|
Iiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiii | |||
|
Sbjct |
5080638 |
Acactggctgagccagtgggtacgtgactacggcatcgatggcttccgcgtcgataccgc |
5080579 |
|
Query |
362 |
Caaacatgtggagatggacgcctggcagcagttaaaaacccaggccaccgccgcgctggc |
421 |
|
Iiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiii | |||
|
Sbjct |
5080578 |
Caaacatgtggagatggacgcctggcagcagttaaaaacccaggccaccgccgcgctggc |
5080519 |
|
Query Sbjct |
422 5080518 |
Ggagtggaagaaagccaatccgg 444 Ggagtggaagaaagccaatccgg 5080496 |
Gambar 2. Hasil BLAST dari hasil sequencing yang merupakan fragmen gen pengkode alpha-amylase pada K. pneumoniae.
Tabel 1. Deskripsi primer blaFOX
Primer Sequence
BlaFOX
Forward AAC ATG GGG TAT CAG GGA
(5’-3’) GAT G
Reverse CAA AGC GCG TAA CCG GAT
(3’- 5’) TGG
Sequencing kemudian dilakukan pada salah satu sampel, yaitu u172 untuk mengetahui gen apa yang terdeteksi tersebut. Hasil sequencing dari sampel u172 diedit dan dianalisis dengan software MEGA, lalu dicocokkan dengan database gen pada GenBank dengan menggunakan Basic Local Alignment Search Tool (BLAST) (https://blast.ncbi.nlm.nih.gov/Blast.cgi) dan didapatkan bahwa gen yang terdeteksi adalah gen yang mengkode enzim alpha-amylase pada K. pneumoniae (Gambar 2), yang bukan merupakan target gen pada penelitian ini.
HASIL PENELITIAN
Sampel isolat klinis K. pneumoniae yang digunakan dalam penelitian ini terdiri dari 5 sampel (u101, u172, u177, u195, u248) yang diisolasi dari urin di Instalasi Mikrobiologi Rumah Sakit Umum Pusat (RSUP) Sanglah selama tahun 2013. Uji genotif lalu dilakukan dengan teknik PCR dilakukan untuk mendeteksi gen ampC blaFOX. Panjang pita yang diharapkan dari produk PCR sesuai dengan primer yang digunakan adalah 189 base pair (bp).4 Dari lima sampel didapatkan empat sampel (u101, u172, u195, u248) yang positif namun menunjukan panjang pita sekitar 550 bp, sehingga tidaksesuai dengan panjang yang diharapkan, seperti yang ditunjukkan pada
PEMBAHASAN
Plasmid mediated AmpC β-lactamases dari isolat K. pneumoniae pertama kali dilaporkan tahun 1989 di Seoul, Korea Selatan. Selanjutnya, 29 jenis gen plasmid mediated AmpC β-lactamases sudah dilaporkan di seluruh dunia. Plasmid mediated AmpC β-lactamases merupakan ancaman karena bersifat resisten terhadap β-lactamase inhibitor dan jika terjadi hilangnya outer membrane porin dapat bersifat resisten terhadap karbapenem juga. Mekanisme resisten di K. pneumoniae seperti ini sudah menyebabkan wabah nosokomial di berbagai negara.6
Belum tersedia data mengenai prevalensi gen ampC yang dimiliki K. pneumoniae penghasil enzim ESBL di Indonesia. Sebuah penelitian mengenai tren resistensi antimikrobial di Asia-
i—∖f^ λ Idirectoryof
OPEN ACCESS L>O∕-∖^J JOURNALS
C^* ≡ι∏tsx
Pasifik melaporkan prevalensi gen ampC yang dimiliki K. pneumoniae yang juga penghasil ESBL sebesar 9,6%. Sedangkan di Amerika Serikat, penelitian sebelumnya mendapatkan prevalensi gen ampC sebesar 5,8% dari K. pneumoniae penghasil ESBL.7,8
Penelitian ini belum berhasil mengamplifikasi fragmen gen ampC yang dimiliki K. pneumoniae penghasil enzim ESBL. Deteksi gen ampC menggunakan primer FOXMF dan FOXMR seharusnya mempunyai ukuran amplikon sebesar 189 bp.4 Namun penelitian ini menemukan ukuran amplicon sebesar 550 bp di keempat sampel (u101, u172, u195, u248). Hasil sequencing dan analisis menunjukan bahwa gen yang terdeteksi ini merupakan fragmen dari gen yang mengkode enzim alpha-amylase pada K. pneumoniae. Alpha-amylase adalah sebuah enzim protein yang menghidrolisis polisakarida seperti pati dan glikogen untuk menghasilkan glukosa dan maltosa.9
Penemuan ukuran pita yang tidak sesuai ini bisa disebabkan oleh primer yang kurang spesifik. Kebanyakan primer dirancang secara kualitatif dan belum mempertimbangkan prinsip – prinsip termodinamika dan struktural. Primer yang ideal seharusnya mampu menekan amplifikasi dari gen yang tidak diinginkan.9,10 Peneliti telah melakukan uji konfirmasi apakah primer yang digunakan spesifik untuk gen ampC dengan menggunakan BLAST dan hasil menunjukkan bahwa primer spesifik untuk ampC, namun pada penelitian ini peneliti belum berhasil menemukan pita 189 bp yang merupakan panjang fragmen gen ampC yang dimaksud.
Suasana PCR yang kurang optimal juga bisa menyebabkan didapatkannya ukuran pita yang tidak sesuai. Suasana optimal PCR sangat bergantung pada suhu annealing. Jika suhu annealing terlalu rendah, annealing primer akan menjadi tidak spesifik dan menyebabkan amplifikasi dari segmen DNA yang tidak diinginkan. Suhu annealing biasanya akan berada 3-5oC lebih rendah dari melting temperature primer yang sudah dihitung. Banyak rumus yang bisa digunakan untuk menghitung melting temperature, tapi tidak ada rumus yang benar -benar akurat untuk semua primer.10 Peneliti telah melakukan optimasi beberapa kali dengan mengatur konsentrasi primer dan suhu penempelan primer, namun selalu didapatkan pita sepanjang 550 bp, sehingga langkah selanjutnya dilakukan sequencing untuk memastikan urutan nukleotidanya.
Penemuan ukuran pita yang tidak sesuai ini seharusnya bisa dibandingkan dengan kontrol positif.10 Tapi dalam penelitian ini sulit untuk mendapatkan kontrol positif dari K. pneumoniae
yang memiliki gen ampC, sehingga tidak bisa dilakukan perbandingan. Hal ini menjadi salah satu keterbatasan dalam penelitian ini.
SIMPULAN
Pada penelitian ini belum ditemukan keberadaan gen ampC (blaFOX) dari isolat klinis K. pneumoniae dengan menggunakan uji genotif PCR. Empat sampel terdeteksi memiliki gen alpha-amylase dari hasil sequencing dan analisis.
Perlu dilakukan penelitian lanjutan dengan menggunakan primer dengan target gen pengkode AmpC lainnya untuk mengetahui epidemiologi molekuler, mekanisme resisten dan hubungan masing – masing antara gen resistensi antibiotik lainnya.
UCAPAN TERIMA KASIH
Peneliti ingin mengucapkan terima kasih yang sebesar – besarnya terhadap semua pihak yang sudah membantu jalannya penelitian ini, terutama Bagian/SMF Mikrobiologi FK Unud/RSUP Sanglah, Instalasi Mikrobiologi Klinis RSUP Sanglah, dan Laboratorium Mikrobiologi FK Unud. Peneliti tidak memiliki konflik kepentingan dalam penelitian ini.
DAFTAR PUSTAKA
-
1. Centers for Disease Control and Prevention (CDC). 2013. Antibiotic Resistance Threats in the United States. Atlanta.
-
2. World Health Organization (WHO). 2014. Antimicrobial Resistance Global Report on Surveillance. Geneva.
-
3. Dhillon, R.H.P., Clark, J. 2012. ESBLs: A Clear and Present Danger?. Critical Care Research and Practice, 2012(1): 111
-
4. Pérez-Pérez, F.J., Hanson, N.D. 2002. Detection of Plasmid-Mediated AmpC β -Lactamase Genes in Clinical Isolates by Using Multiplex PCR. J Clin Microbiol. 40(6): 2153-2162.
-
5. Yan, J., Hsueh, P., Lu, J., Chang, F., Shyr, J., Wan, J., dkk. 2006. Extended-Spectrum β-Lactamases and Plasmid-Mediated AmpC Enzymes among
Clinical Isolates of Escherichia coli and Klebsiella pneumoniae from Seven
Medical Centers in Taiwan. Antimicrob Agents Chemother. 50(5): 1861-1864.
-
6. Mohammudha, P.R., Harish, B.N., Parija, S.C. 2012. Molecular description of plasmid-mediated AmpC β-lactamases
i—∖f^ λ Idirectoryof
OPEN ACCESS L>O∕-∖^J JOURNALS
C^* ≡ι∏tsx
among nosocomial isolates of Escherichia coli & Klebsiella pneumoniae from six different hospitals in India. Indian J Med Res 135, 2012(1): 114-119.
-
7. Sheng, W.H., Badal, R.E., Hsueh, P.R. 2013. Distribution of Extended-Spectrum β-Lactamases, AmpC β-Lactamases, and Carbapenemases among
Enterobacteriaceae Isolates Causing Intra-Abdominal Infections in the Asia-Pacific Region: Results of the Study for Monitoring Antimicrobial Resistance Trends (SMART). Antimicrobial Agents and Chemotherapy. 57(7): 2981-2988.
-
8. Alvarez, M., Tran, J.H., Chow, N., Jacoby, G.A. 2004. Epidemiology of Conjugative Plasmid-Mediated AmpC β-Lactamases in the United States. Antimicrobial agents and Chemotherapy. (48)2: 533-537.
-
9. Tilley, L.P., Smith Jr., F.W.K., Allen, D. 2003. Stedman’s Medical Dictionary. 27th edition. Baltimore: Lippincott
Williams & Wilkins. p. 65.
-
10. Sambrook, J.F., Russell, D.W. 2001. Molecular Cloning: A Laboratory
Manual,Vol. 2. Third edition. Cold Spring Harbor Laboratory Press. p. 8.88.24.
Discussion and feedback