DESAIN DNA PELACAK SECARA IN SILICO SEBAGAI PENDETEKSI MUTASI PADA GEN embB Mycobacterium tuberculosis
on
Cakra Kimia (Indonesian E-Journal of Applied Chemistry)
Volume 8 Nomor 1, Mei 2020
I Ca^ra I ∣ eKimia ∣
DESAIN DNA PELACAK SECARA IN SILICO SEBAGAI PENDETEKSI MUTASI PADA GEN embB Mycobacterium tuberculosis
Ade Ari Sundari1, Ni Putu Monica Rosdiana Dewi Paramitha1, Sagung Chandra Yowani2 dan Putu Sanna Yustiantara2
-
1 Program Studi Farmasi, FMIPA, Universitas Udayana, Bukit Jimbaran, Bali-Indonesia, 80361 arisundari2@gmail.com monica.rosdianadp@gmail.com
-
2 Kelompok Studi MDR-TB & XDR-TB, FMIPA, Universitas Udayana, Bukit Jimbaran, Bali-Indonesia, 80361 cyowani@gmail.com pyustiantara@gmail.com
ABSTRAK: Terapi lini pertama Tuberkulosis (TB) selalu menggunakan terapi kombinasi yaitu rifampisin, isoniazid, etambutol, dan pirazinamid. Penggunaan etambutol dalam terapi kombinasi TB digunakan untuk mencegah terjadinya resistensi terhadap obat lain namun tingkat resistensi etambutol secara bertahap meningkat. Mutasi pada operon embCAB bertanggung jawab pada resistensi etambutol dengan prevalensi tertinggi terjadi pada kodon 306 gen embB. Mutasi pada gen embB kodon 306 juga dikaitkan dengan adanya kecenderungan resistensi akibat peningkatan konsumsi obat sehingga dijadikan sebagai kandidat potential marker untuk board drug resistance, khususnya untuk MDR-TB. Penelitian ini akan mendesain urutan nukleotida TaqMan probe untuk mendeteksi mutasi M306I menggunakan program Clone Manager Suite 9.2. Hasil rancangan probe DNA kemudian dianalisis berdasarkan kriteria probe secara umum dan berdasarkan kriteria pelabelan TaqMan probe. Rancangan probe DNA mutan menggunakan program menghasilkan 10 probe yang memenuhi kriteria probe secara umum untuk mutasi M306I pada gen embB. Berdasarkan analisa pelabelan TaqMan probe, diperoleh 7 probe (E306MI4, E306MI5, E306MI6, E306MI7, E306MI9, E306MI10, dan E306MI13) untuk deteksi mutasi M306I pada gen embB. Hasil rancangan probe mutan yang telah memenuhi kriteria pelabelan TaqMan probe dapat digunakan untuk mendeteksi adanya mutasi kodon 306 gen embB Mycobacterium tuberculosis. Hasil perancangan TaqMan probe perlu diuji secara eksperimental untuk membuktikan efisiensi kerja dari probe tersebut agar dapat digunakan pada metode Real-Time PCR.
Kata kunci: Tuberkulosis, gen embB, in silico, TaqMan probe, Real-Time PCR.
ABSTRACT: First-line Tuberculosis (TB) therapy always uses combination therapy, such as rifampicin, isoniazid, ethambutol, and pyrazinamide. Ethambutol was effective for preventing treatment failures caused by Mycobacterium tuberculosis isolates resistant to other anti-TB drugs however, the resistance rate of ethambutol has gradually increased. Mutations in the embCAB operon have been identified to confer resistance to ethambutol, with embB codon 306 being the most frequently affected. embB306 mutations are associated a tendency for resistance due to increasing numbers of antibiotics consumption so it may be a potential marker for broad drug resistance, especially for MDR-TB. This research design the TaqMan probe nucleotide sequence for the M306I spesific mutation using the Clone Manager Suite 9.2 program. The results of the DNA probe design were then analyzed based on probe criteria in general and based on the TaqMan probe labeling criteria. The mutant DNA probes design using the program produced 10 probes thats have met the general probe criteria for the M306I
mutation in the embB gene. Based on the TaqMan probe labeling analysis, theres 7 probes (E306MI4, E306MI5, E306MI6, E306MI7, E306MI9, E306MI10 and E306MI13) for the detection of M306I mutations in the embB gene. The results of the mutant probe design that has met the TaqMan labeling criteria can be used to detect mutations in M. tuberculosis embB gene codon 306. The results of the TaqMan probe need to be tested experimentally to prove the working efficiency of the probe so that it can be used in the Real-Time PCR method.
Keywords: Tuberculosis, embB gene, in silico, TaqMan probe, Real-Time PCR
Drug-resistant Tuberclosis (TB), khususnya resistensi obat anti tuberkulosis (OAT) lini pertama merupakan salah satu hambatan utama dalam pengendalian TB. Data World Health Organization (WHO) menunjukkan bahwa pada 19% kasus baru TB dan 43% kasus yang sudah ditangani, mengalami resistensi terhadap OAT lini pertama [1]. Pengobatan TB selalu menggunakan kombinasi obat untuk efektivitas terapi. Obat-obat lini pertama yang digunakan adalah isoniazid, rifampisin, etambutol, dan pirazinamid. Etambutol merupakan obat pilihan yang digunakan untuk mencegah terjadinya resistensi terhadap obat lain dalam terapi kombinasi. Awalnya, penggunaan etambutol efektif untuk mencegah kegagalan pengobatan yang disebabkan isolat M. tuberculosis yang resisten terhadap OAT lainnya. Sejalan dengan kondisi tersebut, tingkat resistensi etambutol secara bertahap meningkat dan hampir 50% pada pasien TB retreated [2]. Laporan terakhir WHO tentang resistensi OAT pada tahun 2008 menyatakan bahwa resistensi global terhadap etambutol mencapai 2,5% di antara kasus TB baru dan 10,3% diantara kasus yang telah diobati sebelumnya [3].
Etambutol bekerja melalui penghambatan arabinosil transferase yang dikode oleh operon embCAB serta penghambatan sintesis arabinogalactan dan lipoarabinomannan pada dinding sel M. tuberculosis. Operon tersebut terdiri dari tiga gen yang berdekatan yang terdiri dari embC, embA, dan embB [4]. Mutasi pada gen embB, terutama pada daerah
Ethambutol Resistance Determining Region (ERDR), bertanggung jawab pada resistensi etambutol [5]. Prevalensi tertinggi mutasi gen embB terjadi pada kodon 306 sebesar 30-69%, sehingga identifikasi mutasi khususnya pada gen embB kodon 306, dianggap mewakili metode skrining cepat untuk deteksi resistensi etambutol pada isolat klinis [6]. Mutasi pada kodon 306 menyebabkan perubahan asam amino seperti perubahan metionin menjadi valin (M306V), metionin menjadi leusin (M306L), dan metionin menjadi isoleusin (M306I). Perubahan asam amino yang paling sering terjadi adalah metionin menjadi valin [7, 8]
Beberapa penelitian menyatakan bahwa mutasi pada embB306 tidak selalu berhubungan dengan resistensi etambutol tetapi dikaitkan dengan adanya kecenderungan resistensi akibat peningkatan konsumsi obat-obatan. Pada penelitian Cordoba et al. [8] dinyatakan bahwa isolat yang membawa mutasi pada embB306 juga memiliki resistensi terhadap isoniazid (95%), etambutol (91%), rifampisin (86%), MDR-TB (86%), dan etambutol + isoniazid + rifampisin (82%). Hasil penelitian Li et al. [4] juga menyatakan bahwa mutasi embB306 dapat ditemukan pada 30,4% isolat resisten isoniazid, 37,0% isolat resisten rifampisin, 56,5% isolat resisten etambutol, dan 44,0% isolat MDR-TB. Hubungan yang signifikan antara mutasi embB306 dan resistensi terhadap etambutol, isoniazid, rifampisin, dan MDR-TB, menunjukkan bahwa mutasi di embB306 dapat dijadikan sebagai kandidat potential marker untuk board drug resistance, khususnya untuk MDR-TB
[4,8,9]. Dengan adanya fenomena tersebut, maka deteksi adanya mutasi pada gen embB pada kodon 306 dijadikan fokus dalam penelitian ini.
Deteksi M. tuberculosis yang mengalami mutasi dapat dilakukan dengan metode Real-Time Polymerase Chain Reaction (RT-PCR) menggunakan probe atau pelacak. Probe merupakan molekul asam nukleat berupa DNA beruntai tunggal dengan afinitas yang kuat dengan target spesifik. Urutan probe DNA harus spesifik terhadap urutan DNA target sehingga perancangan probe DNA diperlukan untuk menghindari hibridisasi silang pada urutan yang memiliki banyak persamaan [10]. Sistem deteksi probe DNA yang digunakan untuk RT-PCR terdapat beberapa macam, salah satunya adalah TaqMan probe. TaqMan probe merupakan jenis probe hidrolisis yang bekerja dengan melibatkan aktivitas eksonuklease dari Taq DNA polimerase [11]. TaqMan probe mudah didesain dan disintesis, tetapi jika tidak dirancang dengan baik maka dapat terbentuk struktur sekunder seperti dimer, hairpins, runs, dan repeat selama proses RT-PCR [12].
Penelitian ini akan berfokus pada perancangan DNA pelacak yang digunakan untuk mendeteksi mutasi dengan prevalensi tertinggi gen embB kodon 306 M. tuberculosis dengan metode RT-PCR. Gen tersebut dipilih untuk mendeteksi adanya board drug resistance lebih awal, khususnya untuk MDR-TB.
Bahan yang digunakan dalam penelitian ini yaitu urutan nukleotida gen embB (accession number: NC_000962) M. tuberculosis H37Rv dari database URL://www.ncbi.nlm.nih.gov. Satu pasang primer dengan urutan primer forward 5’ ACCTTCGCCAACTTCGTC 3’ dan primer reverse dengan urutan 5’ CGCCAGCAGGTTGTAATACCAG 3’. Primer tersebut merupakan primer terbaik
yang di desain secara in silico yang memiliki suhu annealing 58°C. Penelitian ini akan menggunakan panjang dan Tm primer sebagai acuan dalam mendesain probe DNA.
Peralatan
Peralatan yang digunakan diantaranya komputer dengan spesifikasi Windows 10, jaringan internet, dan program Clone Manager Suite 9.2 untuk mendesain serta menganalisis hasil rancangan probe DNA.
-
2.2 METODE
Pemilihan Sekuens Target Rancangan Probe DNA
Perancangan probe DNA
menggunakan keseluruhan urutan
nukleotida gen embB yang terdiri dari 3297 pb. Pemilihan kodon 306 gen embB sebagai target perancangan probe DNA didasarkan pada frekuensi mutasi yang paling sering terjadi dan berkaitan dengan adanya board drug resistance khususnya untuk MDR-TB. Perancangan probe DNA akan mengacu pada perubahan asam amino pada kodon 306 gen embB yaitu metionin menjadi isoleusin (M306I) dengan perubahan nukleotida ATG menjadi ATA. Gambar 1. menunjukkan posisi kodon 306 gen embB ditandai dengan warna kuning.

Gambar 1. Posisi kodon 306 gen embB sebagai kodon target perancangan probe DNA
Perancangan Probe DNA
Perancangan probe DNA dilakukan secara in silico dengan mengunduh urutan nukleotida gen embB (accession number: NC_000962) M. tuberculosis H37Rv dalam
format data FASTA dan disimpan dalam notepad sebagai template. Perancangan probe mutan dengan program Clone Manager Suite 9.2 dimulai dengan perancangan probe wild-type, yang dilakukan dengan diklik ‘Primer’ pada program lalu pilih ‘Design’ kemudian dimasukkan data template gen embB dan klik ‘OK’. Kemudian pada ‘Primer Type’ pilih ‘Probe’ dan pada kolom ‘Design Detail’ dimasukkan panjang probe DNA yang divariasikan dari 18-30 basa pada bagian ‘length’ dan diatur daerah target yang diinginkan, lalu klik ‘OK’. Selanjutnya program akan memberikan beberapa data sekuens probe DNA dan diklik ‘Primer report’ untuk menampilkan urutan nukleotida, panjang, komposisi dan kriteria dari probe. Urutan nukleotida yang mencakup posisi mutasi diskrining. Desain probe wild-type selanjutnya diubah menjadi desain probe mutan melalui penggantian basa nukleotida pada menu direct entry. Penggantian dilakukan agar diperoleh desain probe mutan yang spesifik terhadap perubahan nukleotida pada kodon kodon 306 gen embB.
-
3. HASIL DAN PEMBAHASAN Pemilihan Sekuens Target Rancangan Probe DNA
Perancangan probe DNA untuk gen embB dilakukan pada daerah ERDR gen embB M. tuberculosis. Mutasi pada gen embB yang paling umum terdeteksi terjadi pada kodon 306, 406, dan 497 [13, 14, 15]. Prevalensi tertinggi mutasi gen embB terjadi pada kodon 306 sebesar 30-69% [6]. Mutasi pada kodon 306 menyebabkan perubahan asam amino seperti perubahan metionin menjadi valin (M306V), metionin menjadi leusin (M306L), dan metionin menjadi isoleusin (M306I). Perubahan asam amino yang paling sering terjadi adalah metionin menjadi valin. Penelitian ini akan merancang probe DNA mutan untuk mendeteksi mutasi M306I. Perancangan probe DNA mutan dilakukan menggunakan urutan nukleotida wild-type dari gen embB (accession number:
NC_000962) M. tuberculosis H37Rv yang diperoleh dari database National Center for Biotechnology Information (NCBI).
Hasil Perancangan Probe DNA
Penelitian ini menggunakan data panjang dan Tm dari primer yang dijadikan sebagai acuan untuk menentukan kriteria dari probe mutan yang akan dirancang. Perancangan probe secara in silico merupakan suatu teknik perancangan urutan nukleotida probe yang cepat dan sederhana. Suatu probe dapat dibuat dengan memasukkan urutan nukleotida lengkap. Program secara otomatis memberikan informasi mengenai kompatibilitas probe untuk target yang diinginkan, serta disajikan dalam bentuk daftar probe terpilih. Kriteria dari masing-masing rancangan probe juga dapat diperoleh lebih cepat karena secara langsung ditampilkan oleh program sesuai urutan nukleotidanya.
Probe mutan yang dirancang merupakan TaqMan probe. Jenis probe ini memiliki desain yang sederhana dan fluoresensinya dapat ditentukan pada akhir fase ekstensi [12]. Desain TaqMan probe berupa susunan oligonukleotida linear yang dilabel reporter pada ujung 5’ dan quencher pada ujung 3’ [16]. TaqMan probe bersifat spesifik sehingga dapat digunakan untuk mendeteksi satu perubahan basa pada urutan nukleotida target [17]. Perancangan probe mutan dengan program Clone Manager Suite 9.2 dimulai dengan perancangan probe wild-type. Hasil desain probe wild-type yang diperoleh tidak khusus mencakup daerah target mutasi yang diinginkan. Oleh karena itu, seleksi berdasarkan cakupan daerah target dilakukan untuk memperoleh desain probe wild-type yang mencakup rentang daerah mutasi pada posisi 918 bp dari kodon start. Desain probe wild-type yang telah terpilih selanjutnya diubah menjadi desain probe mutan melalui penggantian basa nukleotida pada menu direct entry. Penggantian dilakukan agar diperoleh desain probe
mutan yang spesifik terhadap perubahan nukleotida pada kodon 306 gen embB.
Hasil Analisis Rancangan DNA Probe
Analisis perancangan probe DNA dilakukan melalui 2 tahap yaitu analisis tahap awal sesuai dengan kriteria probe secara umum dan analisis tahap akhir sesuai dengan kriteria pelabelan TaqMan probe. Analisis tahap awal dilakukan berdasarkan kriteria probe secara umum yang meliputi panjang nukleotida, kandungan %GC, Tm, runs, repeats, dimer, dan hairpin. Desain yang memenuhi semua kriteria umum probe akan ditampilkan dengan tanda centang (√) pada program, sedangkan probe yang tidak memenuhi kriteria akan ditampilkan dengan tanda peringatan (!).
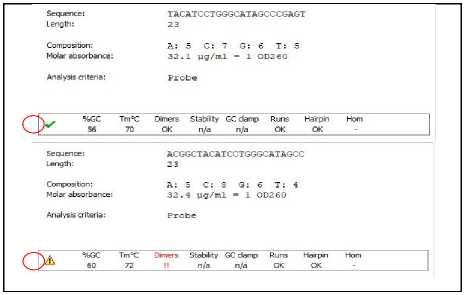
Gambar 2. Contoh tampilan hasil analisis probe pada program Clone Manager Suite
9.2
Beberapa kriteria analisis tahap awal probe didasarkan pada kriteria primer yang digunakan [18]. Kriteria primer yang dijadikan acuan pada penelitian ini adalah panjang dan nilai Tm primer untuk menentukan panjang dan nilai Tm probe. Hal ini perlu dilakukan untuk mencegah penempelan tidak spesifik dari primer atau probe karena berada dalam satu reaksi yang sama selama PCR [19]. Kriteria analisis tahap awal meliputi, panjang probe 18-30 basa, kandungan GC 35-65%, Tm probe 5-10ºC lebih tinggi dari Tm primer, dimer kurang dari 5 basa, tidak terbentuk hairpin, serta jumlah runs dan repeats ≤ 4.
Tabel 1. Hasil Perancangan Probe DNA Mutan untuk Deteksi Mutasi M306I Gen embB
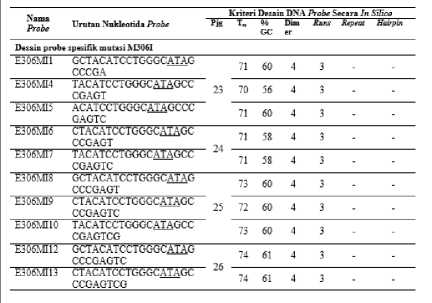
Keterangan: Nama probe misalnya E306MI1: E = menunjukkan gen embB M. tuberculosis; 306 = menunjukkan kodon target; M = menunjukkan jenis rancangan probe mutan; I = menunjukkan asam amino yang terbentuk akibat mutasi; 1 = nomor desain probe.
Probe mutan hasil desain memiliki variasi panjang 23-26 basa. Probe dirancang melebihi panjang primer yang digunakan. Hal ini dimaksudkan untuk memperoleh Tm probe yang lebih tinggi dibandingkan Tm primer untuk mencegah degradasi probe yang tidak efisien. Panjang probe yang diperoleh sudah sesuai dengan kriteria panjang probe yang baik yaitu berkisar 18-30 basa. Probe DNA yang terlalu panjang dapat menghasilkan waktu hibridisasi yang lebih lama dan kurang spesifik untuk membedakan mismatch. Hal ini dapat menyebabkan hasil positif palsu, khususnya ketika perbedaan di antara 2 atau lebih probe yang digunakan hanya terletak pada nukleotida tunggal [17]. Sedangkan probe yang terlalu pendek akan memiliki waktu annealing yang singkat, tetapi dapat menghasilkan spesifisitas yang rendah [20].
Kriteria kedua yaitu %GC sebesar 3565%. Probe mutan hasil desain memiki kandungan %GC dengan 56-65% sehingga masih memenuhi kriteria probe yang baik. Nilai %GC berhubungan dengan Tm dimana semakin tinggi GC maka nilai Tm juga semakin tinggi. Hal tersebut disebabkan oleh pasangan basa G-C dihubungkan oleh ikatan hidrogen rangkap 3 dan pasangan basa A-T yang
dihubungkan oleh ikatan hidrogen rangkap 2 [18].
Semua desain probe mutan kemudian diseleksi berdasarkan nilai Tm. Kriteria dari probe yang baik yaitu memiliki Tm 5-10°C lebih tinggi dari Tm primer [19].Tm probe mutan untuk kodon 306 gen embB memiliki rentang Tm 72-74°C dengan Tm primer yang digunakan yaitu 62°C dan 65°C. Tm probe yang lebih tinggi akan menyebabkan probe tetap terhibridisasi dengan DNA target sebelum proses pemanjangan primer. Kompleks hibridisasi yang stabil akan menghasilkan aktivitas eksonuklease maksimum selama proses PCR. Apabila tidak terjadi proses degradasi probe yang efisiensi, maka amplifikasi dalam PCR dapat berlangsung, tetapi amplikon tidak akan terdeteksi [21].
Probe mutan selanjutnya dianalisis berdasarkan jumlah runs, repeats, dan hairpins. Jumlah runs dari seluruh desain probe DNA mutan yang diperoleh yaitu tidak lebih dari 4 basa dan tidak memiliki repeats dan hairpins. Jumlah runs dan repeats yang dapat diterima sebagai probe yang baik yaitu tidak lebih dari 4. Probe DNA sebaiknya harus menghindari runs nukleotida yang sama, khususnya runs basa G sebanyak 4 atau lebih untuk mencegah terbentuknya struktur sekunder [11]. Probe DNA juga tidak boleh memiliki hairpins karena dapat menghambat proses hibridisasi probe pada DNA target [16].
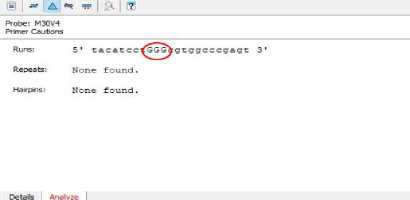
Gambar 3. Tampilan hasil analisis runs, repeats, dan hairpins pada program Clone Manager Suite 9.2.
Analisis struktur sekunder selain hairpins adalah dimer. Adanya dimer pada probe dapat mengurangi efisiensi hibridisasi sehingga menimbulkan
penurunan sensitivitas deteksi [18]. Dimer ditemukan sebanyak 4 basa pada seluruh sekuens probe. Dimer yang terbentuk merupakan dimers any yaitu dimer yang terbentuk pada daerah selain 3’. Pada program Clone Manager Suite 9.2 kriteria dimers any yang diperbolehkan yaitu tidak melebihi 5 basa homolog, sehingga hasil perancangan probe dengan 4 dimers any masih memenuhi kriteria probe yang baik. Dimer dibentuk oleh interaksi intermolekuler antara dua probe [22]. Contoh tampilan dimer dapat dilihat pada Gambar 4.
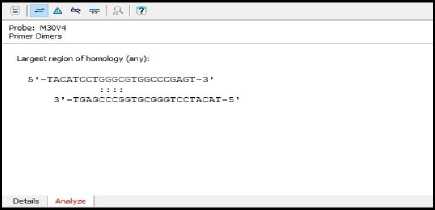
Gambar 4. Tampilan hasil analisis dimer pada program Clone Manager Suite 9.2.
Desain-desain probe mutan terpilih pada analisis tahap awal kemudian dianalisis berdasarkan kriteria pelabelan TaqMan probe. TaqMan probe merupakan salah satu jenis probe hidrolisis yang bekerja melibatkan aktivitas eksonuklease 5’→3’ Taq DNA polymerase. Aktivitas 5’ eksonuklease dari enzim polimerase akan memecah probe sehingga FRET tidak terjadi lagi dan dihasilkan sinyal fluoresensi. Fluoresensi akan semakin meningkat sebanding dengan peningkatan pemecahan probe [11]. TaqMan probe diberi label dengan dua pewarna fluoresens yang memancarkan pada panjang gelombang yang berbeda. Label TaqMan probe terdiri dari reporter yang melekat pada ujung 5’ dan quencher pada ujung 3’ [23]. Kriteria pertama dari pelabelan TaqMan probe yaitu tidak terdapat basa Guanin (G) pada posisi pertama dan kedua dari ujung 5’ [11]. Basa G tidak dianjurkan berada pada ujung 5’ karena basa G merupakan quencher yang dapat
memadamkan fluoresens dari reporter [24]. Kemampuan basa G mengabsorbsi fluoresensi terjadi melalui tranfer elektron yang diinduksi cahaya [25]. Berdasarkan
kriteria tersebut, diperoleh 7 probe yaitu probe E306MI4, E306MI5, E306MI6,
E306MI7, E306MI9, E306MI10, dan
E306MI13 untuk mutasi M306I gen embB. Kriteria kedua yaitu probe mengandung jumlah basa sitosin (C) lebih banyak atau sama dengan basa G [11]. Jumlah dan posisi basa G sangat perlu dipertimbangkan agar diperoleh efisiensi sinyal deteksi yang optimal. Basa G dapat mengganggu FRET dengan kemampuan donor elektron yang kuat. Struktur basa G mengandung paling banyak atom dengan sifat elektronegatif yang dapat menyumbangkan pasangan elektron bebas. Hal ini menyebabkan kemampuan donor elektron basa G paling besar dibandingkan basa nukleotida lain [26]. Setelah diseleksi kembali, probe yang memenuhi kriteria pelabelan TaqMan probe yaitu probe E306MI4, E306MI5,
E306MI6, E306MI7, E306MI9, E306MI10, dan E306MI13 untuk gen embB. Untuk mendeteksi mutasi M306I
direkomendasikan menggunakan probe E306MI4 karena memiliki panjang, %GC, dan Tm yang lebih rendah serta tidak mengandung repeats, sehingga probe mutan dapat terhibridisasi dengan lebih baik.
Probe yang telah memenuhi kriteria pelabelan TaqMan probe dapat ditempelkan label yang terdiri dari reporter dan quencher. Label yang biasanya digunakan untuk TaqMan probe adalah FAM sebagai reporter dan TAMRA sebagai quencher [18, 19]. Reporter FAM merupakan label pewarna donor dengan signal flouresen kuat yang memonitoring hasil amplifikasi di setiap siklus PCR dengan berikatan spesifik dengan probe DNA [18]. Sedangkan, TAMRA dipilih sebagai quencher karena label ini kompatibel dengan FAM dan memiliki panjang gelombang absorbsi pada 558 nm yang tumpang tindih dengan panjang
gelombang emisi FAM pada 517 nm sehingga FRET tidak lagi terjadi [19, 27].
E306Miι → 5’ -Qctacatcctgggcatagcccga - 3,
IE306MI4 → 5’ - TACATCCTgJGGC ATAGCCCGAGT - 3’
Keterangan:
O : posisi basa G (Guanin)
Gambar 5. Posisi basa G pada probe E306MI1 tidak memenuhi kriteria dan
E306MI4 memenuhi kriteria pelabelan TaqMan probe
Label reporter dan quencher ditempelkan pada ujung berlawanan pada nukleotida probe mutan. Reporter direkomendasikan agar dilekatkan pada basa C atau T. Sedangkan quencher ditempelkan pada ujung 3’ atau di posisi sentral dari oligonukleotida dan dapat ditempel pada semua nukleotida [21, 28]. Pada pemasangan label, jarak optimal antara dua label perlu dipertimbangkan untuk memperoleh efisiensi pemadaman label reporter yang baik. Quencher sebaiknya memiliki jarak 3-30 nukleotida dari reporter. Quencher pada jarak 2 basa dari reporter akan mengakibatkan enzim Taq polymerase tidak mampu melakukan aktivitas eksonuklease yang berperan dalam degradasi probe untuk menghasilkan sinyal fluoresensi. Apabila jarak antara repoter dengan quencher terlalu jauh maka quencher tidak dapat dengan cukup menerima tranfer energi dari reporter sebelum reporter terlepas dari sekuens, sehingga proses deteksi tidak dapat berlangsung dengan baik [28, 29].

φ---k GAC GAC GGC TAC ATC CTG GGC ATG GCC CGA GTC GCC
©----k 299 3« 301 302 303 304 305 306 307 308 309 310
Keteranaan : 1 . Kodon sen embB M tuberculosis
2 : Nukleotada Wildtype gen embB M. tuberculosis
3 : Nukleotida probe mutan E306MI4
Gambar 6. Pelabelan desain probe mutan E306MI4 yang spesifik terhadap perubahan A → G pada kodon 306 pada gen embB.
Pemasangan label pada desain probe mutan E306MI4, label reporter FAM ditempelkan pada basa C di ujung 5’, sedangkan label quencher TAMRA ditempelkan pada basa T di ujung 3’.
Desain TaqMan probe yang dihasilkan dapat digunakan secara spesifik untuk mendeteksi mutasi M306I pada gen embB. Hasil perancangan TaqMan probe secara in silico perlu dilakukan pengujian lebih lanjut secara eksperimental di laboratorium untuk membuktikan bahwa hasil rancangan TaqMan probe mampu mendeteksi adanya mutasi pada kodon 306 gen embB M. tuberculosis dengan metode RT-PCR.
Terdapat 7 probe (E306MI4, E306MI5, E306MI6, E306MI7, E306MI9, E306MI10, dan E306MI13) untuk deteksi spesifik mutasi M306I pada gen embB. Probe E306MI4 dengan urutan 5’-FAM-TAC ATC CTG GGC ATA GCC CGA GT-TAMRA-3’ direkomendasikan untuk digunakan dalam deteksi mutasi M306I pada gen embB untuk memperoleh hibridisasi probe yang lebih baik.
-
[1] World Health Organization (WHO).
2018. Global Tuberculosis Report, World Health Organization, Geneva.
-
[2] Cheng, S., Cui, Z., Li, Y., and Hu, Z.
2014. Diagnostic Accuracy of a Molecular Drug Susceptibility Testing Method for the Antituberculosis Drug Ethambutol: a Systematic Review and Meta-Analysis, Journal of Clinical Microbiology, 52: 2913-2924.
-
[3] Moure, R., Español, M., Tudó, G., Vicente, E., Coll, P., Martin, J.G., Mick, V., Salvadó, M., and Alcaide, F. 2013. Characterization of the embB gene in Mycobacterium tuberculosis isolates from Barcelona and rapid detection of main mutations related to ethambutol resistance using a low-
density DNA array. Journal of Antimicrobial Chemotherapy, 69 : 947954
-
[4] Li, Y., Wang, Y., Zhang, Z., Gao, H., Wang, H., Cao, J., Zhang, S., Liu, Y., Lu, J., Xu, Z., and Daia, E., 2016. Association between embB Codon 306 Mutations, Phenotypic Resistance
Profiles, and Genotypic
Characterization in Clinical
Mycobacterium tuberculosis Isolates from Hebei, China, Antimicrobial Agents and Chemotheramy, 60: 72957301.
-
[5] Mohammadi, B., Mohajeri, P., Rouhi, S., and Ramazanzadeh, R., 2018. The relationship between embB306 and embB406 mutations and ethambutol resistant in Mycobacterium
tuberculosis isolated from patiens in west of Iran, Medical Journal of the Islamic Republic of Iran, 32: 1-4.
-
[6] Rezaei, F.M., Haeili, P., Mohajeri, A.H., Shahraki, Fooladi, A.I., Zahednamazi, F., Feizabadi, M., 2016, Frequency of mutational changes in the embB among the ethambutol resistant strains of Mycobacterium tuberculosis in Iran, The Journal of Infection in Developing Countries, 10: 363-368.
-
[7] Bakula, Z., Napiorkowska, A., Bielecki, J., Kopec, E.A., Zwolska, Z., and Jagielski, T., 2013. Mutations in the embB Gene and Their Association with Ethambutol Resistance in Multidrug-Resistant Mycobacterium tuberculosis Clinical Isolates from Poland, BioMed Research
International.
-
[8] Cordoba, B.C., Eusebio, D.M.J., Velasco, R.A., Salazar, R.M., Laborin, R.L., and Cuevas, R.Z., 2015.
Mutation at embB Codon 306, a Potential Marker for the Identification of Multidrug Resistance Associated with Ethambutol in Mycobacterium tuberculosis, Antimicrobial Agents and Chemotherapy, 59: 5455-5462.
-
[9] Zhang, H., Chen, X., Wang, Z., Ren, Z., Wu, J., Sun, H. and Bai, X., 2015.
Pyrosequencing analysis for mutations in embB codon306 among clinical Mycobacterium tuberculosis isolates from Qingdao, China. Int J Clin Exp Med. 8 : 11276-11282.
-
[10] Chou, C., Chen, C.H., Lee, T., and Peck, K., 2004. Optimization of probe length and the number of probes per gene for optimal microarray analysis of gene expression, Nucleic Acids Research, 32: 1-8
-
[11] McPherson, M and Moller, S., 2006. PCR Edisi 2, Taylor & Francis Group, New York.
-
[12] Navarro, E., Heras, G.S., Castano and Solera, J., 2015. Real-Time PCR
Detection Chemistry, Clinica Chimica Acta, 439: 231-250.
-
[13] Zhao, L., Sun, Q., Liu, H., Xiao, T., Zhao, X., Li, G., Jiang, Y., Zeng, C., Wan, K., 2015. Analysis of embCAB Mutations Associated with Ethambutol Resistance in Multidrug-Resistant Mycobacterium tuberculosis Isolates from China, Antimicrobial Agents and Chemotherapy, 59: 2045-2050.
-
[14] Sun, Q., Xiao, T., Liu, H., Zhao, X., Liu, Z., Li, Y., Zeng, H., Zhao, L., and Wan, K., 2018. Mutations within
embCAB Are Associated with Variable Level of Ethambutol Resistance in Mycobacterium tuberculosis Isolates from China, Antimicrobial Agents and Chemotherapy, 62: 1-8.
-
[15] Khosravi, A.D., Meghdadi, H., Ghadiri, A., Alami, A., Sina, A.H., Mirsaeidi, M., 2018. rpoB gene
mutations among Mycobacterium tuberculosis isolates from
extrapulmonary sites. Journal of
Pathology, Microbiology and
Immunology, 126: 241-247.
-
[16] Walker, J.M. dan Rapley, R., 2008. Medical Biomethods Handbook, Humana Press, Totowa, New Jersey.
-
[17] Alvandi, E., and Koohdani, F., 2014. Zip nucleic acid: a new reliable method to increase the melting temperature of real-time PCR probes, Journal of
Diabetes & Metabolic Disorders, 13: 1-4.
-
[18] Dorak, M.T. 2006, Real-Time PCR, Taylor and Francis Group, New York.
-
[19] Anonim, 2006. Real-Time PCR Application Guide, Bio-Rad
Laboratories Inc, USA.
-
[20] Walker, J.M. and Rapley, R., 2005. Medical Biomethods Handbook,
Humana Press, Totowa, New Jersey.
-
[21] Bishop, J.L., Campbell, S. A., Farrell, P., Fitzgerald, M., Haugen, M., Kocmond, W., Madden, D. E., Murray, W. E., and Persing, D. H., 2015.
Designing Real Time Assays on the SmartCycler® II System, Cepheid
Technical Support, United States
-
[22] Borah, P., 2011. Primer Designing for PCR, Science Vision, 11 : 134-136.
-
[23] Butler, J.M. 2011, Advanced topics in forensic DNA typing: methodology, Academic press, USA.
-
[24] Johansson, M.K., 2006. Choosing
Reporter-Ouencher Pairs for Efficient Quenching Through Formation of
Intramolecular Dimers, Methods in Molecular Biology, 335: 17-29.
-
[25] Murray, J. L., Hu, P., dan Shafer D. A., 2014. Seven Novel Probe System for RealTime PCR Provide Absolute Single-Base Discrimination, Higher Signaling, and Generic Components, The Journal of Molecular Diagnostics, 16: 627-638.
-
[26] Nazarenko, I., Pires, R., Lowe, B., Obaidy, M., dan Rashtchian, A., 2002. Effect of Primary and Secondary Structure of Oligodeoxyribonucleotides on The Fluorescent Properties of Conjugated Dyes. Nucleic Acids Research, 30: 2089-2195.
-
[27] Didenko, V. V. 2006. Fluorescent Energy Transfer Nucleic Acid Probe Design and Protocols. Humana Press Inc, Totowa, New Jersey. 17-80.
-
[28] Livak, K. J., Flood S. J. A., Marmaro, J., Giusti, W., dan Deetz, K., 1995. Oligonucleotides With Fluorescent Dyes At Opposite Ends Provide A Quenched Probe System Useful For
Detecting PCR Product and Nucleic Acid Hybridization. Genome Research. 4: 357-362.
-
[29] Marras, S.A., 2006. ‘Selection of
fluorophore and quencher pairs for fluorescent nucleic acid hybridization probes’ in Didenko, Fluorescent energy transfer nucleic acid probes, Humana Press, Totowa, pp 3-16.
50
Discussion and feedback