STUDI IN SILICO CONVERSE REGION ETOPOSITE BINDING DOMAIN Pada ISOZIM HUMAN DNA TOPOISOMERASE II
on
Cakra Kimia (Indonesian E-Journal of Applied Chemistry)
Volume 4, Nomor 1, Mei 2016
STUDI IN SILICO CONVERSE REGION ETOPOSITE BINDING DOMAIN Pada ISOZIM HUMAN DNA TOPOISOMERASE II
Tirta Setiawan*1, Laksmi Ambarsari1,2, Tony Ibnu Sumaryada2,3
1Departemen Biokimia, Fakultas Matematika dan Ilmu Pengetahuan Alam, Institut Pertanian Bogor, 16680
2Pusat Penelitian Biofarmaka, Institute Pertanian Bogor, Jalan Taman Kencana No.3, Bogor
16128
3
3Departemen Fisika, Fakultas Matematika dan Ilmu Pengetahuan Alam, Institut Pertanian Bogor,16680
Email: Tirtasetiawan@apps.ipb.ac.id1, ami_icha@yahoo.com1,2, sumaryada@gmail.com2,3
ABSTRAK: DNA topoisomerase II (Top2) adalah enzim penting yang menangani permasalahan topologi pada DNA. Terdapat dua jenis Top2 pada manusia yaitu Top2A dan Top2B. Top2A diekspresikan pada sel yang aktif membelah seperti sel kanker sedangkan Top2B diekspresikan saat sel dalam fase istirahat. Peran penting inilah yang membuat DNA Top2A menjadi target obat melawan kanker salah satunya obat etoposite. Sisi aktif pengikatan etoposite terhadap Top2B sudah dilakukan namun pada Top2A belum dilakukan. Studi pencarian area yang rapat dengan pensejajaran banyak sekuen pada Top2A dengan Top2B sebagai acuan bertujuan untuk pencarian sisi aktif pengikatan etoposite pada Top2A. Area yang rapat dengan kesamaan residu asam amino yang tinggi dapat digunakan untuk memprediksikan fungsi penting asam amino dalam enzim tersebut. Top2A dan Top2B memiliki kesamaan asam amino sebesar 80% dengan kesamaan struktur 71.81%. Asam amino aktif pengikatan etoposite pada Top2B Gly478, Asp479, Arg503, Met782 dan Gln778, dinyatakan mirip dan rapat terhadap residu Gly462, Asp463, Arg487, Met763 pada Top2A kecuali residu Gln778 dari Top2B namun penggantian ini tidak merubah struktur dari enzim tersebut karena sama-sama membentuk struktur alfa-heliks dan terletak pada posisi yang sama sehingga diprediksikan sisi aktif pengikatan etoposite pada enzim Top2B yaitu Gly478, Asp479, Arg503, Met782 dan Gln778memiliki fungsi yang sama dengan residu Gly462, Asp463, Arg487, Met763 dan Met767 dari Top2A. Hal ini menyebabkan etoposite juga dapat menghambat kerja enzim Top2A pada residu yang sama.
Keyword: DNA topoisomerase, area yang rapat, Pensejajaran banyak sekuen.
ABSTRACT: DNA topoisomerase II (Top2) is essential enzyme that solves the topological problems of DNA. Top2A and Top2B are two kind of Top2 in human. Top2A is expressed at differentiation active cells as cancer cells whereas Top2B is expressed at non differentiation cells or quiescent cells. Their critical role makes Top2B an attractive drug target against cancer as etoposite known as Top2 inhibitor. Active site for etoposite binding domain to Top2B has been done but in Top2A has not been done yet. Conserve region study by multiple sequence alignment between Top2A and Top2B as Top2B to be a template was aimed to search an active site for etoposite binding domain in Top2A. A conserve amino acid area with highest similarity was
used to predict essential amino acid activity in the enzyme. Homology study shows that Top2A and Top2B have the same similarity of amino acids of 80% with structure similarity of 71.81%. Active amino acids on Top2B for etoposite binding domain such as Gly478, Asp479, Arg503, Met782 dan Gln778 were found to be highly conserve with amino acid Gly462, Asp463, Arg487, Met763 on Top2A except Gln778 from Top2B but this does not change of structure of the enzyme because they have the same alfa-helix formations and positions so that it could be predicted that amino acids Gly478, Asp479, Arg503, Met782 and Gln778 on Top2B have similar activity and function with Gly462, Asp463, Arg487, Met763 and Met767 on Top2A. This etoposite could inhibit the activity of the Top2A enzyme at same site.
Keyword: DNA topoisomerase, Conserve region, Multiple sequence alignment
1.
PENDAHULUAN
Enzim Topoisomerase adalah enzim yang mampu memodifikasi topologi DNA dan berperan aktif dalam proses replikasi, transkripsi dan segregasi kromosom [1]. Sel manusia memiliki dua anggota keluarga Topoisomerase yaitu: Top1 dan Top2. Ada dua isoform Top2 yaitu Top2A dan Top2B. Sehubungan dengan fungsinya pada pembelahan sel, ekspresi Top2A berkaitan erat dengan proliferasi sel. Ekspresi Top2A sedikit pada fase G1, meningkat pada fase S dan fase G2/M, sedangkan ekspresi Top2B relatif konstan selama siklus sel. Top2A khusus diekspresikan pada sel yang sedang berproliferasi dan Top2B pada sel yang tidak berproliferasi atau istirahat [2,3].
Enzim Top2 membutuhkan dua ATP untuk berkerja, sehingga memiliki bagian ATP binding domain. Berdasarkan aktivitasnya enzim ini memiliki sisi katalitik Tyr yang bertugas memotong dan menyambungkan kembali ikatan fosfodiester utas DNA. Selain itu dalam beberapa penelitian, telah dikembangkan beberapa jenis obat yang menargetkan enzim topoisomerase 2 untuk menekan laju proliferasi sel-sel kanker. Mekanisme penghambatannya dibagi dalam dua kategori aktivitas, pertama menyerang domain N-terminal ATPase [4,5]
dan mencegahnya untuk beraktivitas dan meracuni Top2 sehingga menginduksi pemotongan dan mencegah penyambungan kembali DNA [6,7,8].
Etoposite adalah salah satu obat golongan antrasiklin yang berkerja menghambat enzim Top2 dengan kategori aktivitas meracuni Top2. Studi mengenai penggunaanya dapat menghambat kedua jenis enzim Top2 masih belum banyak dilakukan. Walaupun enzim topoisomerase 2A dan 2B merupakan isozim dan mempunyai fungsi spesifik yang sama, namun kedua diekspresikan pada waktu yang berbeda.Pemetaan posisi dan struktur 3D dengan Xray kompleks Top 2B dengan etoposite (PDB ID: 3QX3) telah dilakukan oleh Wu [9] sehingga sudah diketahui binding domain Top 2B dengan etoposite namun untuk Top 2A belum diketahui. Enzim Top 2A lebih banyak diekspresikan pada sel-sel kanker yang aktif membelah. Sel mcf-7 diketahui mengekspresikan enzim Top 2A yang sangat tinggi [10]. Setiap aktifitas tersebut pasti disebabkan oleh adanya peran beberapa residu yang rapat (conserved) pada enzim tersebut yang tentunya penting dan selalu dipertahankan keberadaannya agar tidak hilang atau termutasi. Berubahnya salah residu ini akan mengakibatkan enzim kehilangan aktivitasnya [11]. Untuk mengetahui residu
asam amino yang berinteraksi dengan etoposite pada Top 2A perlu dilakukan analisis lebih lanjut pada kedua enzim.
Bioinformatika atau in silico memiliki potensi yang besar untuk menganalisa dan memprediksikan proses biokimia terutama sekali untuk menentukan hubungan antara struktur dan fungsi biomolekul. Dengan bioinformatika, hubungan residu terhadap struktur dan fungsi dapat diketahui lebih mendalam salah satunya adalah metode homologi protein dan multiple alignment untuk melihat daerah yang conserved.Conserved region dapat digunakan untuk melihat hubungan evolusi antar sekuen, melihat area berulang pada sekuen yang berbeda dapat diprediksikan memiliki struktur yang mirip, jika pada DNA dapat digunakan untuk mencari sekuen regulator dan dapat digunakan untuk menyusun probe atau primer DNA yang dapat digunakan untuk penelitian biologi molekular seperti PCR [12]. Selain itu urutan asam amino khas yang dipertahankan terhadap proses evolusi menunjukkan bahwa asam amino yang rapat (conserved) tersebut memiliki peran penting dan diprediksikan sebagai salah satu sisi aktif [13].
Penelitian ini bertujuan untuk melihat asam amino yang rapat (conserved)etoposite binding domain pada enzim Top 2A berdasarkan template Top 2B yang memiliki kompleks dengan etoposite (3QX3) dengan memanfaatkan metode multiple sequence alignmentbioinformatika.
Sekuen protein Top2A dan Top2B diambil dari protein data bank (www.rcsb.org/pdb/) kemudian masing di didownload file berekstensi (.Fasta).
Blast dilakukan dengan metode BLASTp, masing-masing file fasta di proses dalam fasilitas Blast (www.uniprot.org/blast)diatur masing-masing dengan target database: UniProtKB, EThreshold: 10, Matrix: Auto, Filtering : None, Gapped: yes dan Hitts: 50.
Hitts yang tertampil dari hasil masing-masing data BLASTp enzim topoisomerase 2A dan topoisomerase 2B di download dalam bentuk file (.fasta) untuk kemudian digabungkan dalam satu file (.fasta). Multiple alignment dilakukan dengan metode ClustalW2 (www.ebi.ac.uk/tools/ msa/clustalw2) kemudian diinput file fasta gabungan tadi. File akan diproses 1 x 24 jam.Publikasi struktur X-ray Top2B yang berkompleks dengan etoposite telah dilakukan oleh Wu et al. (2011) [9]dan dijadikan template untuk melihat asam aminoconsevedyang sama pada Top2A.
Setelah dianalisis conserved region untuk binding domain etoposite pada Top2A, dilakukan superimpose struktur sekunder 3D tiap-tiap residu dengan menggunakan VMD 1.9.1 berdasarkan termplate Top2B yang memiliki kompleks dengan etoposite, untuk melihat posisi residu tersebut berjauhan atau berdekatan.
Sekuen protein yang digunakan sebagai bahan penelitian adalah sekuen yang berasal dari topoisomerase 2A manusia (Top2A) dengan template topoisomerase 2B (Top2B) berkode PBD ID: 3QX3 yang dipublikasi oleh Wu et al. (2011) dengan struktur kristal X-ray Top2Bberkompleks dengan etoposite. Pemilihan Top2B sebagai template dikarenakan enzim ini memiliki kompleks dengan etoposite karena etoposite merupakan obat antikanker yang bekerja
dengan spesifikasi meracuni Top2, membuat DNA dan Top2 tidak dapat lepas, banyak DNA yang tidak tersambung kembali yang berujung kepada kematian sel.Karena kompleks etoposite-DNA-Top2B sudah berhasil dipetakan, residu yang berinteraksi dengan etoposite juga sudah berhasil dipetakan dengan kode 3QX3_A_AC1_10 (gambar 2) yang dinamai sebagai site recordetoposide binding domain(sisi aktif pengikatan etoposite) pada Top2B dengan residunya yaituGly478, Asp479, Arg503, Met782 dan Gln778[14]. Kelima asam amino inilah yang kemudian akan disejajarkan dengan Top2A dari manusia untuk dilihat kerapatan(conserve )asam amino.
Conserved region dapat digunakan untuk melihat hubungan evolusi antar sekuen, melihat area berulang pada sekuen yang berbeda dapat diprediksikan memiliki struktur yang mirip, jika pada DNA dapat digunakan untuk mencari sekuen regulator dan dapat digunakan untuk menyusun probe atau primer DNA yang dapat digunakan untuk penelitian biologi molekular seperti PCR [12]. Selain itu daerah yang dipertahankan terhadap proses evolusi evolusi dan semua organisme menunjukkan bahwa residu yang conserved tersebut memiliki peran penting dan diprediksikan sebagai salah satu sisi aktif [13].
Pemilihan Top2A dikarenakan enzim Top2A khusus diekspresikan pada sel yang sedang berproliferasi dan enzim Top2B pada sel yang tidak berproliferasi atau istirahat (quiescent) [2]. Ekspresi enzim Top2A sedikit pada fase G1, meningkat pada fase S dan fase G2/M, sedangkan ekspresi enzim Top2B relatif konstan selama siklus sel. Enzim Top2A ini diketahui memiliki tingkat ekspresi yang tinggi pada sel-sel kanker dan paling tinggi ekspresinya pada kanker Hati (Hep27) dan kanker payudara (mcf-7) [15]. Sel line mcf-7 adalah sel yang diisolasi dari jaringan manusia dan tergolong dalam jenis kanker payudara menunjukkan penurunan drastis tingkat ekspresi top2A ketika diberikan
perlakuan etoposite dan doksorubisin [10]. Enzim Top2 ini merupakan target penting obat kemoterapi golongan antrasiklin seperti etoposite dan doksorubisin.Namundata site record obat dengan enzim Top2A juga belum ada publikasinya sehingga perlu dilakukanmultiple alignmentsequence dan analisisdaerah yang rapat (conserve region) untuk memprediksikan sisi aktif pengikatan etoposite.
Hasil BLASTp dengan Top2B berkode 3QX3 sebagai template ditemukan 250 hasil dengan rincian sebelas isozim Top2 dari manusia dan lebih dari seratus yang diketahui berasal dari 14 spesies organisme yang berbeda (Tabel 1) dan semuanya adalah isozim Top2B tidak terdapat satupun dari jenis Top2A.Top2B dari 14 organisme tersebut terlihat memiliki homologi yang sangat tinggi sehingga diprediksikan memiliki kesamaan asam amino dan struktur.
Tabel 1. Hasil BLASTp Top2B Bukan Manusia Dengan Template Top2B Manusia
|
Query |
Spesies |
Homologi % |
|
H2QCW5 |
Pan troglodytes |
99 |
|
K7BYJ3 |
Pan troglodytes |
99 |
|
G3RAE1 |
Gorilla gorilla gorilla |
99 |
|
A0A096P5G9 |
Papio anubis |
99 |
|
F7EVK2 |
Macaca mulatta |
99 |
|
F7EVJ4 |
Macaca mulatta |
99 |
|
H9YVX9 |
Macaca mulatta |
99 |
|
G3S3V9 |
Gorilla gorilla gorilla |
99 |
|
F6T9B4 |
Callithrix jacchus |
99 |
|
F6S0Y0 |
Callithrix jacchus |
99 |
|
H2NU77 |
Pongo abelii |
99 |
|
O46374 |
Sus scrofa |
98 |
|
L9JX05 |
Tupaia chinensis |
98 |
|
G1LN78 |
Ailuropoda melanoleuca |
98 |
|
G9KUN2 |
Mustela putorius furo |
98 |
|
W5PY48 |
Ovis aries |
98 |
|
I3NGU9 |
Spermaphilus tridecemlineatus |
98 |
|
I3M4Z3 |
Spermaphilus tridecemlineatus |
98 |
|
L8IH03 |
Bos mutus |
98 |
|
F1MDU7 |
Bos taurus |
98 |
|
H0WJ18 |
Otolemur garnettii |
97 |
Terdapat sebelas isozim Top2 hasil BLASPp asal manusia (Gambar 1) dengan rincian empat enzim Top2A manusia dengan homologi 80% terhadap templatenya Top2B Sedangkan untuk enzim Top2B ditemukan tujuh dengan homologi diatas 90%.Data ini menunjukkan bahwa antara enzim Top2A dan Top2B memiliki perbedaan banyak asam amino. Untuk melihat seberapa jauh perbedaan dan komposisi asam aminonya dilakukan multiple alignment sequence antara Top2A dan Top2B untuk melihat area yang conserve. Namun dua data dengan kode Q59480 dan Q6W6M6 tidak dapat dilakukan alignment karena Match Hits nya berada jauh dari template dan akan berakibat menurunkan ketelitian analisis area yang rapat ketikan menggunakan ClustaW [13].
Dari hasil multiple alignment sequence dengan menggunakantemplate Top2B
didapatkan data bahwa active site record etoposite binding domain pada Top2B
(gambar 3-5)pada residu Gly478, Asp479,
Arg503, Met782 dan Gln778[14], conserve terhadap residu Gly462, Asp463, Arg487,
Met763(tanda titik biru diatas menunjukkan asam amino conserve terhadap site record 778 etoposite untuk Top2B).Namun residu Gln778 dari Top2B digantikan dengan oleh residu Met767. Namun penggantian ini tidak merubah struktur dari enzim tersebut karena sama-sama membentuk struktur alfa-heliks. Residu yang diblok berwana pink (Gambar 3-5) menunjukkan residu yang berperan dalam pembentuka struktur alfa-Heliks, warna coklat berperan dalam pembentukan struktur beta.
Selanjutnya adalah alignment struktur 3D (superimpose)untuk melihat kemiripan bentuk struktur 3D-nya. Pencarian struktur 3D X-ray Top2 pada PDB bank (Tabel 2) didapatkan data bahwa empat enzim Top2A dari hasil BLASTp memiliki struktur 3D Xray pada PBD bank dengan kode PBD 4RIF,
1ZXM, 1ZXN dan 4FM9. Dilihat dari urutan asam aminonya, Top2A berkode 4FM9 satu-satunya yang memiliki kesaman match hits, oleh karena itu digunakan untuk mewakili enzim Top2A untuk dilakukan alignment struktur 3D. Sebelum dilakukan alignment 3D, dilakukan minimisasi energi (www.zhang-lab.org), hal ini bertujuan agar didapatkan konfromasi yang paling stabil. Hasil alignment struktur 3D didapatkan hasil kecocokan antara Top2A dan Top2B adalah sebesar 71.81 %. Nilai QH menunjukkan keidentikan bentuk struktur sekunder antara kedua protein, nilai QH 0.1-0.3 menunjukkan keidentikan yang sangat jauh, nilai QH = 1 menunjukkan sangat identik, nilai QH untuk Top2A dan B adalah 0.7181 sehingga dikatan tidak identik namun terlalu tidak jauh berbeda [16,17]. Komposisi asam amino dari kedua enzim (Tabel 3) menunjukkan jumlah yang berbeda-beda namun memiliki persentase kompoisi yang tidak jauh berbeda, hal ini menunjukan bahwa kedua enzim Top2A dan Top2B adalah Isozim.
Untuk melihat letak residu untuk daerah aktif etoposite antara Top2A dan Top2B, maka dilakukan superimpose struktur dengan menggunakan VMD 1.9.1 dengan fasilitas stamp alignment dan multySeq [18]. Kemudian dilakukan visulisasi antara area yang conserved pada sisi aktif daerah pengikatan etoposite dari keduanya yaitu pada residu Gly478, Asp479, Arg503, Met782 dan Gln778dari Top2B dan Gly462, Asp463, Arg487, Met763 dan Met767 dari Top2A. Dari hasil visualisasi terlihat bahwa semua residu terletak pada area dan posisi yang sama (Gambar 7).
|
Entry |
Protein names |
Match hit ⅛ 2k ⅛ 4 ⅛' |
Identity |
Q02880-2
Q02880
B4DKD0
Q71UH4
E9PCY5
P11388-4
P11388-3
P11388-2
P11388
Q59H80
Q6W6M6
Ti Isoform Beta-I of DNAtopotsomerase 2-beta (Homo sapiens)
⅛ DNA topoisomerase 2-beta (Homo sapiens)
DNA topoisomerase 2 (Homo sapiens)
DNA topoisomerase 2 (Homo sapiens)
DNA topoisomerase 2 (Homo sapiens)
⅛ Isoform 4 of DNA topoisomerase 2-alpha (Homo sapiens)
⅛ Isoform 3 of DNA topoisomerase 2-alpha (Homo sapiens)
⅛ Isoform 2 of DNA topoisomerase 2-alpha (Homo sapiens)
⅞ DNA topoisomerase 2-alpha (Homo sapiens)
DNA topoisomerase II, beta isozyme variant (Homo sapiens)
Antigen MLAA-44 (Homo sapiens)
99.0%
99.0%
99.0%
99.0%
100.0%
80.0%
80.0%
80.0%
80.0%
99.0%
97.0%
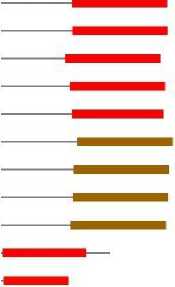
Gambar 1. Hasil BLASTp Top 2 dengan Top 2B sebagai template pada manusia. (tanda bintang memiliki makna terlah tersedia PDB file)

BINDING SITE FOR RESIDUE EVP A 1 (SOFTWARE)
Gambar 2. Site recorddaerah aktif etoposite binding domain pada topoisomerase 2B yaitu Gly478, Asp479, Arg503, Met782 dan Gln778[14].
|
Q02880-2 |
T0P2B HUMAN |
813 |
Gkdaaspryiftmlstlarllfpavddnllkflyddnqrvepewyipiipmvlingaegi |
|
Q0288Θ |
T0P2B HUMAN |
818 |
Gkdaaspriiftmlstlarllfpavddnllkflyddnqrvepewyipiipmvlingaegi |
|
B4DKDΘ |
B4DKD0 HUMAN |
737 |
Gkdaaspryiftmlstlarllfpavddnllkflyddnqrvepewyipiipmvlingaegi |
|
Q71UH4 |
Q71UH4 HUMAN |
790 |
Gkdaaspryiftmlstlarllfpavddnllkflyddnqrvepewyipiipmvlingaegi |
|
E9PCY5 |
E9PCY5 HUMAN |
813 |
Gkdaaspryiftmlstlarllfpavddnllkflyddnqrvepewyipiipmvlingaegi |
|
P11388-4 |
T0P2A HUMAN |
878 |
Gkdsaspryiftmlsslarllfppkddhtlkflyddnqrvepewyipiipmvlingaegi |
|
P11388-3 |
T0P2A HUMAN |
833 |
Gkdsaspryiftmlsslarllfppkddhtlkflyddnqrvepewyipiipmvlingaegi |
|
P11388-2 |
T0P2A HUMAN |
823 |
Gkdsaspryiftmlsslarllfppkddhtlkflyddnqrvepewyipiipmvlingaegi |
|
P11388 |
T0P2A HUMAN |
797 |
Gkdsaspriiftmlsslarllfppkddhtlkflyddnqrvepewyipiipmvlingaegi |
Gambar 3. Multiple alignment Tyr antara topoisomerase 2A dan 2B
|
QΘ2880-2 |
T0P2B HUMAN |
453 |
Gipklddandaggkhslectliltegdsakslavsglgvigrdrygvfplrgkilnvrea |
|
QΘ2880 |
T0P2B HUMAN |
458 |
Gipklddandaggkhslectliltegdsaks LavsglgvigrdrygvfplrgIil∣vrea |
|
B4DKD0 |
B4DKD0 HUMAN |
377 |
Gipklddandaggkhslectliltegdsakslavsglgvigrdrygvfplrgkilnvrea |
|
Q71UH4 |
Q71UH4 HUMAN |
430 |
Gipklddandaggkhslectliltegdsakslavsglgvigrdrygvfplrgkilnvrea |
|
E9PCY5 |
E9PCY5 HUMAN |
453 |
Gipklddandaggkhslectliltegdsakslavsglgvigrdrygvfplrgkilnvrea |
|
P11388-4 |
T0P2A HUMAN |
518 |
Gipklddandaggrnstectliltegdsaktlavsglgvvgrdkygvfplrgkilnvrea |
|
P11388-3 |
T0P2A HUMAN |
473 |
Gipklddandaggrnstectliltegdsaktlavsglgwgrdkygvfplrgkilnvrea |
|
P11388-2 |
T0P2A HUMAN |
463 |
Gipklddandaggrnstectliltegdsaktlavsglgwgrdkygvfplrgkilnvrea |
|
P11388 |
T0P2A HUMAN |
437 |
Gipklddandaggrnstectliltegdsaktlavsglgwgi >k∖ ■ pi rg∣il∣vrea |
Gambar 4. Multiple alignment residu Gly478, Asp479, Arg503 antara topoisomerase tipe 2 A dan 2B
|
Q02880-2 |
T0P2B HUMAN |
753 |
|
Q02880 |
T0P2B HUMAN |
758 |
|
B4DKD0 |
B4DKD0 HUMAN |
677 |
|
Q71∪H4 |
Q71UH4 HUMAN |
730 |
|
E9PCY5 |
E9PCY5 HUMAN |
753 |
|
P11388-4 |
T0P2A HUMAN |
S18 |
|
P11388-3 |
T0P2A HUMAN |
773 |
|
P11388-2 |
T0P2A HUMAN |
763 |
|
P11388 |
T0P2A HUMAN |
737 |
Ndkrevkvaqlagsvaemsayhhgeqalmmtivnlaqnfvgsnninllqpigqfgtrlhg NdkrevkvaolagsvaemsayhhgeqalmmtivnLaonfvgsnninllqpigqfgtrlhg Ndkrevkvaqlagsvaemsayhhgeqalmmtivnlaqnfvgsnninllqpigqfgtrlhg Ndkrevkvaqlagsvaemsayhhgeqalmmtivnlaqnfvgsnninllqpigqfgtrlhg Ndkrevkvaqlagsvaemsayhhgeqalmmtivnlaqnfvgsnninllqpigqfgtrlhg Ndkrevkvaqlagsvaemssyhhgemslmmtiinlaqnfvgsnnlnllqpigqfgtrlhg Ndkrevkvaqlagsvaemssyhhgemslmmtiinlaqnfvgsnnlnllqpigqfgtrlhg Ndkrevkvaqlagsvaemssyhhgemslmmtiinlaqnfvgsnnlnllqpigqfgtrlhg ndkrevi<vaqlag≤vaemsξ∣hhgem∣lmmtiinlaqnfvgsnnlnllqpigqfgtrlhg
Gambar 5. Multiple alignment residu Met782, dan Gln778antara topoisomerase tipe 2A dan 2B
Tabel 2. Hasil Pencarian Struktur 3D X-Ray Kristal Top2A dan Top2B.
|
Enzim |
Query |
PDB |
Nomor residu |
Kompleks | |
|
DNA |
Ligand | ||||
|
Topoisomerase II A |
P11388-4 |
4RIF |
29-428 |
- |
ADP & SO4 |
|
P11388-3 |
1ZXM |
29-428 |
- |
AMP-PNP | |
|
P11388-2 |
1ZXN |
29-428 |
- |
ADP | |
|
P11388 |
4FM9 |
431-1193 |
YA |
Gliserol | |
|
Topoisomerase II B |
Q02880 |
3QX3 |
459-1206 |
YA |
Etoposite |
|
Q02880-2 |
4G0U |
450-1212 |
YA |
Amsacrin | |
|
E9PCY5 |
- |
450-1212 |
YA |
Mitosantron | |
|
Q71UH4 |
- |
450-1212 |
YA |
Ametantron | |
B.
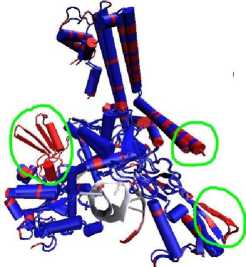
Gambar 6. A) superimpose struktur 3D Top2A dengan template Top2B, warna biru (kesamaan residu) dan warna merah (perbedaan residu), B). indeks alignment dari tools multi sekuen menggunakan VMD 1.8.1. dan lingkaran hijau menandakan perbedaan struktur yang sangat nyata.
Tabel 3. Perbandingan Komposisi Asam Amino Pada 3qx3 (Top2B) Dengan 4fm9 (Top2A).
|
No |
Asam amino |
Simbol |
Singkatan |
Jumlah 3qx3 4fm9 |
Klasifikasi |
Persentase (%) 3qx3 4fm9 |
|
1 Alanin |
Ala |
A |
49 |
31 | |
|
2 Valin |
Val |
V |
41 |
37 | |
|
3 Leusinsin |
Leu |
L |
76 |
81 |
Asam amino |
|
4 Isoleusin |
Ile |
I |
49 |
49 |
dengan 38.605 38.0079 |
|
5 Prolin |
Pro |
P |
30 |
32 |
Gugus -R |
|
6 Fenilalanin |
Phe |
F |
33 |
29 |
tak mengkutub |
|
7 Triptofan |
Trp |
W |
12 |
11 | |
|
8 Metionin |
Met |
M |
20 |
20 | |
|
9 Glisin |
Gly |
G |
50 |
44 | |
|
10 Serin |
Ser |
S |
48 |
44 |
Asam amino |
|
11 Treonin |
Thr |
T |
39 |
43 |
dengan |
|
12 Sistein |
Cys |
C |
5 |
6 |
Gugus -R 30.012 30.2752 |
|
13 Tirosin |
Tyr |
Y |
27 |
28 |
mengkutub |
|
14 Aspargarin |
Asn |
N |
39 |
40 |
tak bermuatan |
|
15 Glutamin |
Gln |
Q |
33 |
26 | |
|
16 Asam aspartat |
Asp |
D |
55 |
47 |
„ . ' f Asam amino sam am no 14.446 15.3342 |
|
17 Asam glutamat |
Glu |
E |
61 |
70 |
R Negatif |
|
18 Lisin |
Lys |
K |
72 |
74 |
Asam amino |
|
19 Arginin |
Arg |
R |
35 |
36 |
Basa 16.936 16.3827 |
|
20 Histidin |
His |
H |
29 |
15 | |
|
Total Asam amino |
803 |
763 | |||
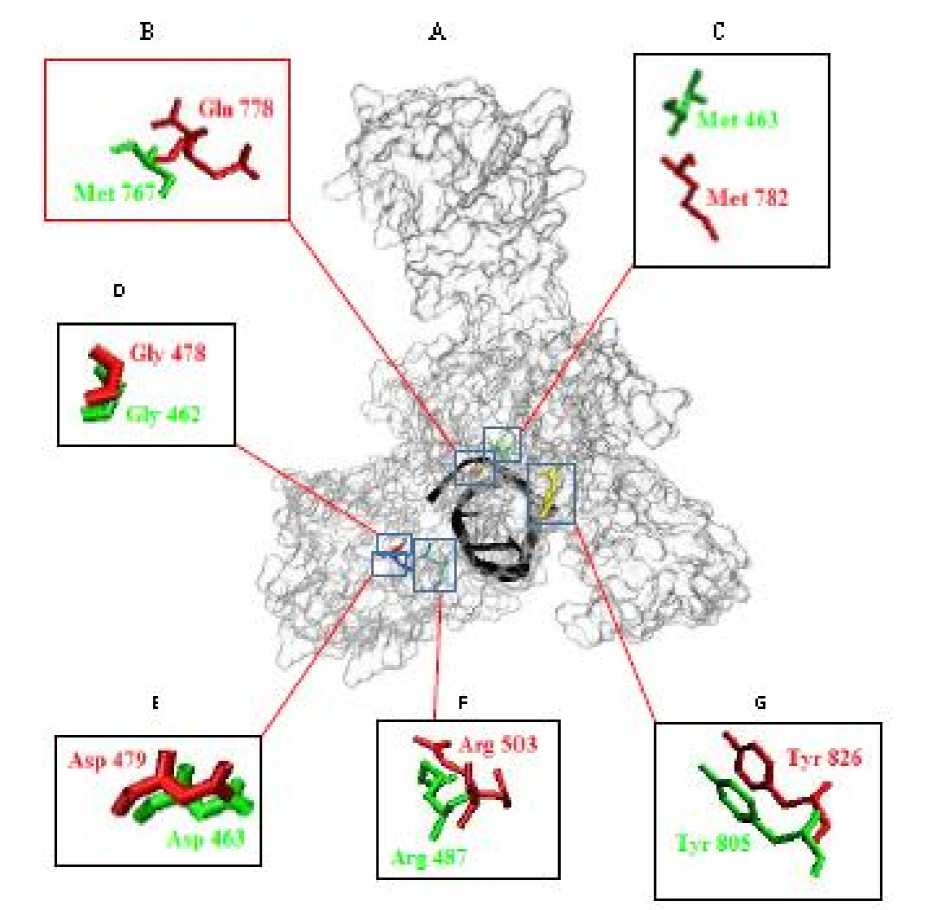
Gambar 7. A) superimpose Top2A dan B (subunit A), B) Met782, C) Gln778, D) Gly478, E) Asp479, F) Arg503 dan G) Tyr 826.
A.
Selain sisi aktif untuk pengikatan etoposite, enzim Top2 juga memiliki daerah aktif yang berfungsi memotong ikatan fosfodiester utas DNA yang dilakukan oleh residu Tyr826 (Wu et al. 2011) pada top2B, dan dari hasil multiple alignment sequence, conserve region serta visualisasi, Top2A juga memiliki residu Tyr805 yang conserve terhadap
residu Tyr826 pada Top2B (Gambar 3) dengan posisi yang sama (Gambar7). Dari penelitian ini maka dapat dikatakan bahwa sisi aktif pengikatan etoposite pada enzim Top2B yaitu Gly478, Asp479, Arg503, Met782 dan Gln778diprediksikan memiliki fungsi yang sama dengan residu Gly462, Asp463, Arg487, Met763 dan Met767 dari Top2A. Sehingga
etoposite juga dapat menghambat kerja enzim Top2A pada residu yang sama.
Terimakasih kepada kepala
laboratorium Mikrobiologi PSSP (pusat studi satwa primata), seluruh staf pengajar Departemen Biokimia MIPA IPB dan komunitas online Research gate yang telah memberikan solusi pada forum tanya jawabnya.
DAFTAR PUSTAKA
-
[1] Nitis JL, Beck WT. 1996, “Antitopoisomerase drug action and resistance”, Eur J Cancer. 10: 68-75.
-
[2] Fry AM, Chresta CM, Davies SM. 1991, “Relationship between topoisomerase II level and chemosensitivity in human tumor sell lines”, Cancer Res. 51: 65926595.
-
[3] Bauman ME, Holden JA, Brown KA, Harker WG, Perkins SR. 1997,
”Differenttial immunohistochemical
staining for DNA topoisomerase IIA and B in human tissue and for DNA topoisomerase IIB in non Hodkin limphoma”, Mod Pathol. 10: 168-175.
-
[4] Baird CL, Gordon MS, Andrenyak DM, Marecek JF, Lindsley JE. 2001,”The ATPase reaction cycle of yeast DNA topoisomerase II: slow rates of ATP resynthesis and Pi release”, J Biol Chem. 276: 27893-27898.
-
[5] Robinson HM, Bratlie-Thoresen S,
Brown R, Gillespie DA. 2007,”Chk1 is required for G2/M check-point response induced by the catalitic topoisomerase II inhibitor ICRF-1993”, Cell Cycle. 6: 1265-1267.
-
[6] Chow KC, Macdonald TL, Ross WE.
1998, “DNA binding by epipodophyllo-toxins and N-acyl antracyclines: Implications for mechanism of
topoisomerase inhbition”, Mol.
Pharmacol. 34: 467-473.
-
[7] Osheroff N. 1989, “Effect of antineoplastic agent on the DNA cleavage/religation equlibrium of eukariotic topoisomerase II: inhibition of DNA religation by etoposide”, Biochemistry. 28: 6157-6160.
-
[8] Beck LS, Deguzman L, Lee WP, Xu Y, Siegel MW, Amento EP. 1993,”One systemic administration of transforming growth factor-beta 1 reverses age or glucocorticoid impaired wound
healing”, J Clin Invest. 92: 2841-2849.
-
[9] Wu CC, Li TK, Far HL, Lin LY, Lin TS, YU YJ, Ten TJ, Chiang CW, Chan NL. 2011, ”Structural basis of type II topoisomerase inhibition by the anticancer drug etoposite”, J science. 333: 459-462.
-
[10] Kaplan E, Gunduz U. 2011, “Expression analysis of TOP2A, MSH2 adn MLH1 genes in MCF7 cells at different levels of etoposite resistance”, Eur J Pharm. 66: 29-35.
-
[11] Nelson D, Cox M. 2008. Principle of Biochemistry (5th Edn.). W.H. freeman and company, USA. ISBN-13: 978-07176-7108-1.
-
[12] Mount DW. 2004. Bioinformatics sequences and genome analysis. Cold Spring Harbor Laboratory Press. New York.
-
[13] Baxevanis AD, Ouellette BF. 2001. Bioinformatics A Practical Guide to the Analysis of Genes and Proteins Second Edition. 1-457.
-
[14] Golovin A, Dimitropolos D, Oldfield T, Rachedi A, Henrick K. 2005. MSDsite: a data base search and retrieval system for the analysis and viewing of bound ligands and active site. Protein. 58: 190199.
-
[15] AbuHammad S, Zihlif M. 2012, “Gene expression alteration in doxorunicin resistant MCF-7 breast cancer cell line”, Genomic. 101: 213-220.
-
[16] Thompson JD. 1994, “CLUSTAL W: improving the sensitivity of progressive multiple sequence alignment through sequence weighting, position-specific gap penalties and weight matrix choice”, Nucleic Acids Res. 22: 4673–4680.
-
[17] Larkin MA, Blackshields G, Brown NP, Chenna R, McGettingan PA, Mc
William H, Valentin F, Wallace IM, Wilm A, Lopez R, Thompson JD, Gibson TJ, Higgins DG. 2007, clustal w and clustal x version 2.0. Bioinformatic. 23: 2947-2948.
-
[18] Russell RB, Breed J, Barton GJ. 1992, “Conservation analysis and secondary structure prediction of the sh2 family of phosphotyrosine binding domains”, Proteins: Struct., Funct., Genet. 304, 15-20.
70

Discussion and feedback