EFFICIENCY OF USING TAQ POLYMERASE ENZYME IN POLYMERASE CHAIN REACTION TESTING
on
Buletin Veteriner Udayana Volume 15 No. 5: 772-779
pISSN: 2085-2495; eISSN: 2477-2712 Oktober 2023
Online pada: http://ojs.unud.ac.id/index.php/buletinvet https://doi.org/10.24843/bulvet.2023.v15.i05.p10
Terakreditasi Nasional Sinta 4, berdasarkan Keputusan Direktur Jenderal Pendidikan Tinggi, Riset, dan Teknologi No. 158/E/KPT/2021
Efisiensi Penggunaan Enzim Taq Polymerase pada Pengujian Polymerase Chain Reaction
(EFFICIENCY OF USING TAQ POLYMERASE ENZYME IN POLYMERASE CHAIN REACTION TESTING)
-
1L
Luh Dewi Anggreni1*, Ni Made Ritha Krisna Dewi2, Ni Putu Sutrisna Dewi2, I Gusti Nngurah Kade Mahardika2
-
1Laboratorium Diagnosa Klinik, Patologi Klinik dan Radiologi Veteriner Fakultas Kedokteran Hewan, Universitas Udayana, Jl. Raya Sesetan, Gg. Markisa No. 6 Denpasar Selatan, Denpasar, Bali, Indonesia;
-
2Laboratorium Biomedik dan Biologi Molekuler Hewan, Fakultas Kedokteran Hewan, Universitas Udayana, Jl. Raya Sesetan, Gg. Markisa No. 6 Denpasar Selatan, Denpasar, Bali,
Indonesia;
*Corresponding author email: dewi.anggreni@unud.ac.id
Abstrak
Polymerase Chain Reaction (PCR) merupakan uji diagnostik yang sangat sensitif. Enzim Taq Polymerase merupakan salah satu komponen paling penting dalam pengujian Polymerase Chain Reaction (PCR). Enzim taq polymerase merupakan komponen yang harganya paling mahal. Penggunaan taq polymerase pada uji PCR kurang efisien. Untuk mengetahui efisiensi penggunaan enzim Taq Polymerase dan pada volume terkecil berapa masih dapat digunakan dalam metode pengujian PCR, maka dilakukanlah penelitian dengan membuat volume PCR lebih kecil (5µl, 10 µl, dan 25 µl) dari standard (50 µl). Tiga isolat swab hidung dan anus anjing terinfeksi Parvovirus diekstraksi menggunakan Kit DNA Purifikasi. Hasil ekstraksi dijadikan DNA Template pada pengujian PCR. Amplifikasi menggunakan mesin PCR Apply Biosistem Thermal Cycler. Visualisasi produk amplifikasi menggunakan elektroforesis gel agarose 1%. Hasil pengujian menunjukan bahwa volume PCR lebih kecil dari standard mampu memvisualisasikan produk PCR dengan baik. Pada PCR volume 5µl, tidak semua sampel teramplifikasi dengan baik. Sampel 1 dan 3 menunjukkan band tebal pada posisi sama dengan kontrol positif. Sedangkan sampel 2 tidak menunjukkan adanya band. Pada volume PCR (10µl, 25 µl dan 50µl) semua sampel teramplifikasi dengan baik. Sampel 1 dan 3 menunjukkan band tebal pada posisi sama dengan kontrol positif. Amplifikasi sampel 1 dan 3, gen memiliki panjang nukleotida 910bp (sesuai oligonukleotida), sedangkan sampel 2 juga menunjukkan adanya band tetapi posisinya lebih rendah dan tipis dari sampel 1, 3 dan kontrol positif. Adanya perbedaan tebal tipisnya band yang teramplifikasi disebabkan oleh perbedaan konsentrasi DNA hasil ekstraksi. Perbedaan tinggi band yang teramplifikasi pada sampel 2, berarti bahwa gen yang teramplifikasi bukan gen spesifik parvo. Simpulan penelitian ini adalah pada volume pengujian PCR dengan volume yang lebih kecil dari standard mampu memvisualisasikan gen DNA virus parvo dengan baik. Untuk mengefisienkan penggunaan enzim taq polymerase pada pengujian PCR, volume PCR 10µl dapat direkomendasikan sebagai protokol pengujian PCR untuk DNA Virus Parvo.
Kata kunci: Band; isolate; PCR; taq polymerase; volume
Abstract
Polymerase Chain Reaction (PCR) is a very sensitive diagnostic test. The Taq Polymerase enzyme is one of the most important components in the Polymerase Chain Reaction (PCR) assay. The taq polymerase enzyme is the most expensive component. The use of taq polymerase in the PCR test is less efficient. To determine the efficiency of using the Taq Polymerase enzyme and at what smallest volume it can still be used in the PCR test method, a study was conducted by making the PCR volume smaller (5µl, 10µl, and 25µl) than the standard (50 µl). Three isolates of nasal and anal swabs of dogs
infected with Parvovirus were extracted using the DNA Purification Kit. The extraction results are used as DNA templates for PCR testing. Amplification using the PCR Apply Biosystem Thermal Cycler machine. Visualization of amplification products using 1% agarose gel electrophoresis. The test results show that the PCR volume is smaller than the standard and is able to visualize the PCR product well. In 5µl volume PCR, not all samples were amplified well. Samples 1 and 3 show a thick band at the same position as the positive control. While sample 2 does not show any bands. At the PCR volume (10µl, 25 l and 50µl) all samples were amplified well. Samples 1 and 3 show a thick band at the same position as the positive control. Amplification of samples 1 and 3, the gene has a nucleotide length of 910bp (according to oligonucleotides), while sample 2 also shows the presence of a band but its position is lower and thinner than samples 1, 3 and positive control. The difference in the thickness of the amplified band was caused by differences in the concentration of the extracted DNA. The difference in amplified band height in sample 2 means that the amplified gene is not a parvo-specific gene. The conclusion of this study is that the volume of PCR testing with a volume that is smaller than the standard is able to visualize the parvo virus DNA gene well. To efficiently use the taq polymerase enzyme in PCR testing, a PCR volume of 10 l can be recommended as a PCR testing protocol for Parvo Virus DNA.
Keywords: Band; isolate; PCR; tag polymerase; volume
PENDAHULUAN
Polymerase Chain Reaction (PCR) merupakan uji diagnostik yang sangat sensitif. Metode PCR adalah suatu metode enzimatis untuk amplifikasi DNA dengan cara in vitro (Ausubel et al., 2003). Proses PCR memerlukan beberapa komponen utama, yaitu DNA template, oligonukleotida primer, Deoksinukleotida trifosfat (dNTP), buffer, dan enzim taq polymerase (Yusuf, 2010). Polymerase Chain Reaction (PCR) melibatkan beberapa tahap yang berulang (siklus). Setiap siklus terjadi duplikasi jumlah target DNA untai ganda, penempelan primer pada template (annealing), pemanjangan primer (extension); dan pemantapan (post-extension) (Handoyono dan Rudiretna, 2001).
Enzim Taq Polymerase merupakan salah satu komponen paling penting dalam pengujian Polymerase Chain Reaction (PCR). Dari semua komponen PCR, enzim taq polymerase merupakan komponen yang harganya paling mahal. Enzim ini bersifat stabil pada suhu diatas 95°C. Namun enzim taq polymerase mempunyai keterbatasan yaitu tidak mampu memperbanyak rantai potongan DNA yang berukuran diatas 3000 pasang basa (3000bp) (Hutapea et al., 2015). Enzim Taq Polymerase berfungsi sebagai katalisis untuk polimerasi DNA. Pada proses PCR
enzim ini diperlukan untuk tahap ekstensi DNA) (Handoyono dan Rudiretna, 2001).
Penggunaan taq polymerase pada uji PCR kurang efisien. Untuk mendapatkan volume PCR (dalam penggunaan enzim taq polymerase) perlu dilakukan dengan cara memvariasikan beberapa volume PCR dengan volume standart dari pabrik. Volume standard penggunaan buffer dan enzim taq polymerase dalam pengujian PCR adalah 50µl. Namun pada visualisasi produk PCR pada gel agarose hanya membutuhkan volume 2-4µl. Oleh karena itu penting melakukan pengujian pada volume yang lebih kecil.
METODE PENELITIAN
Sampel penelitian
Sampel yang telah tekumpul sebanyak 3 buah (S1, S2 dan S3) dari anak anjing umur 3 bulan yang terdiagnosis menderita parvovirus. Sampel yang diambil berupa sampel swab (hidung dan anus) menggunakan cotton swab steril. Sampel kemudian dimasukkan ke dalam microtube yang berisi transport medium M199. Pada saat pengambilan sampel dipilih swab pada hidung dan anus karena sampel yang diambil pada anak anjing yang berumur dibawah enam bulan, dan diketahui bahwa predileksi Virus Parvo ada di bagian pencernaan dan pernafasan (Prittie, 2004). Predileksi virus pada daerah limfoid
menyebabkan deplesi limfosit dan predileksi pada usus menyebabkan nekrosis kripte dan vili-vili usus halus. Pada kondisi ini, anjing menunjukkan gejala klinis demam, muntah, tidak mau makan dan diare akibat peradangan dan tidak berfungsinya vili-vili usus halus (Goddard et al., 2010). Virus CPV pada feses dapat terdeteksi selama 10–14 hari (Sendow, 2006).
Ekstraksi DNA
Ektraksi DNA sesuai dengan panduan manual KIT DNA Purifikasi GeneJet®. Sampel swab yang ada di dalam tube yang berisi transport Medium M199 ditambahkan 400 µl Lysis Solution dan 20µl Proteinase K kemudian di vortek selama 15-30 detik. Setelah itu campuran diinkubasikan pada suhu 56°C selama 30 menit kemudian ditambahkan 20 µl RNase lalu votex selama 15-30 detik kemudian diinkubasikan pada suhu ruangan selama 10 menit. Setelah itu campuran tadi ditambahkan 200µl Lysis Solution dan vortex selama 15-30 menit sampai homogen. Selanjutnya campuran tersebut ditambahkan 400 µl Ethanol 50% dan vortex, kemudian campuran tersebut dimasukkan ke dalam collum collection tube dan disentrifuse selama 1 menit dengan kecepatan 6000rpm. Tube bagian bawah collum dibuang dan diganti dengan tube baru.
Proses pencucian DNA dimulai dengan menambahkan 500µl wash buffer I pada lysate. Catridge disentrifuse selama 1 menit pada kecepatan 8000rpm setelah itu cairan bagian bawah dibuang. Selanjutnya catridge ditambahkan 500µl Wash Buffer II kemudian disentrifuse selama 3 menit pada kecepatan 12000rpm. Selanjutnya proses Eluting DNA dimulai dengan menambahkan 200µl elution buffer pada catridge yang telah dipindahkan pada mikrotube 1,5ml kemudian kemudian diinkubasikan pada suhu ruangan selama 2 menit setelah itu disentrifuse selama 1 menit pada kecepatan 8000rpm. Setelah itu, catridge bisa dibuang. Penyimpanan DNA murni dalam waktu yang lama
dilakukan dalam elution buffer pada suhu -20°C. Sedangkan penyimpanan DNA murni dilakukan pada suhu 4°C untuk penggunaan langsung atau aliquot dengan cara menghindari pengulangan freezing dan thawing dari DNA
Amplifikasi DNA
Amplifikasi menggunakan mesin PCR Apply Biosistem Thermal Cycler. Satu siklus dalam proses amplifikasi meliputi tiga tahapan yaitu denaturasi, annealing primer, dan ekstensi. Gen yang diamplifikasi adalah Gen Virus Parvo dengan sepasang primer sebesar 910bp. Pasangan primer yang digunakan primer forward dengan urutan nukleotida HPRM (5’-CAGGTGATGAATTTGCTACA-3’) (Lin et al., 2014) dan reverse dengan urutan nukleotida VPRM (5’-TTCTAGGTGCTAGTTGAG-3’) (Buonavoglia et al., 2014).
Volume total reaksi amplifikasi bervariasi ada 4 volume yaitu 5 μl, 10 μl, 25 μl dan 50 μl. Untuk volume 5 μl terdiri dari 2,5 μl Go Taq DNA Polymerase Green Master 2x (Buffer pH 8,5, MgCl2 3mM dan masing-masing dNTP 0,4mM), masing-masing primer 0,25 μl, DNA template 1 μl dan ddH2O 2 μl. Untuk volume total reaksi amplifikasi 10μl terdiri dari 5 μl Go Taq DNA Polymerase Green Master 2x (Buffer pH 8,5, MgCl2 3mM dan masing-masing dNTP 0,4mM), masing-masing primer 0,5 μl, DNA template 1 μl dan ddH2O 3 μl. Untuk volume total reaksi amplifikasi 25 μl terdiri dari 12,5 μl Go Taq DNA Polymerase Green Master 2x (Buffer pH 8,5, MgCl2 3mM dan masing-masing dNTP 0,4mM), masing-masing primer 1,2 μl, DNA template 2 μl dan ddH2O 8,1 μl. Untuk volume total reaksi amplifikasi 50 μl terdiri dari 25 μl Go Taq DNA Polymerase Green Master 2x (Buffer pH 8,5, MgCl2 3mM dan masing-masing dNTP 0,4mM), masing-masing primer 2,4 μl, DNA template 3 μl dan ddH2O 16,2 μl.
Kondisi PCR yang digunakan untuk amplifikasi terdiri atas tahap pradenaturasi
pada suhu 95°C selama 7 menit. Tahap selanjutnya 40 siklus dengan kondisi denaturasi pada suhu 94°C selama 45 detik, penempelan primer pada suhu 52°C selama 45 detik dan elongasi/ekstension pada suhu 72°C selama 1 menit. Elongasi/ekstension akhir pad suhu 72°C selama 5 menit. Suhu penyimpanan pada suhu 22°C.
Visualisasi Produk Amplifikasi
Visualisasi produk amplifikasi
dimigrasikan dengan menggunakan teknik elektroforesis gel agarose 1%. Elektroforesis DNA hasil amplifikasi dianalisa lebih lanjut menggunakan elektroforesis untuk dapat melihat apakah sampel yang diperiksa positif atau negatif. Pada prinsipnya adalah DNA yang bermuatan negatif akan berpindah menuju ke arah muatan positif dalam medan listrik. Menurut Seow dan Bahaman (2012) bahwa perpindahan tersebut tergantung pada kondisi buffer, suhu, durasi waktu, konsentrasi gel dan ukuran molekul. Tahapan dalam proses elektroforesis adalah pembuatan gel agarose, elektroforesis, dan pewarnaan.
Proses elektroforesis diawali dengan pembuatan dibuat media elektroforesis berupa gel agarose 1% (melarutkan 1gram agarose dengan 100ml 1X TAE Buffer) kemudian dipanaskan sampai mendidih dan berwarna bening. Tiga mikroliter etidium bromide ditambahkan kedalam larutan. Larutan kemudian dicetak dalam cetakan gel yang telah berisi sisir. Setelah mengeras gel agarose diletakkan ke dalam camber gel elektroforesis yang telah mengandung TAE Buffer 1X. Sepuluh persen (10%) dari volume produk PCR dimasukkan kedalam setiap sumuran. Sumuran pertama pada gel agarose dimasukkan 100bp ladder (Invitrogen®), sedangkan lubang selanjutnya dimasukkan DNA hasil amplifikasi. Mesin Elektroforesis kemudian diberi tegangan 100V selama 30 menit dan 400mA. Visualisasi DNA dilakukan dengan meletakkan gel agarose di atas UV
Transluminator. Setelah UV
Transluminator dinyalakan, pita DNA akan berpendar. Hasil dari sampel dapat dilihat dilayar monitor yang terhubung dengan UV Transluminator. Band atau pita DNA yang muncul pada sampel dibandingkan dengan band yang muncul pada kontrol positif. Hasil positif ditunjukkan dengan adanya band (pita) terang yang sama dengan kontrol positif (Seow dan Bahaman, 2012). Pada penelitian ini sampel yang dinyatakan positif apabila band sampel ada pada ukuran 910bp atau sejajar dengan band pada kontrol positif. Hasil elektroforesis kemudian didokumentasikan
menggunakan kamera digital.
HASIL DAN PEMBAHASAN
Hasil
Hasil ekstraksi selanjutnya dilakukan pengujian dengan metode Polymerase Chain Reaction (PCR) pada volume 5µL. Pada sampel 1 dan sampel 3 menunjukkan adanya pita (band) yang tebal pada posisi yang sama dengan kontrol positif yaitu panjang basa sekitar 910bp (sesuai dengan panjang primer/oligonukleotida yang digunakan). Sedangkan pada sampel 2 menunjukkan pita yang sangat tipis sehingga pada saat didokumentasikan tidak terekam oleh kamera. Hasil pengujian PCR pada Volume 5µL dapat dilihat pada Gambar 1 berikut
Hasil konfirmasi diagnosis dengan uji PCR pada volume 5µl, tidak semua sampel dapat teramplifikasi dengan baik sehingga perlu diuji kembali dengan volume yang lebih besar yaitu volume uji PCR 10µl. Berdasarkan band hasil elektroforesis DNA, terlihat semua sampel (S1, S2, dan S3) terlihat adanya band. Pada sampel 1 dan 3 menunjukkan adanya pita (band) yang tebal pada posisi yang sama dengan kontrol positif. Sampel 1 dan 3 gen yang teramplifikasi memiliki panjang nukleotida yaitu 910bp (sesuai dengan panjang primer/oligonukleotida yang digunakan), sedangkan pada sampel 2 juga menunjukkan posisi pita (band) lebih
rendah dan ukuran band yang lebih tipis dari sampel 1, 3 dan kontrol positifHasil pengujian PCR pada volume 10µl dapat dilihat pada Gambar 2 berikut.
Hasil konfirmasi pengujian PCR volume 25 µl, terlihat semua sampel (S1, S2, dan S3) terlihat menunjukkan adanya band masih stabil sama seperti pada pengujian volume 10µl. Pada sampel 1 dan 3 menunjukkan adanya pita (band) yang tebal pada posisi yang sama dengan kontrol positif. Sampel 1 dan sampel 2 gen yang teramplifikasi memiliki panjang nukleotida yaitu 910bp (sesuai dengan panjang primer/oligonukleotida), sedangkan pada sampel 2 juga menunjukkan posisi pita (band) lebih rendah dan ukuran band yang lebih tipis dari sampel 1, 3 dan kontrol positif. Hasil pengujian PCR pada volume 25µl dapat dilihat pada Gambar 3 berikut.
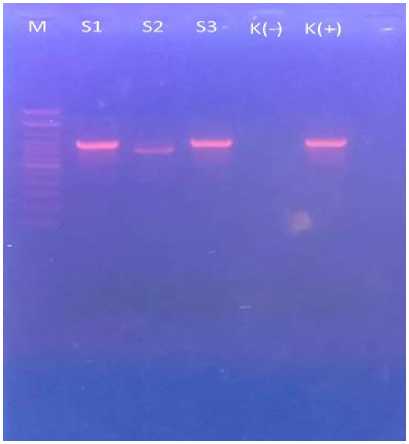
Hasil pengujian PCR pada volume 50µl, hasilnya sama seperti pada pengujian PCR volume 10µl dan 25µl. Pada sampel 1 dan sampel 3 menunjukkan adanya pita (band) yang tebal pada posisi yang sama dengan kontrol positif yaitu panjang basa sekitar 910bp (sesuai dengan panjang primer/oligonukleotida yang digunakan). Sedangkan pada sampel 2 juga menunjukkan adanya pita tetapi lebih tipis dari sampel 1 dan 3. Posisi pita (band) pada sampel 2 lebih rendah dari sampel 1, 3 dan kontrol positifHasil pengujian PCR pada volume 50µl dapat dilihat pada Gambar 4 berikut.
Pembahasan
Hasil pengujian PCR dilakukan pada volume 5µl, tidak semua sampel menunjukkan adanya reaksi positif (band). Berdasarkan band hasil elektroforesis DNA, terlihat bahwa pada sampel 1 dan 3 menunjukkan adanya pita (band) yang tebal pada posisi yang sama dengan kontrol positif. Gen Virus Parvo yang teramplifikasi memiliki panjang nukleotida 910bp (sesuai dengan panjang oligonukleotida). Sedangkan pada sampel 2 menunjukkan pita yang sangat tipis sehingga pada saat didokumentasikan tidak
terekam oleh kamera. Pada sampel 2 tidak teramplifikasi dengan baik pada volume uji PCR 5µl, hal tersebut disebabkan karena konsentrasi DNA template (cetakan) sedikit sehingga tidak teramplifikasi dengan baik. Hasil amplifikasi yang kurang baik dapat disebabkan oleh ketidaksesuaian primer, efisiensi, dan optimasi proses PCR. Primer yang tidak spesifik atau sesuai dapat menyebabkan teramplifikasinya daerah dalam genom yang tidak dijadikan sasaran atau sebaliknya tidak ada daerah genom yang teramplifikasi (Azizah, 2009). Ada beberapa faktor yang sangat mempengaruhi keberhasilan proses elektroforesis diantaranya ukuran molekul DNA, konsentrasi gel agarose, konfirmasi DNA, voltase, keberadaan pewarna DNA, komposisi buffer elektroforesis (Ardhana, 2011). Keberadaan pewarna DNA sangat menentukan tampak atau tidaknya pita DNA saat didokumentasikan dengan UV Transluminar/Geldoc. Pada penelitian ini penulis menggunakan Etidhium Bromide (EtBr) sebagai pewarna. Sesuai dengan pendapat (Wicaksono et al., 2009) bahwa bahwa etidium bromide merupakan sebuah molekul yang dapat mengikat kuat pada DNA. Digunakan untuk memvisualisasi potongan-potongan DNA yang telah dipisahkan pada gel elektroforesis. Etidium mengikat dengan cara menyisip diantara ikatan basa pada untai ganda DNA.
Pada pengujian PCR volume 10 µl, 25µl, dan 50µl, berdasarkan hasil elektroforesis DNA, terlihat semua sampel (S1, S2, dan S3) terlihat adanya band. Pada sampel 1 dan 3 menunjukkan adanya pita (band) yang tebal pada posisi yang sama dengan kontrol positif. Sampel 1 dan 3 gen yang teramplifikasi memiliki panjang nukleotida yaitu 910bp (sesuai dengan panjang primer/oligonukleotida yang digunakan), sedangkan pada sampel 2 juga menunjukkan posisi pita (band) lebih rendah dan ukuran band yang lebih tipis dari sampel 1, 3 dan kontrol positif. Adanya perbedaan tebal tipisnya band
yang teramplifikasi disebabkan oleh perbedaan konsentrasi DNA hasil dari ekstraksi DNA. Pita fragmen yang tipis diasosiasikan dengan kondisi kualitas DNA yang rendah atau template yang sedikit (Satriya et al., 2016). Adanya
perbedaan tinggi band yang teramplifikasi pada sampel 2 (posisi lebih rendah dari kontrol positif), hal tersebut berarti bahwa gen yang teramplifikasi pada uji PCR bukan gen spesifik virus parvo sesuai dengan target primer/oligonukleotida. Primer PCR merupakan oligonukleotida yang berperan untuk mengawali proses amplifikasi molekul DNA (Tri et al., 2011). Untuk membuktikan gen yang teramplifikasi pada sampel 2 perlu dilanjutkan ke tahap sekuensing. Optimasi PCR juga diperlukan untuk menghasilkan karakter yang diinginkan. Optimasi ini menyangkut suhu denaturasi dan annealing DNA dalam mesin PCR. Suhu denaturasi yang rendah dapat menyebabkan belum terbukanya DNA utas ganda sehingga tidak dimungkinkan terjadinya
penempelan primer. Proses penempelan primer pada utas DNA yang sudah terbuka memerlukan suhu optimum, sebab suhu yang terlalu tinggi dapat menyebabkan amplifikasi tidak terjadi karena primer tidak menempel atau sebaliknya suhu yang terlalu rendah menyebabkan primer menempel pada sisi lain genom yang bukan sisi homolognya. Hal ini menyebabkan teramplifikasi banyak daerah tidak spesifik dalam genom tersebut. Suhu penempelan (annealing) ini ditentukan berdasarkan primer (Azizah, 2009).
SIMPULAN DAN SARAN
Simpulan
Adapun simpulan yang dapat diambil pada penelitian ini adalah pada volume pengujian PCR dengan volume yang lebih kecil dari standard yang disarankan oleh produsen masih mampu
memvisualisasikan gen DNA virus parvo dengan baik. Volume PCR 10 µl dapat direkomendasikan sebagai protokol
volume pengujian PCR DNA Virus Parvo. Pada volume 10µl hasilnya sama dan tetap stabil seperti pada pengujian volume 25 µl dan 50µl sesuai dengan standard yang dianjurkan oleh produsen.
Saran
Adapun saran yang dapat diberikan pada penelitian ini adalah perlu dilakukan pengujian PCR pada volume yang sama untuk DNA virus lain.
UCAPAN TERIMAKASIH
Terimakasih yang sebesar besarnya peneliti sampaikan kepada LPPM UNUD yang telah memberikan hibah Pranata Laboratorium dari dana DIPA PNBP Universitas Udayana TA-2022, sesuai dengan Surat Perjanjian Penugasan Pelaksanaan Penelitian Nomor:
B/78.881/UN14.4.A/PT.01.03/2022, tanggal 10 Mei 2022.
DAFTAR PUSTAKA
Ardhana P. 2011. Unjuk kerja aplikasi sistem pendinginan pada alat elektroforesis termoelektrik. Skripsi. Departemen Teknik Mesin Fakultas Teknik Universitas Indonesia, Depok.
Ausubel FM, Brent R, Kingston RE, Moore DD, Seidman JG, Smith JA, Struhl K. 2003. Current protocols in molecular biology. Unites States of America: John Willey and sons. Pp. 7577, 82-83.
Azizah A. 2009. Perbandingan pola pita amplifikasi dna daun, bunga kelapa sawit normal dan abnormal. Institut Pertanian Bogor. Bogor.
Buonavoglia C, Martella V, Pratelli A, Tempesta M, Cavalli A, Buonoglia D, Bozzo G, Elia G, Decaro N,
Charmichael L. 2001. Vidence for
evolution of canin parvovirus type 2 in Italy. J. General Virol. 82: 3021-3025.
Goddard A, Leisewitz AL. 2010. Canine parvovirus. Vet. Clin. North Am. Small Anim. Pract. 40: 1041-1053.
Handoyono D, Rudiretna A. 2001. Prinsip umum dan pelaksanaan polymerase
chain reaction (PCR). Unitas. 9(1): 1729.
Hutapea H, Debbie R, Rahman EG, Rostinawati T. 2015. Teknik long polymerase polymerase chain reaction (LPCR) untuk perbanyakan kerangka baca terbuka gen pengkode polimerase virus Hepatitis B. J. Plasma. 1(2): 4552.
Lin CN, Chien CH, Chiou MT, Chueh LL, Hung MY, Hsu HS. 2014. Genetic characterization of type 2a canine parvoviruses from Taiwan reveals the emergence of an Ile324 mutation in VP2. Virol. J. 11(1): 39.
Prittie J. 2004. Canine parvovirus enteritis: A review of diagnosis,
managemet and prevention. J. Vet. Emerg. Crit. Care. 14: 167-176.
Satriya P, Wirajana N, Suarsa W. 2016.
Amplifikasi fragmen gen 18rRNA pada DNA metagenomik madu dengan teknik PCR. Indon. J. Legal Forensic Sci. 2(3): 45-47.
Sendow I. 2006. Canine parvo virus pada
anjing. Balai Penelitian Veteriner, PO Box 151, Bogor 16114.
Seow VL, Bahaman AR. 2012.
Discriminatory power of agarose gel electrophoresis in DNA fragments analysis. In: Magdeldin S. Gel
Electrophoresis
–
Principles and
Basics. In Tech. Croatia. Pp. 41-56.
Tri J, Nanda K, Sedyo H. 2011. Optimasi metode PCR untuk deteksi pectobacterium carotovorum penyebab penyakit busuk lunak anggrek. J. Perlindungan Tanaman Indon. 17(2): 54–59.
Wicaksono BD, Yohana AH, Enos T, Irawan W, Dina Y, Aldrin N, Ferry S. 2009. Antiproliferative effect of the methanol extract of Piper crocatum ruiz & pav leaves on human breast (T47D) cells In- vitro. Trop. J. Pharm. Res. 8: 345-352.
Yusuf Zuhriana K. 2010. Polymerase Chain Reaction (PCR). J. Saintek. 5(6): 1-6.
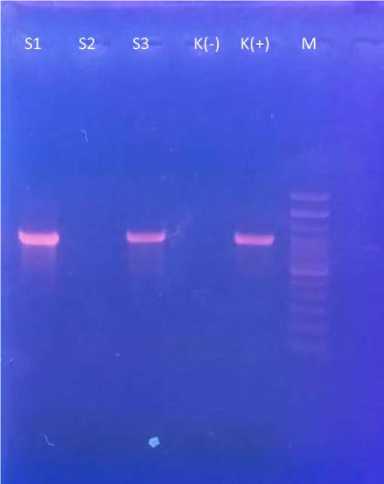
Gambar 1. keterangan dari posisi kiri ke
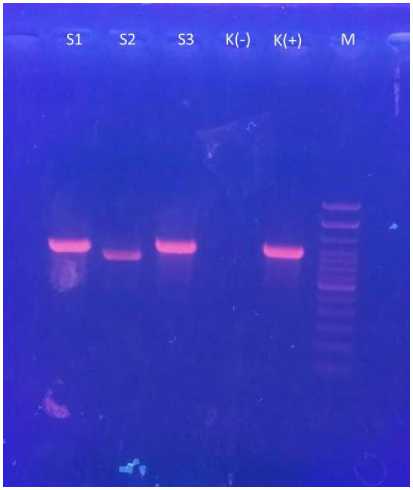
Gambar 2. keterangan dari posisi kiri ke kanan S1: Sampel 1; S2: Sampel 2; S3: Sampel 3; K(-): Kontrol Negatif; K(+):
Kontrol Positif; M: Marker (100bp)
kanan S1: Sampel 1; S2: Sampel 2; S3: Sampel 3; K(-): Kontrol Negatif; K(+): Kontrol Positif; M: Marker (100bp)
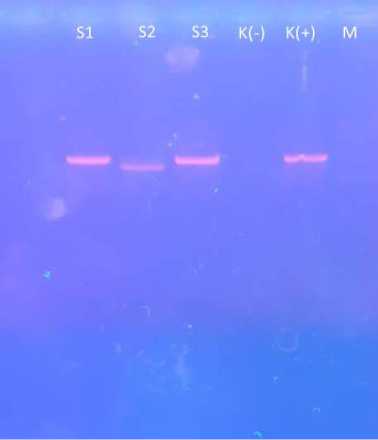
Gambar 3. Keterangan dari posisi kiri ke kanan; M: Marker (100bp Ladder); S1: Sampel 1; S2: Sampel 2; S3: Sampel 3; K(-): Kontrol Negatif; K(+): Kontrol Positif
Gambar 4. Keterangan dari posisi kiri ke kanan; M: Marker (100bp Ladder);S1: Sampel 1; S2: Sampel 2; S3: Sampel 3; K(-): Kontrol Negatif; K(+): Kontrol Positif
779
Discussion and feedback