DETECTION AND SEQUENCING GENES IRON, IUTA, AND HLYF IN AVIAN PATHOGENIC ESCHERICIA COLI
on
Volume 11 No. 2: 229-238
Agustus 2019
DOI: 10.24843/bulvet.2019.v11.i02.p16
Buletin Veteriner Udayana
pISSN: 2085-2495; eISSN: 2477-2712
Online pada: http://ojs.unud.ac.id/index.php/buletinvet
Terakreditasi Nasional Peringkat 3, DJPRP Kementerian Ristekdikti
No. 21/E/KPT/2018, Tanggal 9 Juli 2018
Deteksi dan Sekuensing Gen iroN, iutA, dan hlyF pada Avian Pathogenic Eschericia coli
(DETECTION AND SEQUENCING GENES IRON, IUTA, AND HLYF IN AVIAN PATHOGENIC ESCHERICIA COLI )
Nyoman Anandiya Ramaditya1*, I Nengah Kerta Besung2, I Gusti Ngurah Kade Mahardika2
1PT. Tekad Mandiri Citra, Jl. Siulan Gg. Sekar Sari IV A No. 4B, Penatih, Denpasar Timur, Bali, Indonesia 80238;
2Laboratorium Mikrobiologi Veteriner, Fakultas Kedokteran Hewan Universitas Udayana, Jl. PB. Sudirman Denpasar, Bali, Indonesia 80225;
3Laboratorium Virologi Veteriner, Fakultas Kedokteran Hewan Universitas Udayana, Jl. PB. Sudirman Denpasar, Bali, Indonesia 80225.
*Email: anandiya.ramaditya@gmail.com
ABSTRAK
Penelitian telah dilakukan untuk mendeteksi gen patogenik Avian Pathogenic Eschericia coli (APEC) yaitu gen iroN, iutA, dan hlyF pada bakteri Escherichia coli yang diisolasi dari organ pada ayam yang sakit di Bali, serta untuk mengetahui hubungan filogenetik antara gen penanda patogenik di Bali dengan di negara lain di dunia. Penelitian ini dilakukan secara eksploratif observasional dengan menggunakan enam isolat bakteri Escherichia dengan kode E2, E3, E7, E8, E9, dan E10 yang diisolasi dari ayam buras pada tahun 2018. Semua gen isolat dideteksi dengan metode Polymerase Chain Reaction (PCR). Hasil penelitian menunjukkan semua sampel positif memiliki gen iroN, iutA, dan hlyF. Sekuensing pada ketiga gen diperoleh hasil, bahwa gen ironN yang terbaca dengan baik adalah 659 bp, gen hlyF adalah 518 bp, dan gen iutA adalah 250 bp. Hasil analisis sekuens memiliki DNA yang homolog. Berdasarkan hasil analisis filogentik, bakteri E. coli patogen di Bali berada dalam satu klaster dengan bakteri E. coli patogen yang ada di dunia.
Kata kunci: iroN; iutA; hlyF; Escherichia coli patogen; Bali
ABSTRACT
Research has been carried out to detect pathogenic gene markers of Avian Pathogenic Eschericia coli (APEC) iroN, iutA, and hlyF in Escherichia coli bacteria isolated from organs of sick chickens in Bali, and to determine phylogenetic relationships between those marker genes in Bali and in the other countries in the world. Six isolates of E. coli bacteria with codes E2, E3, E7, E8, E9, and E10 were used in this study. The isolates were isolated from domestic chicken in 2018. All genes were detected using the Polymerase Chain Reaction (PCR) method. The genes of iroN, iutA, and hlyF could be detected from all isolates. Well readable sequence of iroN, hlyF, and iutA was 659 bp, 518 bp, and 250 bp, respectively. All three genes were homogenous. Phylogical analysis shows that all pathogenic markers share same cluster with the pathogenic E. coli from all countries in the world.
Keywords: iroN; iutA; hlyF; pathogenic Escherichia coli; Bali.
PENDAHULUAN
Koliseptisemia adalah penyakit penting yang ditimbulkan oleh strain Avian Pathogenic Eschericia coli (APEC) (Nakazato et al., 2009). APEC merupakan agen infeksius ekstra intestinal yang disebabkan oleh bakteri Eschericia coli (E. coli) pada unggas yang dimiliki oleh
kelompok Extraintestinal Pathogenic E. coli (ExPEC) (Filho et al., 2015). Infeksi koliseptisemia pada unggas terjadi pada usia 4 sampai 9 minggu, dengan mortalitas sebesar 20% (Ewers et al., 2007). Landman dan Eck (2016) menyatakan bahwa data tentang kuantifikasi kerugian karena kolibasilosis pada unggas belum banyak
dilaporkan padahal kerugian akibat penyakit ini dapat dirasakan langsung oleh peternak.
Avian Pathogenic E. coli (APEC) memberikan dampak kerugian besar dalam industri unggas. Pada ayam pedaging, E. coli strain APEC juga menyebabkan selulitis yang ditandai oleh dermatitis nekrotik pada perut dan paha. Lesi tersebut menyebabkan karkas yang terinfeksi harus diafkir (Moulin-Schouleur et al., 2007). Infeksi APEC pada ayam petelur juga menyebabkan penurunan produksi dan kualitas telur. Konsumsi unggas yang kurang matang dapat menginfeksi manusia. APEC dilaporkan dapat menjadi E. coli patogen ekstraintestinal pada manusia (Johnson et al., 2008).
Selama ini, diagnosis E. coli dilakukan dengan beberapa metode konvensional seperti metode biakan (kultur), uji biokimiawi, dan uji biologis. Cara tersebut memerlukan waktu lama dan hasil yang diperoleh tidak spesifik (Sudrajat et al., 2000). Teknik diagnosa dengan teknik biologi molekuler, yaitu dengan cara amplifikasi gen yang spesifik yang terdapat pada genom bakteri E. coli telah terbukti lebih sensitif dan spesifik (Radji et al., 2010).
Kemampuan APEC (Avian Pathogenic E. coli) menyebabkan penyakit tergantung banyak faktor virulensi. Faktor virulensi APEC dikendalikan oleh beberapa gen yang terdapat di dalam plasmid (Ashraf et al., 2013). Menurut penelitian Rodriguez et al. (2005), gen-gen tersebut antara lain cvaC, tsh, sitA, iutA, ompT, dan iroN yang ditemukan pada plasmid ColV. Pada penelitian Johnson et al. (2006) gen yang ditemukan antara lain etsABCD, cluster eitABC, dan hlyF. Penelitian Johnson et al. (2006) juga menyatakan bahwa lebih dari 80% isolat-isolat E. coli yang mempunyai gen iroN, iutA, dan hlyF menyebabkan koliseptisemia pada unggas.
Kasus kolibasilosis pada unggas yang terinfeksi telah banyak dilaporkan di berbagai daerah di Indonesia (Weidosari dan Wahyuwardani, 2015; Hidayati et al.,
2016; Jamin et al., 2015; Wibowo dan Wahyuni, 2008; Poernomo dan Juarini, 1996), namun laporan tentang gen penanda APEC belum pernah dilaporkan di Indonesia. Di Bali, penyakit kolibasilosis ditermukan di berbagai peternakan unggas (Barus et al., 2013). Penelitian ini bertujuan untuk mengidentifikasi dan mendeteksi gen patogenik APEC (iroN, iutA, dan hlyF) pada ayam di Provinsi Bali serta untuk mengetahui hubungan genetik dan filogenetik antara gen penanda patogenik dari APEC di Bali dengan gen penanda patogenik APEC di negara lain.
METODE PENELITIAN
Sampel yang digunakan dalam penelitian ini adalah bakteri E. coli galur patogen penyebab Avian Pathogenic E. coli (APEC) pada ayam. Isolat berasal dari kasus lapangan dan telah dimurnikan di Laboratorium Bakteriologi Fakultas Kedokteran Hewan Universitas Udayana. Berdasarkan hasil isolasi dan identifikasi diperoleh sebanyak enam isolat. Isolat positif memiliki kode isolat E2, E3, E7, E8, E9, E10 yang diisolasi dari ayam buras. Keenam isolat tersebut berasal dari kabupaten Tabanan (E2, E3, E8, E9, E10) dan Badung (E7) pada tahun 2018.
Isolasi DNA dilakukan dengan menggunakan larutan chelex 10% (Walsh et al., 1991). Bakteri E. coli pada media cair diambil sebanyak 200 μl kemudian dipindahkan kedalam microtube yang telah berisi larutan chelex 10%. Larutan chelex 10% yang sudah berisi sampel di vortex selama lima menit, kemudian disentrifugasi selama satu menit. Selanjutnya larutan chelex 10% yang sudah berisi sampel dipanaskan pada heating blok pada suhu 94oC kurang lebih selama 45 menit, setelah itu sampel siap untuk dilakukan amplifikasi DNA dengan PCR.
Reaksi PCR dibuat volume 10 μl dengan komposisi 3 μl dd H2O, 5 μl 2x Taq plus PCR Mastermix TIANGEN®, 0,5 μl primer forward, 0,5 μl primer reverse, dan 1 μl template DNA. Kondisi thermal cycler dengan parameter predenaturasi pada suhu
94oC selama tujuh menit, denaturasi pada suhu 94oC selama satu menit, annealing 50-59oC selama 45 detik, ekstensi 72oC selama satu menit 30 detik, siklus diulang sebanyak 35 kali, dan post ekstensi pada 72oC selama lima menit. Masing-masing
primer dilakukan kalibrasi terlebih dahulu untuk menentukan suhu annealing yang sesuai. Dalam penelitian ini menggunakan tiga pasang primer nukleotida dengan perbedaan suhu annealing sebagai berikut.
Tabel 1. Sekuen nukleotida DNA primer iroN, iutA, dan hlyF
|
No. |
Nama |
Sekuen DNA (5’-3’) |
Target (bp) |
Suhu annealing (oC) |
|
1 |
IroNF |
AAGTCAAAGCAGGGGTTGCCCG |
667 |
59 |
|
IroNR |
GACGCCGACATTAAGACGCAG | |||
|
2 |
IutAF |
AAGTCAAAGCAGGGGTTGCCCG |
302 |
55 |
|
IutAR |
GGCTGGACATCATGGGAACTGG | |||
|
3 |
HlyF |
GGCGATTTAGGCATTCCGATACTC |
599 |
50 |
|
HlyR |
ACGGGGTCGCTAGTTAAGGAG |
DNA hasil amplifikasi divisualisasi dengan melakukan elektroforesis pada gel agarosa 1% sebanyak 0,75g dalam 10X buffer TBE sebanyak 75ml. Proses
Elektroforesis dilakukan pada mesin elektroforesis (BIO-RADTM PowerPac)
pada tegangan 100 V, 400mA, selama 30 menit. Setelah proses elektroforesis selesai, pita-pita fragmen DNA diamati dengan UV transilluminator (BIO-RADTM UVITEC)
untuk mengetahui panjang DNA. Ukuran DNA hasil PCR dibandingkan dengan penanda (ladder) untuk selanjutnya
didokumentasikan menggunakan kamera. Sekuensing DNA
Primer yang digunakan untuk amplifikasi digunakan juga untuk sekuensing. Proses sekuensing DNA dilakukan di PT Genetika Science Indonesia.
Analisis Data
Sekuens nukleotida gen iroN, iutA, dan hlyF bakteri E. coli dari ayam penderita APEC yang diperoleh dari laboratorium bakteriologi Fakultas Kedokteran Hewan Universitas Udayana disepadankan dengan Clustal W dari program MEGA 6.0 (Tamura et al., 2013). Data yang dianalisis adalah konfirmasi hasil sekuensing dengan program Basic Local Alignment Search
Tool (BLAST), analisis filogenetik dengan nukleotida dan asam amino yang tersedia di GenBank. Sekuens gen iroN, iutA, dan hlyF dari berbagai negara diunduh dari GenBank. Hubungan kekerabatan (estimasi jarak genetik dan filogenetik) dianalisis dengan metode Unweighted Pair Group Method with Arithmatic Mean (UPGMA). Jarak evolusi dihitung menggunakan metode Kimura-2. Analisis secara filogenetik dilakukan dengan menggunakan program MEGA 6.0 Software (Tamura et al., 2013).
HASIL DAN PEMBAHASAN
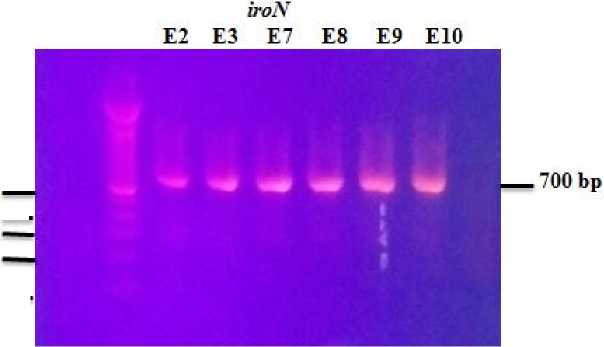
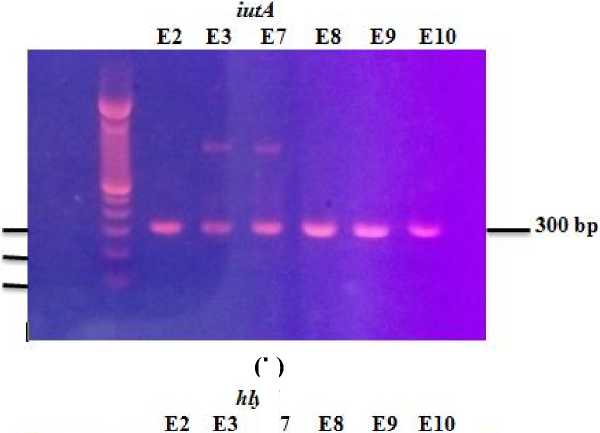
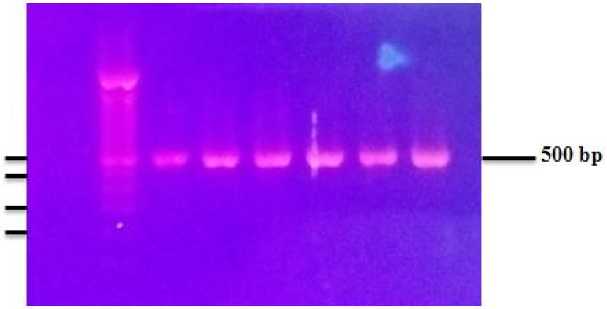
Isolat bakteri E. coli dari laboratotium bakteriologi Fakultas kedokteran Hewan Universitas Udayana berdasarkan hasil isolasi dan identifikasi diperoleh sebanyak enam isolat. Isolat positif memiliki kode isolat E2, E3, E7, E8, E9, E10 yang diisolasi dari ayam buras. Keenam isolat tersebut berasal dari kabupaten Tabanan (E2, E3, E8, E9, E10) dan Badung (E7) pada tahun 2018. Gen iroN, iutA, dan hlyF dapat dideteksi dari semua isolat positif dengan PCR. Gambar 1 merupakan gambar hasil uji PCR dengan menggunakan primer spesifik iroN, iutA, dan hlyF.
700 bp
500 bp__
400 bp
300 bp
IOO bp^—
(a)
300 bp
200 bp
100 bp
(b)
500 bp
400 bp
200 bp IOObp
(c)
Gambar 1. Hasil elektrofesis PCR menggunakan gen iroN, iutA, dan hlyF. Ket: (a) gen iroN, (b) gen iutA, dan (c) gen hlyF
Hasil sekuensing menggunakan ketiga gen yaitu iroN, iutA, dan hlyF. diperoleh hasil gen iroN yang terbaca dengan baik adalah 659 bp, gen iutA adalah 250 bp, dan gen hlyF adalah 518 bp. Hasil tersebut menunjukkan bahwa isolat bakteri E. coli
mempunyai sekuens yang sama pada masing-masing gen (homologi 100%). Sampel E10 asal Kabupaten Tabanan teramplifikasi pada uji PCR, tetapi pada hasil sekuensing tidak teranalisis secara baik untuk itu sampel E10 tidak digunakan
dalam analisis filogenetik. Sampel E10 memiliki sekuen yang sama dengan sampel lainnya. Data sekuens semua gen tersebut telah diregistrasi di Genbank dengan kode akses (accession number) akan dipaparkan berikut ini.
Hasil data sekuens gen iroN, iutA, dan hlyF untuk masimg-masing sampel sudah teregistrasi di Genbank dengan accession number untuk gen iroN MK776774 – MK776779, untuk gen iutA adalah MK776780 – MK 776785, dan untuk gen hlyF adalah MK776769 - MK776773. Data hasil sekuen masing-masing gen digunakan sebagai acuan dalam pengunduhan data sekunder gen bakteri E. coli yang tersedia di GenBank.. Data sekunder yang diunduh dari GenBank, dikelompokan menggunakan metode neighbor-joining dengan metode bootstrap (500 pengulangan). Data sekunder diseleksi dengan cara mengambil satu negara dengan tahun isolasi terbaru dan terlama pada masing-masing kelompok, serta bakteri lainnya yang memiliki hubungan kekerabatan terdekat dengan masing-masing gen yang dimiliki oleh E. coli.
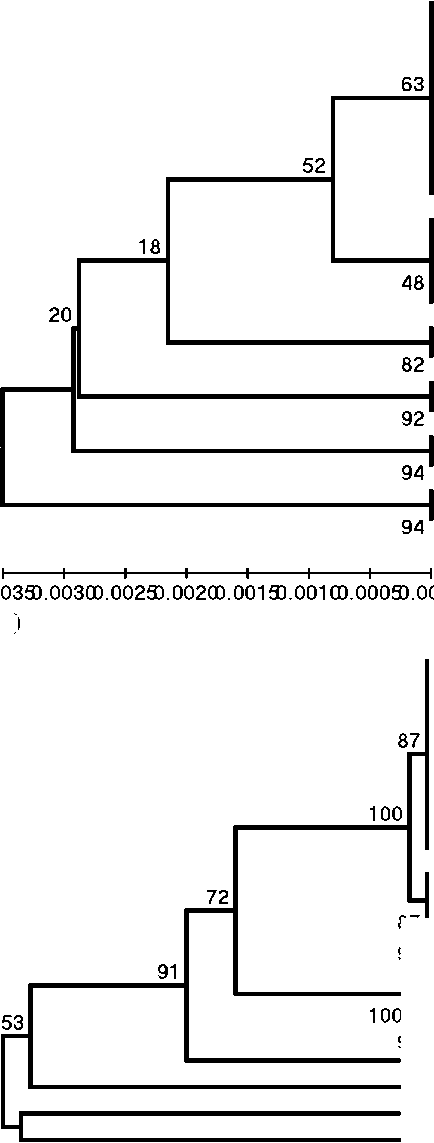
Hasil data setelah pengelompokan, dikelompokan kembali ke dalam beberapa grup dan data diluar kelompok (outgroup). Data hasil pengeompokan secara grup diseleksi secara acak untuk masing-masing grup, pengecualian dilakukan terhadap gen iroN yang berasal dari spesies selain Escherichia coli untuk tetap dipertahankan. Tahapan selanjutnya data dianalisis filogenik kembali dengan menggunakan Neighbor-joining tree dengan metode bootstrap (500 pengulangan). Hasil berupa pohon filogenik dari masing-masing gen ditampilkan pada gambar 2.
Laporan tentang gen penanda APEC belum pernah dilaporkan di Bali ataupun di Indonesia. Pada penelitian ini keenam sampel isolat bakteri E. coli yang diisolasi dari ayam buras yaitu E2, E3, E8, E9, dan E10 asal Kabupaten Tabanan dan E7 asal Kabupaten Badung diisolasi menggunakan
metode cheelex. Sampel E2, E3, E8, E9, dan E10 asal Kabupaten Tabanan dan E7 asal Kabupaten Badung positif memiliki gen iroN, iutA, dan hlyF. Hasil ini membuktikan bahwa isolat lapangan yang diuji menunjukkan bakteri yang patogen dan dapat menimbulkan infeksi pada hewan. Penelitian Johnson et al. (2006) menyatakan bahwa lebih dari 80% isolat-isolat E. coli yang memiliki gen iroN, iutA, dan hlyF berperan dalam koliseptisemia pada unggas. Vandekerchove et al. (2005) juga menyatakan bahwa gen iutA merupakan gen patogenik penyebab APEC. Pada penelitian Ashraf et al. (2013) juga dikatakan bahwa gen iutA dapat ditemukan pada 80% unggas penderita APEC. Penelitian Filho et al. (2015) menyatakan bahwa dari 994 sampel APEC yang diuji, sebesar 83% disebabkan oleh gen iroN dan hlyF.
Pada Penelitian ini ketiga gen yaitu iroN, iutA, dan hlyF berhasil diamplifikasi menggunakan DNA primer yang telah dipublikasikan sebelumnya, yaitu untuk gen iroN pada penelitian Mellata et al. (2009), gen iutA pada penelitian Johnson et al. (2006), sedangkan gen hlyF pada
penelitian (Morales et al., 2004). Hasil sekuensing pada ketiga gen yaitu iroN, iutA, dan hlyF diperoleh hasil gen iron yang terbaca dengan baik adalah 659 bp, gen hlyF adalah 518 bp, dan gen iutA adalah 250 bp.
Penelitian Vidotto et al. (1991) dan Johnson et al. (2006) menyatakan bahwa gen iroN merupakan gen patogen pada APEC yang berperan dalam reseptor siderophore aerobactin untuk proses penyadapan zat besi pada protein sel inang, dan gen iroN juga berperan dalam mekanisme transfer senyawa di dalam sel. Gen iutA merupakan gen patogen pada strain APEC yang berperan dalam reseptor siderophore aerobactin ferric (Fe3+) dalam proses penyadapan zat besi pada protein sel inang (Rodriguez et al., 2005).
E9 Tabanan Bali iroN
E10 Tabanan Bali iroN
MF174860.1 Australia 2017
E2 Tabanan Bali iroN
E7 Badung Bali iroN
MF474175.1 Hongkong 2017
E3 Tabanan Bali iroN
E8 Tabanan Bali iroN
CP032832.1 Klebsiella pneumoniae Australia 2018
CP019777.1 Singapore 1986
CP026786.1 Shigella dysenteriae USA 2003
CP022731.1 Saudi Arabia 2012
HG428755.1 Belgium 2013
CP002090.1 Salmonella enterica USA 1989
CP031256.1 USA 2018
CP022686.1 Germany 1917
CP025251.1 Brazil 1974
CP016358.1 Switzerland 2016
LT601384.1 UK 2016
CP019778.1 Germany 1885
(a)
E8 Tabanan Bali iutA
LT599825 Germany 2016
E10 Tabanan Bali iutA
E2 Tabanan Bali iutA
E9 Tabanan Bali iutA
E3 Tabanan Bali iutA
E7 Badung Bali iutA
CP033635 China 2016
MH422552 Spain 2015
MH753063 Malaysia 2018
CP022069 S.enterica USA 2011
CP023061 South Korea 2016
CP034067 Shigella sonnei USA 2015
CP014092 USA 2018
AP018808 Japan 2014
LR134204 Citrobacter koseri United Kingdom 2018
CP009459 Cedecea neteri Malaysia 2013
LR134138 Kluyvera intermedia United Kingdom 2018
CP027260 Pectobacterium parmentieri Poland 2013
0.10 0.08 0.06 0.04 0.02 0.00
(b)
E8 Tabanan Bali hlyF
E3 Tabanan Bali hlyF
LS992167 Germany 2018
CP002090 Salmonella enterica USA 2010
E2 Tabanan Bali hlyF
E7 Badung Bali hlyF
E9 Tabanan Bali hlyF
KY007017 Australia 2016
LT985293 France 2018
CP034739 China 2018
AY545598.5 USA 2005
MG825372 China 2018
CP030791 USA 1982
CP031107 China 2018
MG649031 France 2017
-------------1--------------------1--------------------1--------------------1--------------------1
0.0020 0.0015 0.0010 0.0005 0.0000
(c)
Gambar 2. Hasil analisis filogenik gen iroN (a), gen iutA (b), dan gen hlyF (c)
Ket: Analisis filogenetik menggunakan metode UPGMA untuk data primer sekuens masing-masing gen dengan data gen iroN, iutA, dan hlyF dari berbagai negara di dunia yang telah diseleksi secara acak. Data dianalisis dengan metode bootstrap sebanyak 500 pengulangan, dimana presentase ulangan ditampilkan di sebelah cabang. Pohon ditarik sesuai skala dalam satuan yang sama untuk mengetahui jarak evolusi pada pohon filogenetik. Jarak evolusi dihitung dengan menggunakan metode Kimura-2 dan berada dalam satuan jumlah substitusi dasar per situs. Analisis evolusi dilakukan dalam MEGA 6.
Gen hlyF merupakan gen patogen pada strain APEC yang berperan dalam pembentukan membran luar pada bakteri E. coli, penyusunan protein hemolysin F yang berfungsi melisiskan sel darah merah inang (Rodriguez et al., 2005).
Hasil Analisis Sekuens E2, E3, E8, E9, E10 asal Kabupaten Tabanan dan E7 asal Kabupaten Badung positif memiliki homologi DNA 100% untuk masing-masing gen yaitu iroN, iutA, dan hlyF. Hasil ini menunjukkan bahwa APEC yang bersirkulasi di Bali berasal dari satu asal usul yang sama, hal ini disebabkan karena DNA cenderung stabil. Denamur dan Matic (2006) menyatakan bahwa tingkat mutasi DNA makhluk hidup diperkirakan 1 kali 10-6.
Pada penelitian ini, enam isolat yang dipelajari positif gen iroN, iutA, dan hlyF. Pada hasil analisis filogenetik dan situs
polimorfik, gen iroN memiliki homologi dengan gen iroN MF174860 asal Australia (2017) dan MF474175 asal Hongkong (2017). Gen iutA memiliki homologi dengan gen iutA CP033635 asal China (2016) dan LT599825 asal Jerman (2016). Gen hlyF memiliki homologi dengan gen hlyF LS992167 asal Jerman (2016) dan KY007017 asal Australia (2016). Berdasarkan pernyataan tersebut, bakteri E. coli patogen di bali berasal dari satu cluster yang sama dengan bakteri E. coli patogen yang ada di dunia.
Pada hasil analisis filogenetik gen iroN, iutA, dan hlyF juga ditemukan pada spesies bakteri lainnya. Hal ini dapat terjadi karena bakteri E. coli mampu melakukan pertukaran materi genetiknya yang diperantarai oleh plasmid ke bakteri E. coli atau spesies bakteri lainnya secara horizontal melalui proses konjugasi,
trasformasi, dan transduksi (Ochman et al., 2000). Proses tersebut mengakibatkan gen patogenik dari bakteri E. coli dapat berpindah ke spesies bakteri lainnya.
Penelitian ini selain mengisolasi bakteri E. coli asal organ, juga melakukan uji biologi molekuler pada sampel feses ayam terinfeksi. Sampel feses ayam buras berasal dari desa Buruan Kabupaten Tabanan. Isolasi dilakukan untuk mengetahui apakah gen patogenik iroN, iutA, dan hlyF juga dapat diisolasi dari feses ayam yang terinfeksi APEC. Pada Hasil PCR beberapa sampel menunjukan hasil postif memiliki gen iroN, iutA, dan hlyF, tetapi pada Isolat E. coli 28 menunjukan hasil negatif terhadap gen hlyF.
Penelitian ini bertujuan untuk mendeteksi gen penanda APEC dapat digunakan dalam penanda patogenik yang tepat untuk diagnosis APEC pada ayam di Bali dan di Indonesia. Gen penanda patogenik APEC yaitu iroN, iutA, dan hlyF berhasil ditemukan dari hasil isolasi organ ayam di Bali (Tabanan dan Badung). Penemuan gen patogen tersebut diharapkan dapat mempermudah dalam diagnosis, tindakan pengobatan, dan pencegahan pada kasus APEC di Bali.
SIMPULAN
Bakteri E. coli yang diisolasi dari organ ayam di Bali (Tabanan dan Badung) memiliki gen patogenik iroN, iutA, dan hlyF. Isolat bakteri E. coli di Bali (Tabanan dan Badung) mempunyai sekuens yang sama pada masing-masing gen dan satu cluster yang sama dengan bakteri E. coli patogen yang ada di dunia.
SARAN
Penelitian lanjutan terhadap deteksi gen penanda patogenik APEC di seluruh kabupaten di Bali ataupun di seluruh Indonesia harus terus dilakukan, sehingga informasi mengenai gen penanda patogenik lainnya dapat diketahui. Uji biologis terhadap gen patogen iroN, iutA, dan hlyF juga perlu dilakuka sebagai uji prosedur
dasar untuk membuktikan patogenitas suatu agen penyakit.
UCAPAN TERIMAKASIH
Melalui kesempatan ini penulis mengucapkan terimakasih yang sebesar-besarnya kepada Dekan Fakultas Kedokteran Hewan Universitas Udayana, dan dosen pembimbing atas fasilitas, arahan dan kesempatan yang diberikan. Penulis juga mengucapkan terimakasih kepada Penulis juga mengucapkan terimakasih kepada The Professor and Promotion Project Universitas Udayana (DIPA-PNPB Tahun 2018, 383-
1/UN14.4A/LT/2018) yang telah
memeberikan dukungan dalam
berlangsungnya penelitian ini.
DAFTAR PUSTAKA
Ashraf HM, Ghanem IA, Eid AM, Sherwood JS, Li G, Nolan LK, Logue CM. 2013. Molecular and phenotypic characterization of Escherichia coli isolated from broiler chicken flocks in Egypt. Avian Dis. 57(3): 602-611.
Barus DO, Gelgel KTP, Suarjana IGK.
2013. Uji kepekaan bakteri Escherichia coli asal ayam pedaging terhadap antibiotik Doksisiklin, Gentamisin, dan Tiamfenikol. Indo. Med. Vet. 2(5): 538545.
Denamur E, Matic I. 2006. Micro review: Evolution of mutation rates in bacteria. Mol. Microbiol. 60(4): 820-827.
Ewers C, Wilking H, Kiessling S, Antáo EM, Laturnus C, Diehl I, Glodde S, Homeier T, Böhnke U, Steinrück H, Philipp HC. 2007. Avian pathogenic, uropathogenic, and newborn
meningitis-causing Escherichia coli: how closely related are they. Int. J. Med. Microbiol. 297: 163-176
Filho HCK, Carvalho D, Grassotti TT, Soares BD, Rossato JM, Cunha AC, Cavalli LS, Brito KCT, Brito BG. 2015. Avian pathogenic Escherichia coli – methods improved diagnosis. World’s Poult. Sci. J. 71: 249-258.
Hidayati SC, Darmawi, Rosmaidar, Armansyah T, Dewi M, Jamin F, Fakhrurrazi. 2016. Pertumbuhan Escherichia Coli yang diisolasi dari feses anak ayam broiler terhadap ekstrak daun salam (Syzygium
polyanthum). J. Med. Vet. 10(2): 101104.
Jamin F, Abrar M, Dewi M, Yanrivina SVS, Fakhrurrazi, Manaf ZF, Syafruddin. 2015. Infeksi bakteri Escherichia Coli pada anak ayam kampung (Gallus domesticus) di Pasar Lambaro Aceh Besar. J. Med. Vet. 9(1): 54-56.
Johnson TJ, Siek KE, Johnson SJ, Nolan LK. 2006. DNA sequence of a CoIV plasmid and prevalence of selected plasmid-encoded virulence genes among avian Escherichia coli strains. J. Bacteriol. 188(2): 745-758.
Johnson TJ, Wannemuehler Y, Doetkott C, Johnson SJ, Rosenberg SC, Nolan LK. 2008. Identification of minimal predictors of avian pathogenic Escherichia coli virulence for use as a rapid diagnostic tool. J. Clin. Microbiol. 46(12): 3987-3996.
Landman WJM, Eck JHH. 2016. The incidence and economic impact of the Escherichia coli peritonitis syndrome in Dutch poultry farming. Avian Pathol. 44(5): 370-378.
Lynne AM, Kariyawasam S,
Wannemuehler Y, Johnson TJ, Johnson SJ, Sinha AS, Lynner DK, Moon HW, Jordan DW, Logue CM, Foley SL, Nolan LK. 2012. Recombinant iss as a Potential Vaccine for Avian Colibacillosis. Avian Disease. 56: 192199
Mellata M. 2009. Full sequence and comparative analysis of the plasmid pAPEC-1 of avian pathogenic E. coli x7122 (O78:K80:H9). Plos One. 4(1): 1-12
Morales C, Lee MD, HofacreC, Maurer JJ. 2004. Detection of A novel virulence gene and a Salmonella virulence
homologue among Escherichia coli isolated from broiler chickens. Foodborne Pathog. Dis. 1(3): 160-165.
Moulin-Schouleur M, Répérant M, Laurent S, Brée A, Mignon-Grasteau S, Germon P, Rasschaert D, Schouler C. 2007. Extraintestinal pathogenic Escherichia coli strains of avian and human origin: Link between phylogenetic
relationships and common virulence patterns. J. Clin. Microbiol. 45: 33663376.
Nakazato G, Tatiana AC, Eliana GS, Brocchi M, Silveira WD. 2009. Virulence factors of avian pathogenic Escherichia coli (APEC). Pesq. Vet. Bras. 29(7): 479-486.
Radji M, Puspaningrum A, Sumiati A. 2010. Deteksi cepat bakteri Escherichia Coli dalam sampel air dengan metode Polymerase Chain Reaction
menggunakan primer 16e1 dan 16e2. Makara. Sains. 14(1): 39-43.
Rodriguez KE, Giddings CW, Doetkott C, Johnson TJ, Nolan LK. 2005. Characterizing the APEC pathotype. Vet. Res. 36: 241–256.
Sudrajat D, Maria LR, Suhadi F. 2000. Deteksi cepat bakteri Escherichia coli enterohemoragik (Ehek) dengan metode PCR (Polymerase Chain Reaction). Risalah Pertemuan Ilmiah Penelitian dan Pengembangan
Teknologi Isotop dan Radiasi. Jakarta
Sutrisno IK, Arundina I, Sosiawan A. 2013. Identifikasi bite marks dengan ekstraksi DNA metode chelex. Dental J. 46(2): 107-112.
Tabrah FL. 2011. Koch’s postulates, carnivorous cows, and tuberculosis today. Hawai. Med. J. 70(7): 144-148.
Tamura K, Stecher G, Peterson D, Filipski A, Kumar S. 2013. MEGA6: Molecular evolutionary genetics analysis version 6.0. Mol. Biol. Evol. 30: 2725-2729.
Vidotto M, Cacao CJM, Goes CR, Santos DS. 1991. Plasmid coding for aerobactin production and drug resistance is involved in virulence of
Escherichia coli avian strains. J. Med. Biol. Res. 24: 677–685.
Walsh PS, Metzger DA, Higuchi. 1991. Chelex-100 as a medium for simple extraction of DNA for PCR based typing from forensic material. Biotechniques. 10: 506-513.
Weidosari E, Wahyuwardani S. 2015. Studi kasus penyakit ayam pedaging di Kabupaten Sukabumi dan Bogor. J. Ked. Hewan. 9(1): 9-13.
Wibowo MH, Wahyuni AETH. 2008. Studi patogenesitas Escherichia coli isolat unggas pada ayam pedaging umur 15 hari. J. Vet. 9(2): 87-93.
238
Discussion and feedback