Optimasi digesti enzim restriksi untuk deteksi mutasi daerah D-loop DNA mitokondria dengan metode PCR-RFLP
on
JURNAL BIOLOGI UDAYANA
P-ISSN: 1410-5292 E-ISSN: 2599-2856
Volume 27 | Nomor 1 | Juni 2023
DOI: https://doi.org/10.24843/JBIOUNUD.2023.v27.i01.p07
Optimasi digesti enzim restriksi untuk deteksi mutasi daerah D-loop DNA mitokondria dengan metode PCR-RFLP
Digestion optimization of restricted enzyme for mutation detection of mitochondrial DNA
D-loop region by PCR-RFLP Method
Ni Putu Senshi Septiasari1*, I Ketut Junitha2, Ni Nyoman Wirasiti2
-
1) Program Studi Diploma IV Teknologi Laboratorium Medis, Fakultas Ilmu-ilmu Kesehatan, Universitas Bali Internasional, Jl. Seroja Gg. Jeruk, Tonja, Denpasar Utara, Denpasar, Bali, Indonesia-80234
-
2) Program Studi Biologi, Fakultas Matematika dan Ilmu Pengetahuan Alam, Universitas Udayana,
Jl.Raya Kampus UNUD, Bukit Jimbaran, Kuta Selatan, Badung, Bali, Indonesia-80361
*Email: senshiseptia@iikmpbali.ac.id
Diterima
19 November 2022
INTISARI
Disetujui
9 April 2023
Metode PCR-RFLP merupakan salah satu metode untuk deteksi mutasi pada daerah D-loop DNA mitokondria. Metode ini menggunakan enzim restriksi untuk dapat memotong DNA mitokondria dan menghasilkan ukuran fragmen DNA yang berbeda-beda. Enzim restriksi memerlukan kondisi yang optimal untuk melakukan pemotongan DNA. Penelitian ini bertujuan untuk mengetahui kondisi optimal enzim restriksi agar dapat melakukan digesti pada daerah D-loop DNA mitokondria. Optimasi dilakukan dengan membuat dua kombinasi formula digesti (formula 1 dan 2) dan empat macam waktu digesti (2 jam, 4 jam, 6 jam dan overnight). Hasil penelitian menunjukkan optimasi dari lima macam enzim restriksi (HaeIII (BsuRI), HindIII, HinfI, MboI dan HpyP31 (DdeI)) didapatkan bahwa ada perbedaan formula dan waktu digesti tergantung dari jenis enzim. Enzim HaeIII(BsuRI), HinfI dan MboI menunjukkan formula 2 merupakan formula optimal, sedangkan enzim HpyP31 (DdeI) formula 1 merupakan formula yang optimal. Waktu digesti 2 jam menunjukkan hasil optimal pada Enzim HaeIII(BsuRI), MboI dan HpyP31 (DdeI), sedangkan enzim HinfI waktu digesti optimal adalah 4 jam. Enzim HindIII tidak mendapatkan hasil potongan fragmen DNA setelah digesti, maka enzim HindIII tidak memiliki situs pemotongan pada daerah D-loop DNA mitokondria.
Kata kunci: optimasi digesti, PCR-RFLP, enzim restriksi, mutasi, D-loop region
ABSTRACT
The PCR-RFLP is a method for detecting mutations in the D-loop region of mitochondrial DNA. This method uses restriction enzymes to digestion mitochondrial DNA and produce DNA fragments of different sizes. Restriction enzymes require optimal conditions for DNA cutting.This study aims to determine the optimal conditions for restriction enzymes to perform digestion in the D-loop region of mitochondrial DNA. Optimization was carried out by making two combinations of digestion formulas (formula 1 and 2) and four different digestion times (2 hours, 4 hours, 6 hours and overnight). The optimization results of five types of restriction enzymes (HaeIII /BsuRI, HindIII, HinfI, MboI and HpyP31 /DdeI) showed that there were different formulas and digestion times depending on the type of enzyme. HaeIII (BsuRI), HinfI and MboI enzymes showed the optimal formula 2, while the HpyP31 (DdeI) enzyme formula 1 was the optimal formula. Digestion time of 2 hours showed optimal results for HaeIII (BsuRI), MboI and HpyP31 (DdeI) enzymes, while the HinfI enzyme digested 4 hours. The HindIII enzyme did not have a cut DNA fragment after digestion, so the HindIII enzyme did not have a cutting site in the D-loop region of mitochondrial DNA.
Keywords: digestion optimization, PCR-RFLP, restriction enzyme, mutation, D-loop region
PENDAHULUAN
Tes DNA merupakan metode untuk identifikasi personal secara molekuler. Metode ini tepat digunakan dalam menangani kasus-kasus forensik dan identifikasi korban secara masal (Yudianto, 2020) serta dapat digunakan untuk tes paternitas (Junitha, 2015). Keunggulan identifikasi secara molekuler adalah mempergunakan sampel yang sedikit mampu mengidentifikasi dengan cepat dan akurat. Perbedaan profil DNA ditentukan oleh adanya lokus polimorfis sebagai sumber untuk identifikasi genetik manusia (Mastura, 2007). Penanda yang umum digunakan untuk tes DNA terdiri dari DNA autosomal, DNA kromosom-Y dan DNA mitokondria (mtDNA). Penggunaan penanda DNA tergantung pada jenis sampel yang ditemukan di TKP (Doosti & Payam, 2011).
Penanda DNA mitokondria merupakan salah satu penanda DNA yang dapat digunakan untuk tes DNA. Penanda DNA mitokondria memiliki beberapa kelebihan yaitu, DNA mitokondria diturunkan secara maternal oleh ibu kepada anaknya baik laki-laki maupun perempuan sehingga identitas DNA korban yang tidak memiliki referensi pembanding antara kedua keluarga dapat menggunakan penanda DNA mitokondria dari saudara yang satu keturunan ibu. Kelebihan lain DNA mitokondria memiliki struktur berbentuk sirkuler yang membuat DNA mitokondria lebih stabil serta resisten terhadap bahaya degradasi sehingga sampel tulang dan rambut yang berumur tahunan cocok menggunakan penanda DNA mitokondria (Yudianto, 2020). DNA mitokondria memiliki jumlah salinan 1000-10000 copy maka sampel dalam jumlah sedikit akan dapat dianalisis. Selain itu, tingkat mutasi DNA mitokondria 5-10 kali lebih cepat dibandingkan DNA autosomal. DNA mitokondria (mtDNA) manusia memiliki tingkat polimorfisme yang lebih tinggi dibandingkan DNA inti, terutama pada daerah D-Loop yang merupakan daerah non coding (Moore & Isenberg, 1999; Mastura, 2005; Luftig et al., 2001).
Metode PCR-RFLP (Polymerase Chain Reaction - Restricted Fragment Length Polymorphism) merupakan metode yang dapat mendeteksi mutasi dan polimorfisme dalam studi evolusi manusia (Siti et al., 2013). Metode ini melakukan penggandaan DNA pada bagian gen yang spesifik dan memotong untai DNA dalam pola tertentu dengan menggunakan enzim endonuclease dan akan dihasilkan fragmen yang panjangnya berbeda-beda (Andrew, 2013). Enzim restriksi memotong molekul DNA dengan memutuskan ikatan fosfodiester antara nukleotida satu dengan nukleotida lainnya. Enzim-enzim ini bekerja dengan memotong DNA pada lokasi yang spesifik mengenali daerah atau urutan DNA pendek dalam molekul DNA yang bersifat palindrome (Kusumawati, 2011).
Enzim restriksi memerlukan kondisi optimal untuk dapat memotong urutan DNA secara sempurna. Kondisi optimal meliputi pH buffer, suhu digesti dan waktu digesti (Putri et al., 2017). Penelitian ini bertujuan untuk memperoleh informasi tentang formula buffer serta waktu digesti yang tepat agar enzim dapat melakukan digesti secara optimal dan menghasilkan fragmen DNA mitokondria yang polimorfis dengan metode PCR-RFLP.
MATERI DAN METODE
Bahan dan alat
Bahan-bahan penelitian yang dibutuhkan antara lain sel epitel mukosa mulut yang berasal dari 2 probandus, masker, sarung tangan, cotton bud steril, lysis buffer DPZ (Deutsche Primaten Zentrum), Sodium Dodecyl Sulfat (SDS)
10%, 1X STE (Sodium Chloride-Tris-EDTA), fenol, Chloroform Isoamyl Alcohol (CIAA) 24 : 1, etanol absolute, etanol 70%, TE (Tris-EDTA), Green Master Mix PCR (Promega), Primer D-loop mtDNA, enzim restriksi (BsuRI/HaeIII, HindIII, HinfI, MboI dan HpyF3I/DdeI (Thermo Scientific), ddH2O, Thermo Scientific GeneRuler 100 bp DNA Ladder, Gel Agarose (Thermo Scientific), 10X Tris-Boric acid-EDTA (TBE) buffer, GelRed (Biotium), dan aquadest.
Instrumen dalam penelitian ini yaitu tabung 1,5 ml, tabung 200 µl, mikro pipet, tip mikro (10-1000 µl), alat sentrifugasi, freezer, Thermal Cycle/Mesin PCR (Veriti), GelDoc (Bio-Rad), alat sterilisasi (autoclave), mesin elektroforesis horisontal (Bio-Rad), shaker rotator, microwave, gelas ukur, timbangan analitik, Erlenmeyer, spatula, gelas beker (beaker glass), inkubator, waterbath, kertas Semi-Log, penggaris, millimeter block, dan kamera.
Metode
Isolasi DNA dari Swab Mukosa dan Amplifikasi DNA
Pengambilan sampel telah mendapatkan kelaikan etik dari Unit Penelitian dan Pengembangan Fakultas Kedokteran Universitas Udayana/RSUP Sanglah Denpasar. Sampel diambil dengan metode non-invasive sampling. Sampel berupa sel epitel mukosa mulut diambil dengan menggunakan cotton bud steril yang berisi lysis buffer DPZ (Deutsche Primaten Zentrum). Proses ekstraksi DNA menggunakan metode fenol kloroform yang dimodifikasi (Junitha et al., 2012). Sampel DNA diamplifikasi dengan primer L16159F (5`-TACTTGACCACCTGTAGTAC-3`) dan H408R (5`-CTGTTAAAA GTGCATACCGCCA-3`) (Candramila, 2002) .
Formula PCR adalah sebagai berikut: 2X Green Master Mix sebanyak 17,5 µl, primer F dan R masing-masing 1,4 µl, H2O sebanyak 11,9 µl, dan 2,8 µl template DNA. Program PCR adalah sebagai berikut: predenaturasi (94oC) selama 5 menit, denaturasi (94oC) selama 1 menit, annealing (52oC) selama 1 menit dan 1 menit untuk extension (72oC), siklus tersebut diulang sebanyak 35x serta siklus terakhir adalah final extension (72oC) selama 10 menit. Hasil PCR divisualisasi dengan menggunakan gel agarose 1 % dalam buffer TBE 1X dengan voltase 50V selama 1 jam.
Optimasi Enzim Restriksi
Produk PCR didigesti dengan lima macam enzim restriksi yaitu HaeIII (BsuRI), HindIII, HinfI, MboI dan HpyP31 (DdeI). Optimasi enzim yang dilakukan dengan membandingkan penggunaan buffer tango dan buffer R (Tabel 1). Masing-masing formula akan dioptimasi pada empat varian waktu digesti yaitu 2 jam, 4 jam, 6 jam dan overnight.
Tabel 1. Formula Digesti Enzim Restriksi
|
Komposisi Reagen Digesti |
Formula 1 |
Formula 2 |
|
Produk PCR |
5 µl |
5 µl |
|
Enzim Restriksi |
0,5 µl |
0,5 µl |
|
Buffer Tango/R |
1 µl* |
1 µl |
|
H20 |
8,5 µl |
8,5 µl |
|
Total Volume |
15 µl |
15 µl |
Keterangan: * Buffer Tango; ** Buffer R
HASIL DAN PEMBAHASAN
Amplifikasi Daerah D-loop DNA Mitokondria
Amplifikasi daerah D-loop DNA mitokondria dengan metode PCR menghasilkan satu fragmen DNA dengan panjang 860 bp (Gambar 1). Amplifikasi DNA pada daerah D-loop mitokondria telah berhasil dilakukan pada DNA dari sampel epitel mukosa mulut. Gambar 1 menunjukkan pita DNA yang tebal dengan ukuran 860 bp. Hal serupa juga dilaporkan oleh Candramila (2002), yang menunjukkan pita DNA dari primer D-loop DNA mitokondria pada masyarakat Betawi memiliki panjang pita DNA yang sama yaitu 860 bp. Pita DNA yang dihasilkan tebal sehingga mampu dilanjutkan untuk tahapan digesti enzim restriksi. Ketebalan pita menunjukkan jumlah amplifikasi atau konsentrasi DNA. Semakin tebal pita DNA yang teramplifikasi maka konsentrasi DNA juga akan tinggi. Konsentrasi DNA yang baik digunakan untuk digesti yaitu 100 ng/µL dan memiliki kemurnian 1,8-2,0 (Fazri et al., 2015)
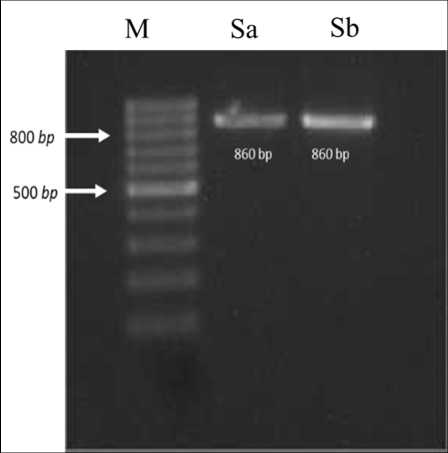
Gambar 1. Elektroforesis Hasil Amplifikasi DNA Mitokondria
Keterangan: M= Ladder DNA (100 bp), Sa= sampel No.1 dan Sb = sampel No. 2
Optimasi Digesti Enzim Restriksi
Lima macam enzim restriksi (HaeIII (BsuRI), HindIII, HinfI, MboI dan HpyP31 (DdeI)) dengan waktu digesti yang optimal dapat dilihat pada Tabel 2. Optimasi penggunaan buffer dilakukan dengan 2 macam formula yaitu Buffer R (1) dan Buffer Tango (2). Pada Gambar 1 perbedaan penggunaan buffer tidak menunjukkan hasil yang berbeda. Penggunaan buffer dengan formula 1 dan 2 menunjukkan keberhasilan proses digesti. Keberhasilan terjadi akibat adanya pengenalan situs pemotongan enzim restriksi pada fragmen tersebut yang mengakibatkan perubahan ukuran panjang fragmen DNA yang awalnya 860 bp menjadi lebih pendek dari 860 bp. Hal ini berarti penggunaan Buffer R dan Buffer Tango tidak mempengaruhi hasil digesti. Namun setiap enzim memiliki compatible buffer yang berbeda sehingga petunjuk pada KIT insert harus dibaca dan diperhatikan. Berdasarkan KIT insert, compatible buffer yang digunakan adalah buffer R pada enzim HaeIII, HindIII, HinfI dan MboI sedangkan pada enzim DdeI menggunakan buffer Tango (Thermo scientific KIT, 2010).
Tabel 2. Jenis Enzim Restriksi dengan suhu dan waktu digesti yang optimal
|
Kode Enzim Enzim Restriksi |
Suhu Suhu Compatible Waktu Situs Pemotongan* Inkubasi* Inaktivasi* Buffer Digesti |
|
BsuRI I (HaeIII) |
5' .GG-CC. 3' 10x Buffer R 2 jam 3' .CC↑GG. 5' 37oC 80 oC |
|
II HindIII |
5' .A-AGCTT. 3' 10x Buffer R - 37oC 80 oC x uffer 3 ...TTCGA,A... 5 |
|
III HinfI |
5' .G-ANTC. 3' 10χ Buffer R 4 jam 37oC 65 oC x uffer jam 3 ...CTNA,G... 5 |
|
IV MboI |
5' .,.-GATC.,. 3' 10χ Buffer R 2 jam 37oC 65 oC x uffer jam 3 .CTAG,. 5 |
|
HpyF3I V (DdeI) |
5' .C-TNAG. 3' 10χ Buffer 2 jam 37oC 65 oC x uffer jam 3 .GANT,C. 5 Tango |
*Sumber : Thermo Scientific (2010)
(e)
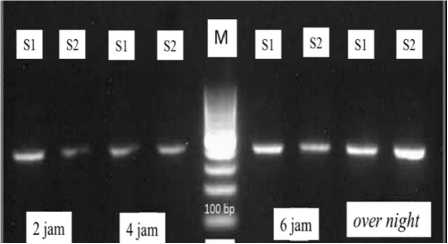
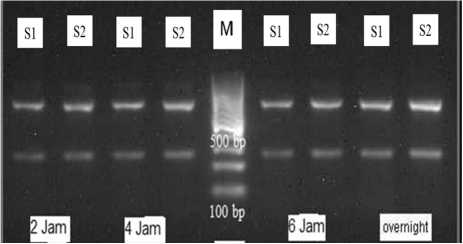
Gambar 2. Hasil optimasi formula PCR dan waktu digesti enzim restriksi (a) Enzim BsuRI ; (b) Enzim HindIII; (c) Enzim MboI; (d) Enzim HpyF3I; (e) Enzim Hinf I. Keterangan: M= Ladder DNA (100 bp); S1= Sampel No.1 Formula 1; S2=Sampel No.1 dengan formula 2
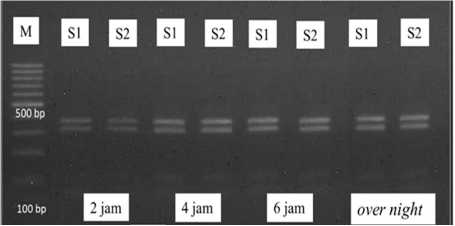
(a)
(b)
(c)
(d)

(a)

(b)
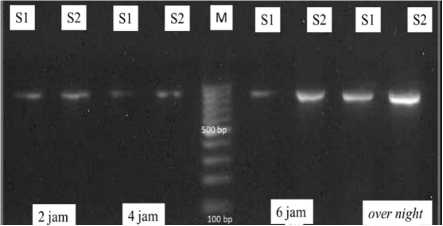
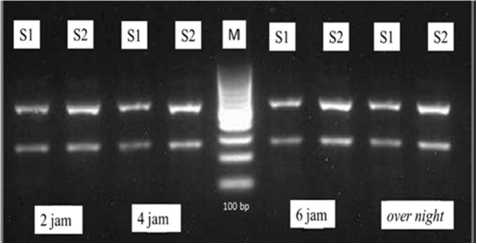
Gambar 3. Hasil optimasi (a). hasil optimasi waktu digesti enzim HinfI dengan sampel yang berbeda. (b) hasil optimasi ulang enzim HinfI. Keterangan: M= Ladder DNA (100 bp); S0 = produk PCR sebelum dipotong enzim; Sa= Sampel No. 1; Sb=Sampel No.2
Waktu optimal digesti merupakan jumlah waktu yang diperlukan untuk dapat memotong fragmen DNA yang dikenali oleh enzim restriksi. Pada KIT insert waktu inkubasi enzim dapat mencapai 16 jam, tetapi diperlukan optimasi waktu tersingkat untuk dapat menghasilkan produk digesti yang baik. Perbedaan waktu inkubasi digesti yang dilakukan yaitu 2 jam, 4 jam, 6 jam dan 16 jam (overnight). Pada Gambar 2 menunjukkan bahwa 2 jam merupakan waktu tersingkat yang dapat menghasilkan potongan fragmen DNA. Maka dianggap 2 jam adalah waktu yang optimal. Namun terdapat perbedaan pada enzim HinfI yang memiliki perbedaan hasil digesti dengan sampel yang berbeda (Gambar 3). Sampel Sa memiliki waktu digesti yang berbeda dengan sampel Sb. Sampel Sa (Gambar 3a) pada waktu 2 jam sudah terlihat 2 fragmen DNA yang terpotong, yaitu 550 bp dan 310 bp, namun pada sampel Sb terlihat 3 fragmen yaitu 860 bp, 550 bp dan 310 bp. Hal tersebut diduga ada bagian fragmen produk PCR awal yang berukuran 860 bp belum terpotong sempurna, sehingga dibutuhkan penambahan optimasi. Optimasi yang dilakukan dengan mengulang waktu digesti sampel Sb dengan penambahan waktu digesti yaitu 4 jam, 6 jam dan 16 jam. Dilihat pada Gambar 3b rentang waktu pemotongan 4 jam sudah terlihat fragmen 860 bp terpotong menghasilkan dua fragmen yaitu 550 bp dan 310 bp. Maka pada enzim HinfI waktu optimal digesti adalah 4 jam. Beberapa faktor yang mempengaruhi perbedaan waktu digesti terjadi karena konsentrasi DNA setiap sampel berbeda, sehingga diperlukan waktu digesti yang lebih panjang. Waktu digesti yang lebih lama mengakibatkan performa dari suatu enzim restriksi meningkat. Oleh karena itu diperlukan adanya pengukuran konsentrasi DNA setiap sampel sebelum lanjut ke tahapan PCR, agar formula digesti dan waktu digesti dapat seragam (Gerstein, 2001).
Optimasi digesti dengan lima macam enzim restriksi (HaeIII (BsuRI), HindIII, HinfI, MboI dan HpyP31 (DdeI)) dilakukan dengan 2 sampel orang Bali yang berbeda dengan kode Sa dan Sb. Enzim tersebut diduga mengenali urutan spesifik nukleotida pada daerah D-loop DNA mitokondria (Candramila, 2002). Proses digesti berhasil jika produk PCR dengan ukuran fragmen 860 bp terpotong menjadi ukuran fragmen-fragmen yang lebih pendek. Enzim HaeIII,
HinfI, MboI dan DdeI merupakan enzim yang berhasil mendapatkan potongan fragmen yang lebih pendek dari ukuran produk PCR (Gambar 2a, 2c, 2d dan 2e). Namun pada enzim HindIII tidak terdapat perbedaan ukuran fragmen DNA sebelum dan sesudah digesti. Fragmen yang dihasilkan tetap pada ukuran 860 bp. Maka diduga enzim HindIII tidak berhasil terdigesti karena urutan enzim HindIII tidak mengenali daerah D-loop DNA mitokondria (Gambar 2b). Hal tersebut terjadi karena tidak terdapat situs pemotongan enzim HindIII pada daerah D-loop DNA mitokondria. Pendapat ini didukung oleh penelitian dari Candramila (2002), enzim HindIII tidak menghasilkan fragmen dengan ukuran lebih kecil dari produk PCR pada masyarakat Betawi. Pengenalan situs pemotongan enzim HindIII juga belum ada dilaporan pada sekuen DNA mitokondria masyarakat di Eropa (Hofmanová et al., 2016). Urutan DNA rCRS juga tidak ditemukan adanya situs pemotongan pada enzim HindIII (Bandelt et al., 2013)
SIMPULAN
Hasil optimasi dari lima macam enzim restriksi (HaeIII (BsuRI), HindIII, HinfI, MboI dan HpyP31 (DdeI)) didapatkan bahwa ada perbedaan formula dan waktu digesti enzim. Enzim HaeIII (BsuRI),HindIII, HinfI dan MboI menunjukkan formula 2 yang optimal, sedangkan enzim HpyP31 (DdeI) formula 1 merupakan formula yang optimal. Waktu digesti 2 jam menunjukkan hasil optimal pada Enzim HaeIII(BsuRI), MboI dan HpyP31 (DdeI), sedangkan enzim HinfI waktu digestinya adalah 4 jam. Enzim HindIII tidak terdapat hasil potongan fragmen DNA setelah digesti, maka enzim HindIII tidak memiliki situs pemotongan pada daerah D-loop DNA mitokondria. Sehingga enzim HindIII tidak dapat dipergunakan untuk tes DNA.
UCAPAN TERIMA KASIH
Penulis mengucapkan terima kasih kepada para probandus, kepala dan staf di Laboratorium Biomedik Terpadu, Fakultas Kedokteran, Universitas Udayana. Serta semua pihak yang terlibat dalam penelitian ini sehingga dapat dilakukan dengan baik.
KEPUSTAKAAN
Andrew MN. 2013. Assignment of Haplotypes groups by Mitochondrial DNA in BTN323 Population using PCR-RFLP analysis. Practical Reports. Department of Biotechnology University of the Western Cape.
Bandelt Hans-J, Anita KB, Martin BR. 2013. The case for the continuing use of the revised Cambridge Reference Sequence (rCRS) and the standardization of notation in human mitochondrial DNA Studies. Journal of Human Genetics 59: 66-77.
Candramila W. 2002. Variasi Genetik Populasi Betawi Berdasarkan Polimorfisme DNA Mitokondrion. Tesis, Bogor, Institut Pertanian Bogor
Doosti A, Payam GD. 2011. Genetic Polymorphisms of Mitochondrial Genome D-loop Region in Bakhtiarian Population by PCR-RFLP. International Journal of Biology 3(4): 4-46.
Fazri HM, Royani JI, Dasumiati. 2015. Isolation and amplification of Keladi tikus (Thyponium flagelliform) DNA for identification of genetic variation. Jurnal Bioteknologi, Teknologi dan Sains Indonesia 2(2):42-48.
Gerstein AS. 2001. Molecular Biology Problem Solver: A Laboratory Guide. Wiley-Liss, Inc Hofmanová Z, Kreutzer S, Hellenthal G, Sell C, Diekmann Y, Díez-Del-Molino D, van Dorp L, López S, Kousathanas A, Link V, Kirsanow K, Cassidy LM, Martiniano R, Strobel M, Scheu A, Kotsakis K, Halstead P, Triantaphyllou S, Kyparissi-Apostolika N, Urem-Kotsou D, Ziota C, Adaktylou F, Gopalan S, Bobo DM, Winkelbach L, Blöcher J, Unterländer M, Leuenberger C, Çilingiroğlu Ç, Horejs B, Gerritsen F, Shennan SJ, Bradley DG, Currat M, Veeramah KR, Wegmann D, Thomas MG, Papageorgopoulou C, Burger J. 2016. Early
farmers from across Europe directly descended from Neolithic Aegeans. Proceedings of the National Academy of Sciences of the United States of America 113(25): 6886-91.
Junitha IK. 2015. Analisis DNA dalam Forensik dan Potensinya untuk Penelusuran Kawitan.
Materi Ceramah Maha Semaya Warga Pande Kabupaten Gianyar. Gianyar
Junitha IK, Pharmawati M, Rosiana W. 2012. Genetic Diversity of Soroh Celagi (Pasek Catur
Sanak Clan) Basedon Y-chromosomal Microsatellites DNA. The 4th International
Conferences on Biosciences and Biotechnology. The 4th International Conferences on Biosciences and Biotechnology Proceeding: Denpasar, 239–243
Kusumawati D. 2011. Enzim Restriksi. Modul Praktikum Biologi Molekul. UPI. Available from: http://www.enzim_restriksi.pdf.
Luftig M, Richey S, Ntroduction I. 2001. DNA and Forensic Science. Corpus ID: 1796608
Mastura. 2007. Haplogrup mtDNA Manusia atas Dasar RFLP dan Posisi Mutasi Urutan
Nukleotida lengkap. Tesis. Institut Teknologi Bandung
Moore JM, Isenberg AR. 1999. Mitochondrial DNA Analysis at the FBI Laboratory. Forensic
Science Communications 1(2) https://archives.fbi.gov/archives/about-us/lab/forensic-science-communications/fsc/july1999/dnalist.htm
Putri IARDN, Yowani SC, Wirajana IN. 2017. Optimasi Digesti Enzim Restriksi Sac II pada Isolat tuberculosis H37Rv untuk Deteksi Mutasi Promoter inhA pada Kasus MDR-TB dengn metode-RFLP. Metamorfosa: Journal of Biological Sciences 4(1): 87-93.
Siti HH, Gun Gumilar G, Natalia D, Saifuddin Noer A. 2013. Variasi Urutan nukleotida Daerah D-Loop DNA Mitokondria Manusia pada Dua Populasi Asli Indonesia Tenggara. Program Studi Kimia, FMIPA, Universitas Pendidikan Indonesia
Yudianto A. 2020. Buku Referensi Pemeriksaan Forensik DNA Tulang dan Gigi. Sintesa Book.
Yogyakarta.
72
Discussion and feedback