DNA barcoding on marine invertebrates
on
JURNAL BIOLOGI UDAYANA 25(1): 46-56
P ISSN: 1410-5292 E ISSN: 2599-2856
DNA barcoding pada invertebrata laut
DNA barcoding on marine invertebrates
Agus Mohammad Hikam1, Nurul Jadid Mubarakati1, Muhammad Dailami2, Abdul Hamid A. Toha3*
-
1Program Studi Biologi, Fakultas Matematika dan Ilmu Pengetahuan Alam, Universitas Islam Malang, Jl. Mayjen Haryono No. 193, Malang65144, East Java, Indonesia
-
2Departemen Budidaya Perairan, Fakultas Perikanan dan Ilmu Kelautan, Universitas Brawijaya. Jl. Veteran No. 16, Malang 65145, East Java, Indonesia
-
3Jurusan Perikanan, Fakultas Perikanan dan Ilmu Kelautan, Universitas Papua. Jl. Gunung Salju, Manokwari 98314, West Papua, Indonesia
*Email: h.toha@unipa.ac.id
Diterima 24 Pebruari 2021 Disetujui 29 April 2021
INTISARI
Banyaknya spesies invertebrata laut yang memiliki kemiripan morfologi menyebabkan kesalahan identifikasi sangat mungkin terjadi. Identifikasi spesies secara molekuler sangat diperlukan dalam mempelajari taksonomi yang akurat. Penelitian ini bertujuan untuk menentukan identitas invertebrata laut dari Perairan Papua dengan teknik DNA barcoding menggunakan marka gen COI (Cytochrome c oxidase I). Penelitian mengidentifikasi 29 individu invertebrata laut, yang terdiri dari teripang (6 sampel), lobster (6 sampel), gurita (6 sampel), chiton (5 sampel), dan bulu babi (6 sampel). Metode identifikasi molekuler terdiri dari isolasi DNA, PCR fragmen gen COI, sekuensing, dan analisis sekuens DNA menggunakan software bioinformatika.Hasil penelitian ini menunjukkan bahwa teripang merupakan spesies Bohadschia marmorata, lobser merupakan spesies Panulirus versicolor, gurita merupakan spesies Octopus cyanea, chiton merupakan spesies Ischnochiton australis, dan bulu babi merupakan spesies Tripneustes gratilla, berdasarkan analisis BLAST dan Boldsystem. Dengan tingkat kemiripan sampel dan rujukan dalam kisaran 84.58 sampai 100.00%. Indeks disparitas, jarak genetik dan pohon filogenetik mendukung hasil ini.
Kata kunci: COI, DNA barcoding, Invertebrata laut
ABSTRACT
A large number of species of marine invertebrates that have similar morphology cause misidentification very possible. Molecular species identification is important in taxonomy study accurately. This study aims to determine the identity of Aquatic Papua marine invertebrates with DNA barcoding technique using markers COI gene (Cytochrome c oxidase I). The study identified 29 individuals of marine invertebrates, which came from sea cucumbers (6 samples), lobsters (6 samples), octopus (6 samples), chitons (5 samples), and sea urchins (6 samples). Molecular identification methods consist of DNA isolation, PCR COI gene fragments, sequencing, and DNA sequence analysis using bioinformatics software. The results showed that the sea cucumber came from the Bohadschia marmorata species, the lobster came from the Panulirus versicolor species, the octopus came from the Octopus cyanea species, the chiton came from the Ischnochitonaustralis species, and the sea urchins came from the Tripneustesgratilla species. The similarity of samples and references are in the range of 84.58 to 100.00%. The disparity index, genetic distance, and phylogenetic tree supported this result.
Keywords: COI, DNABarcoding, Marine invertebrates
PENDAHULUAN
Invertebrata laut memiliki peran penting untuk menjaga ekosistem, sumber pangan ekonomis, maupun obat-obatan secara keseluruhan (Dustin, 2012). Baik fosil maupun terbaru, invertebrata laut merupakan sumber biomineral yang menakjubkan yang sebagian besar muncul pada tahap awal evolusi (Ehrlich, 2019). Invertebrata laut juga memiliki potensi fungsi imunologi ekologis (Ellis et al., 2011). Meskipun demikian, invertebrata laut juga memiliki potensi ancaman dari spesies invasif non-native (Hughes et al., 2020), manusia dan proses alami (Wilkinson, 2004), Sementara itu banyaknya spesies invertebrata laut yang belum teridentifikasi (Mangubhai et al., 2012; Nikijuluw, 2017) dan memiliki sifat dimorfisme, perubahan morfologi selama perkembangan dan penyimpanan spesimen (Ward et al., 2009) menyebabkan kendala pendekatan morfologi untuk identifikasi invertebrata laut. Apalagi bila spesimen invertebrata rusak (Schander & Willasen, 2005), masalah umur sampel (Webb et al., 2006) dan beberapa spesies tampak samar secara morfologi sehingga identifikasi makin sulit (Muchlisin et al., 2013).
Identifikasi spesies secara molekuler sangat diperlukan dalam mengatasi kendala pendekatan morfologi dan mempelajari taksonomi invertebrata laut secara tepat. Hal ini dapat dilakukan melalui DNA Barcoding (Taylor dan Harrist, 2012), yang fokus terutama pada daerah gen sitokrom oksidase subunit I (COI) (Hebert et al., 2003). DNA barcoding memberikan kecepatan dan keakuratan dalam identifikasi spesies (Muchlisin et al., 2013; Karim et al., 2015). DNA Barcode memiliki dua tujuan, yaitu untuk identifikasi molekuler yang sudah terdeskripsikan maupun spesies yang belum terdeskripsikan (Rahayu & Jannah, 2019). Casiraghi et al. (2010) menjelaskan bahwa DNA Barcode adalah alat molekuler dan bioinformatika untuk identifikasi spesies biologi. Kelebihan lain DNA barcoding adalah dapat mengidentifikasi spesies yang sulit dibedakan secara morfologi (Hebert et al., 2004). Identifikasi dengan menggunakan DNA barcode
telah terbukti secara efektif mampu membedakan antar spesies secara akurat (Borges et al., 2016; Fuentes-López et al., 2020; Saleky et al., 2016). Metode taksonomi molekuler ini banyak digunakan untuk melengkapi pendekatan morfologi dalam identifikasi spesies dan dalam membangun hubungan filogenetik (Galan et al., 2018).
Identifikasi dan hubungan genetik spesies invertebrata laut sangat penting diantaranya untuk sumber dan seleksi gen, menentukan hubungan kekerabatan, menentukan proses evolusi dan ekologi serta untuk kepentingan konservasi (Toha, 2007). Penelitian DNA barcode pada invertebrata laut pernah dilakukan pada echinodermata (Ward et al., 2008, Chow et al., 2016), stomatopoda (Barber & Boyce, 2006), larva invertebrata (Webb et al., 2006), gastropoda (Puillandre et al., 2009). Penelitian DNA Barcode invertebrata laut asal Papua diantaranya dilakukan oleh Pranata et al. (2020), Aprilia et al., (2014), Kurniasari et al., (2014), dan Sahriyani et al. (2014).
Penelitian umumnya dilakukan pada tingkat spesies dan genus. Penelitian secara menyeluruh pada tingkat invertebrata belum pernah dilakukan. Penelitian ini bertujuan untuk menentukan identitas spesies invertebrata laut yang dikoleksi secara acak dari Perairan Papua. Penelitian ini penting untuk taksonomi dan status konservasi biota perairan terutama invertebrata laut.
MATERI DAN METODE
Lokasi Penelitian
Sampel penelitian dikoleksi dari perairan Papua (Gambar 1). Jumlah sampel seluruhnya 29 individu yang terdiri dari teripang (6 sampel), lobster (6 sampel), gurita (6 sampel), chiton (5 sampel), bulubabi (6 sampel). Setiap individu diambil jaringan organ dengan ukuran sekitar 4 cm3 (~ 5g). Semua sampel yang dikoleksi disimpan dalam etanol 96% untuk menjaga keawetannya.
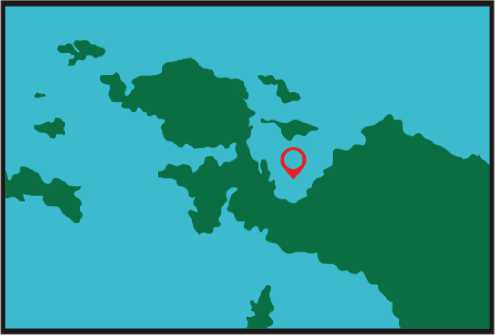
Gambar 1. Lokasi pengambilan sampel tiap jenis invertebrata laut di Perairan Papua.
Analisis Molekuler
Isolasi DNA genom dari sampel lobster dan gurita dilakukan dengan menggunakan reagen Geneaid DNA Isolation Kit (tissue) dengan protokol standar seperti yang digunakan oleh Toha et al., (2020). Untuk ekstraksi DNA genom dari sampel teripang, chiton dan bulu babi dilakukan dengan menggunakan metode chelex 10% (Walsh et al., 2013) sesuai dengan metode yang dilakukan oleh Dailami et al., (2018). Isolat DNA yang diperoleh disimpan dalam freezer dan ekstrak DNA dalam larutan chelex disimpan dalam kulkas sampai digunakan untuk PCR.
Produk PCR yang diperoleh dimurnikan menggunakan Shrimp Alkaline Phosphotase (Amersham Biosciences Corporation, Arlington Heights, Illinois, USA) dan Exonuclease (Amersham) (SAP/EXO) seperti yang dilakukan oleh Dailami et al. (2018). Sekuensing 2 arah (bidirectional) dilakukan dengan metode dideoksiterminasi sanger dengan menggunakan menggunakan Big Dye© terminator chemistry (Perkin Elmer) di Cornell University.
Fragmen gen CO1 diamplifikasi menggunakan pasangan primer universal untuk masing-masing jenis invertebrata sebagaimana tertera pada Tabel 1. Amplifikasi DNA dilakukan dengan Teknik Polymerase Chain Reaction (PCR) menggunakan kit GoTaq Green (Promega) dengan total volume 50 µL yang terdiri dari 2 µL DNA template, 25 µL GoTaq Green Mastermix, 1 µL DMSO, 1 µL BSA, 2,5 µL primer forward, 2,5 µL primer reverse dan sisanya adalah ddH2O. Proses PCR berlangsung parameter suhu 80oC (10 detik), pre-denaturasi 94oC (3 menit), denaturasi 95oC (30 detik), annealing 50oC (30 detik), ekstension 72oC (45 detik), final-extension 72oC (5 menit). Tahap denaturasi, annealing dan extension diulang sebanyak 40 siklus.
Tabel 1. Daftar primer yang digunakan dalam proses PCR
|
Sampel |
Sekuen Primer Referensi |
|
Teripang |
CO1F: 5’-CCT GCA GGA GGAGGAGAY CC-3’ Palumbi dan Benzie, 1991 CO1e: 5'-CCAGAGATTAGAGGGAATCAGTG-3' Palumbi et al., 2002 |
|
Lobster |
LCO-1490:5’-GGTCAACAAATCATAAAGATATTGG-3’ Folmer et al., 1994 HCO-2198:5’-TAAACTTCAGGGTGACCAAAAAATCA-3’ |
Gurita F1: 5’-GATGTGTTGAAATTACGGTCGGT-3´ Metz et al., 1998
R1:5’-TGATCCGGCTTAGTCGGAACTGC-3´
Chiton LCO-1490:5’-GGTCAACAAATCATAAAGATATTGG-3’ Folmer et al., 1994
HCO-2198:5’-TAAACTTCAGGGTGACCAAAAAATCA-3’
Bulu Babi CO1F: 5’-CCTGCAGGAGGAGGAGAYCC-3’
Palumbi dan Benzie, 1991
CO1TR1: 5’-GGCATTCCAGCTAGTCCTARAA-3’
Hasil PCR divisualisasi dengan elektroforesis gel agarosa 1% (b/v) menggunakan SB-buffer (sodium boric buffer). Sebanyak 0,5 gram agarosa dimasukkan ke dalam 50 mL SB-buffer dan
dipanaskan dengan menggunakan microwave hingga cairan berwarna jernih kemudian dicetak dengan cetakan gel dan didiamkan hingga mengeras. Sebanyak 4 µL produk PCR dicampur
dengan 1 µL loading dye kemudian dimasukkan dalam sumur gel. Proses elektroforesis dilakukan dengan tegangan100 volt selama 30 menit. Pewarnaan DNA menggunakan etidiumbromida dengan cara merendam gel dalam larutan EtBr selama 10 menit kemudian dibilas dengan aquades. Pita DNA dilihat dengan bantuan UV transluminator dan didokumentasikan menggunakan kamera digital.
Analisis Data
Pengeditan hasil sekuensing dan penentuan komposisi nukleotida dianalisis dengan MegaX (Kumar et al., 2018). Urutan DNA disejajarkan dengan ClustalW vers. 1.4 (Thompson et al., 1994). Pola dan tingkat substitusi diperkirakan di bawah model parameter-2 Kimura (1980). Identifikasi spesies secara online menggunakan data genbank pada NCBI (http://blast.ncbi.nlm.nih.gov/Blast.cgi) dengan berbagai kode akses dengan metode BLAST (Basic Local Alignment Search Tool) (Altschul et al., 1990) atau Barcode di Boldsystem (Barcode of life data system v4) (Ratnasingham dan Hebert, 2007). Analisis DNA juga dilanjutkan dengan
menghitung jarak genetik antar spesies dan juga analisis pohon filogenetik invertebrata laut menggunakan metode Neighbor-Joining (NJ) (Saitou & Nei, 1987) dengan model Kimura 2-parameter, nilai bootstrap 1000x (Kumar et al., 2018).
HASIL
Karakter Molekuler
Berdasarkan hasil analisis homologi menggunakan BLAST menunjukkan bahwa teripang merupakan spesies Bohadschia marmorata dengan persentase kisaran 98.25% sampai 100.00%. Lobster merupakan spesies Panulirus versicolor dengan persentase kisaran 98.79% sampai 100.00%. Gurita merupakan spesies Octopus cyanea dengan persentase kisaran 99.75% sampai 100.00%. Chiton merupakan spesies Ischnochiton australis dengan persentase kisaran 85.52% sampai 85.90%. Bulu babi merupakan spesies Tripneustes gratilla dengan persentase kisaran 99.83% sampai 100.00%. Hasil identifikasi spesies pada Genbank disajikan pada Tabel 2.
Tabel 2. Hasil identifikasi spesies pada GenBank
|
Kode Sampel |
Jenis Invertebrata Laut |
Nama Spesies |
Panjang Sekuens (bp), Kode Akses dan Referensi |
|
T1, T2, T3, T4, T5, T6 |
Teripang |
Bohadschia marmorata |
656 bp JN543464.1 (Kim et al. 2013) |
|
L1, L2, L3, L4, L5, L6 |
Lobster |
Panulirus versicolor |
676 bp MW041245.1 (Irwani et al. 2020) 15767 bp KC107808.1 (Shen et al. 2013) 630 bp KT001513.1 (Samadi et al. 2015) |
|
G1, G2, G3, G4, G5, G6 |
Gurita |
Octopus cyanea |
626 bp MN977145.1 (Velasco et al. 2020) 600 bp GQ900742.1 (Huffard et al. 2010) |
|
C1, C2, C3, C4, C5 |
Chiton |
Ischnochiton australis |
689 bp EF414417.1 (Palmer et al. 2007) |
|
B1, B2, B3, B4, B5, B6 |
Bulu Babi |
Tripneustes gratilla |
15720 bp KJ680294.1 (Jung et al. 2014) 620 bp AY205442.1 (Lessioset al. 2003) 605 bp JX661100.1, 605 bp JX661098.1, 605 bp JX661091.1 (Casilaganet al. 2013) |
Sementara menurut uji BOLDsystem teripang mempunyai tingkat kemiripan antara 86.96% sampai 100.00% dengan Bohadschia marmorata, lobster mempunyai tingkat kemiripan antara 84.58% sampai 100.00% dengan Panulirus
versicolor, gurita mempunyai tingkat kemiripan antara 99.49% sampai 100.00% dengan Octopus cyanea, bulu babi mempunyai tingkat kemiripan antara 87.10% sampai 100.00% dengan Tripneustes gratilla. Sedangkan chiton tidak
ditemukan. Hal ini dapat terjadi karena belum adanya peneliti yang mendepositkan sekuen spesies tersebut, atau sampel yang diteliti merupakan spesies baru.Untuk lebih jelasnya
hasil analisis kemiripan spesies invertebrata laut berdasarkan uji BLAST dan Boldsystem disajikan pada Tabel 3.
Tabel 3. Hasil analisis kemiripan spesies invertebrata laut berdasarkan BLAST dan Boldsystem
|
No. |
Kode Sampel |
Nama Spesies |
BLAST |
Boldsystem | |||||
|
Kode Akses NCBI |
Query Cover (%) |
Nilai-E |
Nilai Identitas (%) |
Basis Data |
Tinggi (%) |
Rendah (%) | |||
|
Teripang | |||||||||
|
1 |
T1 |
Bohadschia marmorata |
JN543464.1 |
92 |
2e-109 |
98.69% |
Basis Data COI |
98.68 |
88.00 |
|
2 |
T2 |
Bohadschia marmorata |
JN543464.1 |
91 |
2e-109 |
98.69% |
Basis Data COI |
98.68 |
88.00 |
|
3 |
T3 |
Bohadschia marmorata |
JN543464.1 |
93 |
2e-109 |
98.69% |
Basis Data COI |
98.68 |
88.00 |
|
4 |
T4 |
Bohadschia marmorata |
JN543464.1 |
93 |
2e-109 |
98.69% |
Basis Data COI |
98.68 |
88.00 |
|
5 |
T5 |
Bohadschia marmorata |
JN543464.1 |
93 |
2e-103 |
100.00% |
Basis Data COI |
100.00 |
86.96 |
|
6 |
T6 |
Bohadschia marmorata |
JN543464.1 |
91 |
8e-108 |
98.25% |
Basis Data COI |
98.55 |
87.65 |
|
Lobster | |||||||||
|
7 |
L1 |
Panulirus versicolor |
MW041245.1 |
98 |
0.0 |
99.56 |
Basis Data COI |
99.70 |
85.03 |
|
8 |
L2 |
Panulirus versicolor |
KC107808.1 |
99 |
0.0 |
99.12 |
Basis Data COI |
100.00 |
84.95 |
|
9 |
L3 |
Panulirus versicolor |
KC107808.1 |
99 |
0.0 |
99.13 |
Basis Data COI |
100.00 |
85.02 |
|
10 |
L4 |
Panulirus versicolor |
MW041245.1 |
99 |
0.0 |
98.79 |
Basis Data COI |
100.00 |
84.69 |
|
11 |
L5 |
Panulirus versicolor |
MW041245.1 |
98 |
0.0 |
99.70 |
Basis Data COI |
99.85 |
85.03 |
|
12 |
L6 |
Panulirus versicolor |
KT001513.1 |
99 |
0.0 |
100.00 |
Basis Data COI |
100.00 |
84.58 |
|
Gurita | |||||||||
|
13 |
G1 |
Octopus cyanea |
MN977145.1 |
100 |
0.0 |
100.00 |
Basis Data COI |
100.00 |
99.75 |
|
14 |
G2 |
Octopus cyanea |
MN977145.1 |
100 |
0.0 |
100.00 |
Basis Data COI |
100.00 |
99.75 |
|
15 |
G3 |
Octopus cyanea |
GQ900742.1 |
100 |
0.0 |
100.00 |
Basis Data COI |
100.00 |
99.49 |
|
16 |
G4 |
Octopus cyanea |
MN977145.1 |
100 |
0.0 |
100.00 |
Basis Data COI |
100.00 |
99.75 |
|
17 |
G5 |
Octopus cyanea |
MN977145.1 |
100 |
0.0 |
100.00 |
Basis Data COI |
100.00 |
99.75 |
|
18 |
G6 |
Octopus cyanea |
MN977145.1 |
100 |
0.0 |
99.75 |
Basis Data COI |
99.75 |
99.49 |
|
Chiton | |||||||||
|
19 |
C1 |
Ischnochitonaustralis |
EF414417.1 |
99 |
0.0 |
85.90 |
Basis Data COI |
- |
- |
|
20 |
C2 |
Ischnochitonaustralis |
EF414417.1 |
100 |
0.0 |
85.76 |
Basis Data COI |
- |
- |
|
21 |
C3 |
Ischnochitonaustralis |
EF414417.1 |
99 |
2e-171 |
85.52 |
Basis Data COI |
- |
- |
|
22 |
C4 |
Ischnochitonaustralis |
EF414417.1 |
99 |
0.0 |
85.90 |
Basis Data COI |
- |
- |
|
23 |
C5 |
Ischnochitonaustralis |
EF414417.1 |
99 |
0.0 |
85.61 |
Basis Data COI |
- |
- |
|
Bulu Babi | |||||||||
|
24 |
B1 |
Tripneustesgratilla |
KJ680294.1 |
100 |
0.0 |
100.00 |
Basis Data COI |
100.00 |
87.10 |
|
25 |
B2 |
Tripneustesgratilla |
AY205442.1 |
99 |
0.0 |
99.83 |
Basis Data COI |
100.00 |
87.10 |
|
26 |
B3 |
Tripneustesgratilla |
JX661100.1 |
100 |
0.0 |
100.00 |
Basis Data COI |
100.00 |
87.10 |
|
27 |
B4 |
Tripneustesgratilla |
JX661098.1 |
100 |
0.0 |
99.83 |
Basis Data COI |
100.00 |
87.10 |
|
28 |
B5 |
Tripneustesgratilla |
KJ680294.1 |
100 |
0.0 |
100.00 |
Basis Data COI |
100.00 |
87.10 |
|
29 |
B6 |
Tripneustesgratilla |
JX661091.1 |
100 |
0.0 |
100.00 |
Basis Data COI |
100.00 |
87.10 |
Indeks Disparitas
Pada proses identifikasi sekuen invertebrata laut juga memakai analisis indeks disparitas, hal ini bertujuan untuk melihat seberapa jauh sekuens telah berevolusi dengan melihat komposisi dasar antar sekuen. Hasil analisis indeks disparitas antar individu setiap spesies invertebrata laut disajikan pada Tabel 4.
Semua individu dari masing-masing spesies merupakan satu spesies. Semua teripang merupakan spesies Bohadschia marmorata, lobster dari spesies Panulirus versicolor, gurita dari spesies Octopus cyanea, chiton dari Ischnochiton australis dan bulu babi berasal dari spesies Tripneustes gratilla. Hasil indeks disparitas ini mendukung hasil blast dan boldsystem. Indeks disparitas digunakan untuk
menguji hipotesis nol bahwa sekuens telah berevolusi dengan pola substitusi yang sama, seperti yang dinilai dari sejauh mana perbedaan
bias komposisi dasar antar urutan (Kumar & Gadagkar, 2001).
Tabel 4. Indeks Disparitas
|
1 |
2 |
3 |
4 |
5 |
6 |
1 |
2 |
3 |
4 |
5 |
6 | ||
|
T1 |
0,00 |
0,00 |
0,00 |
0,00 |
0,00 |
L1 |
0,00 |
0,00 |
0,00 |
0,00 |
0,00 | ||
|
T2 |
1,00 |
0,00 |
0,00 |
0,00 |
0,00 |
L2 |
0,39 |
0,00 |
0,00 |
0,00 |
0,00 | ||
|
T3 |
1,00 |
1,00 |
0,00 |
0,00 |
0,00 |
L3 |
0,46 |
1,00 |
0,00 |
0,00 |
0,00 | ||
|
T4 |
1,00 |
1,00 |
1,00 |
0,00 |
0,00 |
L4 |
0,44 |
1,00 |
1,00 |
0,00 |
0,00 | ||
|
T5 |
1,00 |
1,00 |
1,00 |
1,00 |
0,00 |
L5 |
1,00 |
1,00 |
1,00 |
1,00 |
0,00 | ||
|
T6 |
1,00 |
1,00 |
1,00 |
1,00 |
1,00 |
L6 |
0,43 |
1,00 |
1,00 |
1,00 |
1,00 | ||
|
1 |
2 |
3 |
4 |
5 |
6 |
1 |
2 |
3 |
4 |
5 |
6 | ||
|
G1 |
0,00 |
0,00 |
0,00 |
0,00 |
0,00 |
B1 |
0,00 |
0,00 |
0,00 |
0,00 |
0,00 | ||
|
G2 |
1,00 |
0,00 |
0,00 |
0,00 |
0,00 |
B2 |
1,00 |
0,00 |
0,00 |
0,00 |
0,00 | ||
|
G3 |
1,00 |
1,00 |
0,00 |
0,00 |
0,00 |
B3 |
1,00 |
1,00 |
0,00 |
0,00 |
0,00 | ||
|
G4 |
1,00 |
1,00 |
1,00 |
0,00 |
0,00 |
B4 |
1,00 |
1,00 |
1,00 |
0,00 |
0,00 | ||
|
G5 |
1,00 |
1,00 |
1,00 |
1,00 |
0,00 |
B5 |
1,00 |
1,00 |
1,00 |
1,00 |
0,00 | ||
|
G6 |
1,00 |
1,00 |
1,00 |
1,00 |
1,00 |
B6 |
1,00 |
1,00 |
1,00 |
1,00 |
1,00 | ||
|
1 |
2 |
3 |
4 |
5 | |||||||||
|
C1 |
0,00 |
0,00 |
0,00 |
0,00 | |||||||||
|
C2 |
1,00 |
0,00 |
0,00 |
0,00 | |||||||||
|
C3 |
1,00 |
1,00 |
0,00 |
0,00 | |||||||||
|
C4 |
1,00 |
1,00 |
1,00 |
0,00 | |||||||||
|
C5 |
1,00 |
1,00 |
1,00 |
1,00 |
Catatan: angka dibawah diagonal adalah selisih jumlah alas. Sedangkan angka diatas diagonal adalah perkiraan kesalahan standar (standar error), data diperoleh melalui prosedur bootstrap (1000 replikasi). T = Teripang. L = Lobster, G = Gurita, C = Chiton, B = Bulu Babi.
Pohon Filogenetik
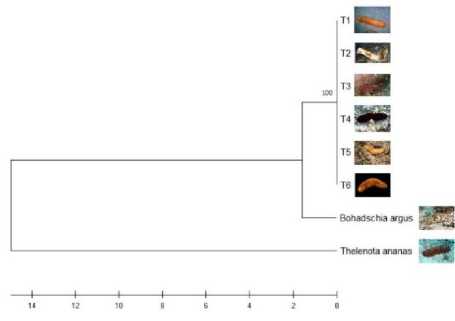
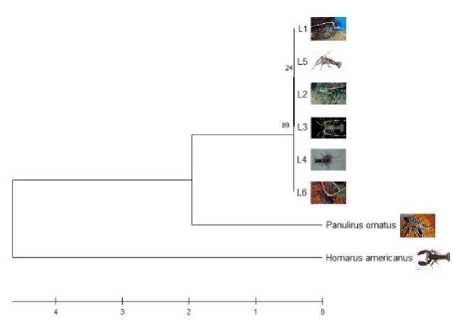
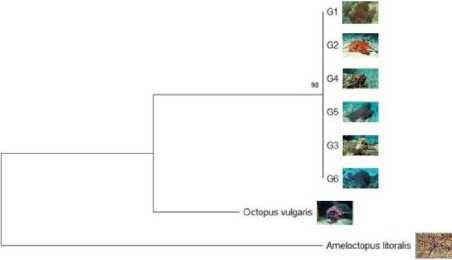
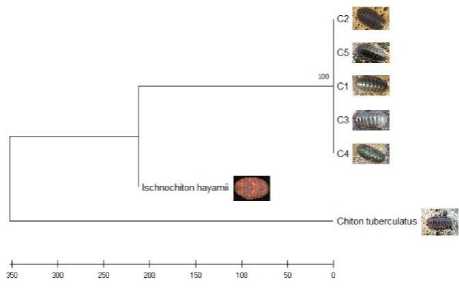
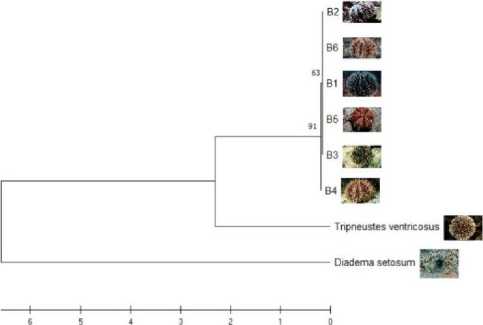
Gen COI selain digunakan dalam mengidentifkasi, mengukur jarak genetik tetapi juga dapat digunakan dalam merekonstruksi filogentik suatu spesies termasuk di dalamnya proses evolusi (Palumbi, 1994). Hasil analisis pohon filogenetik invertebrata laut asal Perairan Papua disajikan pada Gambar 1.
Berdasarkan gambar di atas tampak bahwa pohon filogenetik invertebrata memiliki pola yang hampir sama. Umumnya setiap spesies dengan jumlah sampel 5-6 individu berada dalam satu clade yang berarti memiliki hubungan genetik yang dekat satu dengan yang lain. Satu clade atau kelompok ini menandakan bahwa individu antar spesies memiliki sejarah evolusi
yang sama. Hal ini didukung dengan tingginya nilai bootstrap dan rendahnya jarak genetik dari masing-masing pohon filogenetik yang menggambarkan hubungan antar individu. Teripang, chiton, dan gurita dengan nilai kisaran bootsrap 98-100% menunjukkan kepercayaan pembentukan clade sangat akurat. Hal ini dapat dikatakan bahwa pengelompokan tersebut memiliki tingkat kesamaan yang tinggi (Rahayu et al., 2019). Sementara lobster dan bulu babi memiliki dua clade yang menunjukkan adanya sejarah evolusi berbeda antar kelompok individunya. Lima dari enam individu spesies T. gratilla (B1, 2, 3, 5, dan B6) membentuk kelompok terpisah dari satu individu lain (B4); dan empat dari enam spesies P. versicolor (L1, 2, 3, dan L5) membentuk kelompok terpisah dari dua individu lain (L4 dan L6).
(a)
(b)
4-----------------(-----------------1-----------------I-----------------I-----------------I-----------------I-----------------1
140 120 100 80 60 40 SO D
(c)
(d)
(e)
Gambar 2. Pohon filogenetik tiap jenis invertebrata laut. pohon filogenetik yang dibangun menggunakan Neighbor-Joining Tree, dan bootstrap pengujian dengan 1000 ulangan. (a) teripang, (b) lobster, (c) gurita, (d) chiton, (e) bulu babi.
PEMBAHASAN
E-value 0,0 yang diperoleh pada Tabel 3 menunjukkan keselarasan yang signifikan, artinya pencarian urutan spesimen pada penelitian ini identik yaitu dari genus yang sama bahkan pada tingkat spesies. (Tindi et al., 2017). Nilai query coverseluruh sampel invertebrata laut kisaran 91% -100%. Persentase ini menunjukkan kesesuaian panjang urutan sampel dengan database spesies di GenBank. Semakin tinggi nilai persentase query cover, semakin tinggi tingkat homologinya (Nugraha et al., 2014).
Analisis bootstrap digunakan untuk menguji validitas data sekuen untuk penyusunan cabang pada pohon filogenetik (Dharmayanti, 2011). Metode bootstrap memiliki nilai tertentu untuk menentukan jumlah pengulangan pada penyejajaran sekuen, sehingga dapat diketahui kemungkinan kekerabatan spesies pada pohon filogeni dengan data yang lebih akurat (Hillis dan Bull, 1993). Nilai bootstrappada Gambar 2dapat menunjukkan kestabilan cabang pada pohon filogenetik yang diperoleh. Nilai bootstrap cabang yang semakin tinggi dapat menunjukkan percabangan yang semakin kuat pada pohon filogenetik spesies (Abdullah et al., 2019).
Hasil analisis pohon filogenetik mendukung analisis blast dan boldsystem yang menganalisis spesies berdasarkan kemiripan dan homologi. Tampak bahwa individu setiap spesies terpisah dengan outgroup baik dalam satu genus maupun kelas berbeda.
Pohon filogenetik menggambarkan garis keturunan evolusi dari spesies, organisme atau dari satu nenek moyang berbeda. Kekerabatan antar spesies dilihat menggunakan topologi pohon filogenetik yang merupakan suatu metode yang dapat digunakan untuk mengetahui hubungan kekerabatan pada suatu taksa atau sekuen (Hall, 2013).
SIMPULAN
Penelitian ini berhasil menentukan spesies kelima jenis invertebrata laut asal perairan Papua,
masing-masing teripang merupakan spesies Bohadschia marmorata, lobster merupakan spesies Panulirus versicolor, gurita merupakan spesies Octopus cyanea, chiton merupakan spesies Ischnochiton australis, bulu babi merupakan spesies Tripneustes gratilla. Hasil ini juga didukung dengan pohon filogenetik yang menunjukkan pola yang hampir sama antar individu setiap spesies. Untuk sekuens chiton tidak ditemukan tingkat kemiripan data melalui Boldsystem, hal ini diperkirakan karena belum adanya peneliti yang mendepositkan sekuens spesies tersebut atau sampel yang diteliti merupakan spesies baru.
UCAPAN TERIMAKASIH
Terima kasih disampaikan kepada Laboratorium Genetika Universitas Papua yang membantu analisis data genetik.
KEPUSTAKAAN
Abdullah A, Nurilmala M, Sitaresmi KP. 2019.
DNA mini-barcodes sebagai penanda molekuler untuk ketertelusuran label pangan berbagai produk ikan layur. Jurnal Pengolahan Hasil Perikanan Indonesia 22(1): 33-40.
Altschul SF, Gish W, Miller W, Myers EW, Lipman DJ. 1990. Basic Local Alignment Search Tool. J. Mol. Biol. (215): 403-410.
Aprilia FE, Soewondo A, Widodo, Toha AHA.
2014. Amplifikasi Gen COI dan 16s rRNA dari Invertebrata Laut Plakobranchus ocellatus. Jurnal Biotropika 2(5): 276-278.
Barber PH, Erdmann MV. 2000. Molecular systematic of the Gonodactylidae (Stomatopoda) using mitochondrial
cytochrome oxidase C (subunit 1) DNA sequence data. J. Crustacean Biol. 20: 20–36.
Borges LMS, Hollatz C, Lobo J, Cunha AM, Vilela AP, Calado G, Coelho R, Costa AC, Ferreira MSG, Costa MH, Costa FO. 2016. With a little help from DNA barcoding: Investigating the diversity of Gastropoda from the Portuguese coast. Scientific Reports (6): 1–11.
Casiraghi M, Labra M, Ferri E, Galimberti A, De Mattia F. 2010. DNA barcoding: a six-question tour to improve users' awareness about the method. Briefings in
bioinformatics 11(4): 440-453.
Chow S, Konishi K, Mekuchi M, Tamaki Y, Nohara K, Takagi M, Niwa K, Teramoto W, Manabe H, Kurogi H, Suzuki S, Ando D, Tadao J, Kiyomoto M, Hirose M, Shimomura M, Kurashima A, Ishikawa T, Kiyomoto S. 2016. DNA barcoding and morphological analyses revealed validity of Diadema clarki Ikeda, 1939 (Echinodermata, Echinoidea, Diadematidae). Zookeys 585: 1-16. doi:
10.3897/zookeys.585.8161. eCollection
2016.
Dailami M, Santi D, Murtihapsari, Abubakar H, Toha AHA. 2018. Genetic analisys of cytochrome oxidase sub unit 1 gene fragment from Cirrhilabrus cf. ryukyuensis (Labridae) from Cenderawasih Bay and Raja Ampat. Jurnal Iktiologi Indonesia 18(3): 209.
https://doi.org/10.32491/jii.v18i3.347
Dharmayanti NLPI. 2011. Filogenetika Molekuler: Metode Taksonomi Organisme Berdasarkan Sejarah Evolusi. Wartazoa 21(1).
Dustin P. 2012. Microtubules. Springer Science & Business Media.
Ehrlich H. 2019. Marine biological materials of invertebrate origin. Springer Nature Switzerland AG.
https://doi.org/10.1007/978-3-319-92483-0
Ellis RP, Parry H, Spicer JI, Hutchinson TH, Pipe RK, Widdicombe S. 2011. Immunological function in marine invertebrates: Responses to environmental perturbation. Fish & Shellfish Immunology Journal 30. 1209e1222.
Folmer O, Black M, Hoeh W, Lutz R, Vrijenhoek R. 1994. DNA primers for amplification of mitochondrial cytochrome c oxidase subunit I diverse metazoan invertebrates. Molecular Marine Biology and Biotechnology 3(5): 294-299.
Fuentes-López A, Ruiz C, Galián J, Romera E. 2020. Molecular identification of forensically important fly species in Spain using COI barcodes. Science and Justice 60(3): 293–302.
Galan GL, Mendez NP, Cruz RYD. 2018. DNA bacoding of three selected gastropod species using cytochrome oxidase (COI) gene. Annals of West University of Timisoara, Ser. Biology 21(1): 93–102.
Hall BG. 2013. Building phylogenetic trees from molecular data with MEGA. Molecular Biology and Evolution 30(5): 1229-1235.
Hebert PDN, Cywinska A, Ball SL, Dewaard JR. 2003. Biological identifications through DNA barcodes. Proc. R. Soc. Lond. B. Biol. Sci. 270: 313–321.
Hebert PDN, Penton EH, Burns JM, Janzen DH, Hallwachs W. 2004. Ten Species In One: DNA Barcoding Reveals Cryptic Species In The Neotropical Skipper Butterfly Astraptes fulgerator. PNAS 101(41): 14812-14817.
Hillis DM, Bull JJ. 1993. An empirical test of bootstrapping as a method for assessing confidence in phylogenetic analysis. Systematic Biology 42: 182–192.
Hughes KA, Pescott OL, Peyton J, Adriaens T, Cottier‐Cook EJ, Key G, Rabitsch W, Tricarico E, Barnes DKA, Baxter N, Belchier M, Blake D, Convey P, Dawson W, Frohlich D, Gardiner LM, González‐Moreno P, James R, Malumphy C, Martin S, Martinou AF, Minchin D, Monaco A, Moore N, Morley SA, Ross K, Shanklin J, Turvey K, Vaughan D, Vaux AGC, Werenkraut V, Winfield IJ, Roy HE. 2020. Invasive non‐native species likely to threaten biodiversity and ecosystems in the Antarctic Peninsula region. Global change biology 26(4): 2702–2716. doi:
10.1111/gcb.14938
Karim A, Iqbal A, Akhtar R, Rizwan M, Amar A, Qamar U, Jahan S. 2015. Barcoding of Fresh Water Fishes From Pakistan. Mitochondrial DNA 1- 4.
Kumar S, Gadagkar SR. 2001. Disparity Index: A simple statistic to measure and test the homogeneity of substitution patterns between molecular sequences. Genetics 158: 1321-1327.
Kumar S, Stecher G, Li M, Knyaz C, Tamura K. 2018. MEGA X: Molecular Evolutionary Genetics Analysis across Computing Platforms. Mol. Biol. Evol. 35(6): 1547–
1549.
Kurniasari RD, Soewondo A, Toha AHA. 2014. Identifikasi Synaptula (Echinodermata :
Holothuroidea) Raja Ampat Berdasarkan Gen COI. Jurnal Biotropika 2(5): 265-268.
Mangubhai S, Erdmann MV, Wilson JR, Huffard CL, Ballamu F, Hidayat NI, Wen W. 2012. Papuan Bird’s Head Seascape: Emerging threats and challenges in the global center of marine biodiversity. Marine Pollution Bulletin 64(11): 2279–2295.
Metz EC, Robles-Sikisaka R, Vacquier VD. 1998. Nonsynonymous substitution in abalone spermfertilization genes exceeds substitution in intronsand mitochondrial DNA. Proceedings of the National Academy of Sciences of the United States of America 95: 10676 - 10681.
Muchlisin ZA, Thomy Z, Fadli N, Sarong MA, Siti-Azizah MN. 2013. DNA Barcoding of Freswater Fishes from Lake Laut Tawar, Aceh Province, Indonesia. Acta Ichtyiologica Et Piscatoria 43(1): 21-29.
Nikijuluw VPH. 2017. Biodiversity Conservation of Coastal Fish of Indonesia. Paper Presented at “Simposium Nasional Ikan dan Perikanan Pantai”, Organized by: Masyarakat Iktiologi Indonesia (MII). Bogor, 12-13 September 2017.
Nugraha F, Roslim DI, Ardilla YP, Herman. 2014. Analisis Sebagian Sekuen Gen Ferritin2 pada Padi (Oryza sativa L.) Indragiri Hilir, Riau. Biosaintifika Journal of Biology and Biology Education 6(2): 94-103.
Palumbi SR. 1994. Genetic divergence, reproductive isolation, and marine speciation. In Annual Review of Ecology and Systematics (25): 547–572.
Palumbi SR, Benzie J. 1991. Large mitochondrial DNA differences between morphologically similar Penaeid shrimp. Mol Mar Biol Biotechnol. 1991 Sep;1(1):27-34. PMID: 1669002.
Palumbi SR, Martin A, Romano S, McMillan WO, Stice L, Grabowski G. 2002. The simple fool’s guide to PCR version 2. University of Hawaii, 96822, 1–45.
Pranata B, Toha AHA, Kolibongso D. 2020. Genetic Of Panulirus versicolor Lobster In Cendrawasih Bay Papua And Lombok Waters West Nusa Tenggara. Jurnal Enggano 5(2): 2527-5186.
Puillandre N, Strong EE, Bouchet P, Boisselier MC, Couloux A, Samadi S. 2009. Identifying
gastropod spawn from DNA barcodes: possible but not yet practicable. Mol Ecol Res. 9: 1311–1321.
Rahayu DA, Nugroho ED, Listyorini D. 2019. DNA Barcoding Ikan Introduksi Khas Telaga Sari, Kabupaten Pasuruan. Biotropika: Journal of Tropical Biology 7(2): 51-62.
Rahayu DA, Jannah M. 2019. Dna Barcode Hewan dan Tumbuhan Indonesia. Jakarta Selatan: Yayasan Inspirasi Ide Berdaya.
Ratnasingham S, Hebert PD. 2007. BOLD: The Barcode of Life Data System. Molecular ecology notes 7(3): 355-364.
Sahriyani N, Widodo, Toha, AHA. 2014. Amplifikasi gen COI (Cytochrome C Oxidase Subunit I) pada holothuroidea yang dikoleksi dari kepulauan Raja Ampat. Biotropika: Journal of Tropical Biology 2 (4): 232-234.
Saitou N, Nei M. 1987. The neighbour-joining method: A new method for reconstructing phylogenetic trees. Molecular Biology and Evolution (4): 406-425.
Saleky D, Setyobudiandi I, Toha AHA, Takdir M, Madduppa HH. 2016. Length-weight relationship and population genetic of two marine gastropods species (Turbinidae:
Turbo sparverius and Turbo bruneus) in the Bird Seascape Papua, Indonesia.
Biodiversitas, Journal of Biological Diversity 17(1): 208–217.
Schander C, Willasen E. 2005. What can biological barcoding do for marine biology? Mar Biol Res. 1: 79–83.
Taylor HR, Harrist WE. 2012. An emergent science onthe brink of irrelevance: a review of the past 8 years of DNA barcoding. Molecular Ecology Resources 12(3): 377388.
Thompson JD, Desmond GH, Toby JG. 1994. CLUSTAL W: improving the sensitivity of progressive multiple sequence alignment through sequence weighting, positionspecific gap penalties and weight matrix choice. Nucleic Acids Research 22(22): 4673-4680.
Tindi M, Mamangkey NGF, Wullur S. 2017. DNA Barcode dan analisis filogenetik molekuler beberapa jenis bivalvia asal perairan Sulawesi Utara berdasarkan gen
COI. Jurnal Pesisir dan Laut Tropis 5(2): 32-38
Toha AHA. 2007. Keragaman Genetik Bulu Babi (Echinoidea). Biota. 12(2):131-135.
Toha AHA, Dailami M, Anwar S, Setiawan JB, Jentewo Y, Lapadi I, Madduppa H. 2020. The genetic relationships and indo-pacific connectivity of whale sharks (Rhincodon typus) with particular reference to mitochondrial COI gene sequences from Cendrawasih bay, Papua, Indonesia. Biodiversitas 21(5): 2159–2171.
https://doi.org/10.13057/biodiv/d210544
Walsh PS, Metzger DA, Higuchi R. 2013. Chelex 100 as a Medium for Simple Extraction of DNA for PCR-Based Typing from Forensic Material. BioTechniques 54(3): 506–513.
https://doi.org/10.2144/000114018
Ward AK, Classen HL, Buchanan FC. 2009. Fishy-egg tainting is recessively inherited when brown-shelled layers are fed canola meal. Poult. Sci. 88(4): 714-721
Ward RD, Holmes BH, O’Hara TD. 2008. DNA barcoding discriminates echinoderm species. Mol Ecol Res. 8: 1202–1211.
Webb KE, Barnes DKA, Clark MS, Bowden DA. 2006. DNA barcoding: a molecular tool to identify Antarctic marine larvae. Deep-Sea Res Part II Trop Stud Ocean 53: 1053–1060.
Wilkinson CR. 2004. Status of coral reefs of the world: 2004. Global Coral Reef Monitoring Network GCRMN, Australian Institute of Marine Science, Townsville. Queensland, Australia.
56
Discussion and feedback