IDENTIFIKASI MOLEKULER BAKTERI STREPTOCOCCUS YANG BERASOSIASI DENGAN IKAN KERAPU YANG DIPERJUALBELIKAN DI PASAR-PASAR IKAN DI BALI
on
JURNAL BIOLOGI XVIII (1) : 10 - 13
ISSN : 1410-5292
IDENTIFIKASI MOLEKULER BAKTERI STREPTOCOCCUS YANG BERASOSIASI DENGAN IKAN KERAPU YANG DIPERJUALBELIKAN DI PASAR-PASAR IKAN DI BALI
MOLECULAR IDENTIFICATION OF STREPTOCOCCAL BACTERIA IN GROUPER FISHES TRADED IN BALI FISH MARKETS
I.B. Oka Suyasa1, I G.N.K. Mahardika2, Yan Ramona3 1Politeknik Kesehatan Denpasar, Bali
2Lab.Biomedik dan Biologi Molekuler, FKH Universitas Udayana, Denpasar, Bali 3Laboratorium Biosience dan Biotecnology, Universitas Udayana, Jimbaran, Badung, Bali
INTISARI
Penelitian ini bertujuan untuk mengidentifikasi bakteri Streptococcus sp. yang berasosiasi pada ikan Kerapu dengan menggunakan teknik-teknik molekuler Polymerase Chain Reaction (PCR) agar diperoleh identitas definitive bakteri tersebut. Dalam penelitian ini, sekuen 16S RNA marker dari bakteri-bakteri tersebut dijadikan target untuk diamplifikasi dengan metode PCR. Sekuensing produk PCR dilakukan di Berkeley Sequencing Facility, USA. Hasil sekuen nukleotida yang diperoleh selanjutnya di aligned dan di BLAST dengan menggunakan software MEGA 5.0. Hasil penelitian menunjukkan bahwa teridentifikasi sebanyak dua spesies bakteri Streptococcus (sementara diberi nama Streptococcus sp. isolat A dan B). Streptococcus sp. isolat A ditemukan pada semua lokasi pengambilan sampel ikan, sementara itu isolat B hanya ditemukan pada ikan yang diambil dari pasar ikan Karangasem. Berdasarkan nilai Haplotype Diversity (HD) nya, variasi genetik bakteri Streptococcus sp. pada ikan Kerapu tergolong sangat rendah.
Kata kunci: ikan Kerapu, Streptococcus sp., 16S rRNA, Bali.
ABSTRACT
A research on the molecular identification of streptococcal bacteria associated with grouper fish was conducted in order to find out the definite identity of these bacteria. Polymerase chain reaction (PCR) technique was employed and amplified using 16S rRNA marker. Sequences of each sample ware determined by sequencing technique, performed at the Berkeley Sequencing Facility, USA. Sequences obtained in this study were aligned and BLAST in MEGA 5.0TM. The results showed that two species of streptococcal bacteria (Streptococcus sp. isolates A and B) were successfully identified in this study. The former was found throughout all research locations, while the later was only found in Karangasem fish market. The genetic variation of bacteria belong to this Streptococcal group was found to be extremely low, based on its Haplotype Diversity (HD) value.
Keywords: Grouper, Streptococcus sp., 16S rRNA, Bali.
PENDAHULUAN
Saluran pencernaan (gastrointestinal) ikan laut merupakan salah satu tempat terjadinya interaksi antara bakteri dengan inangnya. Pada saluran pencernaan ikan terdapat ekosistem yang komplek dengan mikrobiota khusus yang terdiri dari bakteri aerobik, anaerob fakultatif, dan anaerob obligat (Cahill, 1990; Gómez and Balcazar, 2008).
Komposisi bakterinya dapat bervariasi sesuai dengan usia, status gizi, kondisi lingkungan, dan kompleksitas sistem pencernaan ikan (Cahill, 1990; Ringo et al., 2003). Stabilitas flora usus merupakan faktor yang sangat penting dalam resistensi alami ikan terhadap infeksi bakteri patogen yang ada di dalam saluran pencernaannya (Ringo et al., 2003).
Populasi mikroorganisme di dalam usus ikan dapat mencapai 107 sel per gram isi usus (Feliatra et al.,2004). Mikroorganisme yang secara alamiah menghuni
saluran pencernaan makhluk hidup (mikroflora) dapat memberi pengaruh positif maupun negatif pada fungsi fisiologis saluran pencernaan ikan. Mikroflora asli saluran pencernaan memanfaatkan inang sebagai tempat hidupnya. Bakteri usus dapat mensintesis vitamin, membantu pencernaan nutrien, dan menekan pertumbuhan bakteri patogen sehingga dapat melindungi inang terhadap penyakit serta merangsang fungsi kekebalan tubuh (Pelczar dan Chan, 1988).
Pada tahun 2004, Feliatra et al. melaporkan sebanyak sembilan spesies bakteri probiotik teridentifikasi di dalam saluran pencernaan ikan Kerapu Macan. Kesembilan spesies bakteri tersebut adalah Lactococcus sp., Carnoacterium sp., Staphylococcus sp., Bacillus sp., Eubacterium sp., Pseudomonas sp., Lactobacillus sp., Micrococcus sp., dan Bifidobacterium sp..
Sementara itu, Shickney (2000) menyatakan bahwa salah satu bakteri penyebab penyakit pada ikan Kerapu dan Snaper adalah kelompok Streptococcus.
Streptococcosis merupakan penyakit yang disebabkan oleh bakteri Streptococcus pada ikan Kerapu. Tanda-tanda dari infeksi ini tidak begitu jelas, namun gejala yang sering terlihat pada ikan adalah kelelahan, berenang tidak teratur, dan terjadi perdarahan pada bagian kornea. Menurut Murtidjo (2002) Streptococcus sp. termasuk bakteri yang resisten terhadap berbagai jenis antibiotik.
Dalam beberapa tahun terakhir, probiotik yang diisolasi dari organisme perairan banyak digunakan sebagai metode alternatif penanggulangan infeksi pada organisme akuakultur. Probiotik dipilih sebagai alternatif pencegahan infeksi pada ikan karena bakteri ini mampu menstimulasi sistem imun inangnya, memproduksi senyawa-senyawa penghambat pertumbuhan patogen, merangsang pertumbuhan, dan memperbaiki kualitas nutrien pada inangnya. Streptococcus cremoris misalnya, merupakan salah satu strain bakteri asam laktat yang mampu menekan pertumbuhan Vibrio alginolyticus secara in vitro dan in vivo pada udang Penaeus indicus (Ajitha et al., 2004).
Berdasarkan pada latar belakang di atas, maka perlu dilakukan ekplorasi bakteri Streptococcus yang berasosiasi dengan ikan Kerapu yang memiliki nilai ekonomis yang cukup tinggi di Bali, sehingga diperoleh data base tentang bakteri yang berperan dalam kesehatan ikan serta dapat diusulkan metode yang sesuai dan ramah lingkungan dalam penanggulangan masalah kesehatan pada ikan Kerapu.
BAHAN DAN METODE
Rancangan penelitian ini menggunakan metode deskriptif eksploratif observasional. Ikan Kerapu diambil dari pasar-pasar ikan yang ada di Kabupaten Badung, Klungkung, Karangasem dan Singaraja. Penelitian ini dilaksanakan dalam kurun waktu Oktober 2012 sampai dengan Mei 2013.
Feses ikan Kerapu (tempat terjadinya asosiasi dengan bakteri) diperoleh dengan cara membedah bagian perut ikan dan memotong bagian ususnya yang berisi feses. Selanjutnya, sebanyak satu ose feses ini ditanam pada media Blood Agar dan Mac Concey Agar, diinkubasikan selama 24 jam pada temperatur 37ºC, dilakukan pewarnaan gram untuk koloni-koloni yang melakukan hemolisis pada medium blood agar, dan hasilnya diamati dengan mikroskop cahaya dengan pembesaran 1000 kali. Koloni-koloni yang sel-selnya berbentuk bulat dan tersusun seperti rantai selanjutnya diisolasi, dimurnikan, dan di subkultur pada medium Gliserol 3%, sebelum dilakukan identifikasi molekuler yang diawali dengan ekstraksi DNA nya.
Ekstraksi DNA bakteri dilakukan dengan cara mengambil satu koloni bakteri yang dicurigai sebagai Streptococcus sp. dengan menggunakan jarum ose, dimasukkan ke dalam tabung eppendorf yang sudah berisi 250 µl chelex 10% (Walsh et al., 1991) dan diberi label, divortex selama 30 detik, dispin dengan kecepatan 15.000 rpm selama 30 detik menggunakan microcentrifuge, diinkubasikan selama 30 menit pada temperatur 95ºC menggunakan heating block, divortex
kembali selama 30 detik, dan dispin selama 30 detik dengan kecepatan 15.000 rpm. Sebelum diamplifikasi dengan PCR, sampel disimpan dalam lemari es pada temperature 2-8ºC selama semalam.
Total volume PCR reaction mixes adalah sebanyak 25 µl yang terdiri dari 14.5 µl ddH2O, 2.5 µl 10X PCR Buffer (Gold), 2.5 µl dNTPs (8 mM), 2.0 µl MgCl2 Solution (25 mM), 1.25 µl dari masing-masing primer (10 µM), 0.125 µl Amplitaq Gold (5 units/µl), dan 1µl DNA template. Primer yang dipakai adalah Primer 16S rRNA untuk Streptococcus (Picard et al., 2004): Str1 (5′-GTACAGTTGCTTCAGGACGTATC-3′) dan Str2 (5′-ACGTTCG ATTTCATCACGTTG-3′).
Amplifikasi DNA dilakukan dengan menggunakan alat Thermal Cycler (Applied Biosystem). Pre PCR dilakukan selama 10 menit pada temperature 94°C dan dilanjutkan dengan 38 siklus PCR dengan tahapan denaturasi selama 30 detik pada temperatur 94°C, annealing selama 30 detik pada temperatur 50°C, dan extention selama 45 detik pada temperatur 72°C. Setelah 38 siklus terlampaui, dilakukan Post PCR selama 10 menit pada temperatur 72°C dan 1 menit pada temperatur 24°C.
Hasil proses PCR ini selanjutnya dielektroforesis untuk melihat tingkat keberhasilan proses. Pada tahap ini, sebanyak 4 µl produk PCR ditambahkan dengan 1µl Loading Dye (Bromphenol-blue dan Cyline Cyanol), dan dielektroforesis pada 1% gel agarose dalam buffer TAE (0,5 gram agarose ditambah dengan 50 ml TAE) yang telah diisi sebanyak 4 µl etidium bromide (EtBr). Elektroforesis dilakukan pada tegangan 100 V dan kuat arus sebesar 400 mA selama 30 menit. Marker yang dipakai adalah 100 bp DNA Ladder (Invitrogen). Hasil elektroforesis ini divisualisasi di bawah sinar ultraviolet dan difoto dengan menggunakan kamera digital.
Pengurutan atau sekuensing DNA hasil PCR diatas dilakukan di Barkeley Sequencing Facility di USA. Data sekuens selanjutnya diedit dengan menggunakan Clustal W dalam program MEGA 5 (Tamura et al., 2011) dan hasilnya dibandingkan dengan sekuen yang ada pada GeneBank dengan menggunakan fasilitas BLAST search yang terdapat pada situs NCBI (http://www.ncbi.nlm. nih.gov).
HASIL
Koloni bakteri streptococcus secara makroskopis tampak berbentuk bundar, berdiameter 1 mm, cembung, halus, dan transparan. Secara mikroskopis (hasil pewarnaan gram) sel-selnya berbentuk bulat tunggal, tersusun berantai, dan gram positif. Dengan mengaplikasikan teknik-teknik molekuler (PCR), terdapat sebanyak 27 fragmen DNA ribosom (lokus 16S rRNA) sampel bakteri yang berhasil diamplifikasi, dengan panjang produk PCR sekitar 198 pasang basa (bp). Salah satu hasil elektroforesis dari produk PCR ditampilkan pada Gambar 1.
Hasil BLAST dari produk PCR yang ditelusuri pada DNA database GeneBank melalui situs National Center for Biotechnology Information (NCBI), National Institute for Health, USA, diperoleh hasil seperti yang ditunjukkan pada Tabel 1.
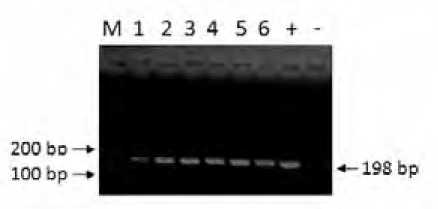
Gambar 1. Produk PCR Bakteri Streptococcus sp. pada Agarose 1%.
Keterangan: M=Marker Low DNA Mass Ladder (Invitrogen), 1-6: DNA sampel bakteri Streptococcus sp.
Tabel 1. Hasil Blast Bakteri Streptococcus
Ho-
Asal No Kode Sekuen
sa o oe e uen Hasil Blast mologi Ket
Sampel (bp) ( % )
|
Kedo-nganan |
1 |
KDIV.OA.05 |
191 |
Streptococcus suis strain 9801, JX436507.1 |
95 |
Unconfirm |
|
(Badung) |
2 |
KDIII.OA.03 |
189 |
Streptococcus suis strain 9801, JX436507.1 |
95 |
Unconfirm |
|
3 |
KDV.OA.03 |
190 |
Streptococcus suis strain 9801, JX436507.1 |
95 |
Unconfirm | |
|
4 |
KDF.02 |
137 |
Streptococcus suis strain 9801, JX436507.1 |
96 |
Unconfirm | |
|
5 |
KDE.04 |
137 |
Streptococcus suis strain 9801, JX436507.1 |
96 |
Unconfirm | |
|
6 |
KDH.01 |
138 |
Streptococcus suis strain 9801, JX436507.1 |
96 |
Unconfirm | |
|
Gerok-gak |
7 |
BLI.02 |
139 |
Lactococcus lactis strain KA-12, HQ694960.1 |
100 |
Confirm (sp lain) |
|
(Singa-raja) |
8 |
BLI.04 |
137 |
Lactococcus lactis strain KA-12, HQ694960.1 |
99 |
Confirm (sp lain) |
|
9 |
BLI.06 |
134 |
Streptococcus suis strain 9801, JX436507.1 |
96 |
Unconfirm | |
|
10 |
BLI.07 |
131 |
Streptococcus suis strain 9801, JX436507.1 |
96 |
Unconfirm | |
|
11 |
BLI.08 |
138 |
Streptococcus suis strain 9801, JX436507.1 |
96 |
Unconfirm | |
|
12 |
BLI.10 |
124 |
Streptococcus suis strain 9801, JX436507.1 |
96 |
Unconfirm | |
|
Kusamba (Klung- |
13 |
KLE.01 |
190 |
Streptococcus suis strain 9801, JX436507.1 |
96 |
Unconfirm |
|
kung) |
14 |
KLE.02 |
134 |
Streptococcus suis strain 9801, JX436507.1 |
96 |
Unconfirm |
|
15 |
KLH.01 |
131 |
Streptococcus suis strain 9801, JX436507.1 |
96 |
Unconfirm | |
|
16 |
KLH.04 |
146 |
Streptococcus suis strain 9801, JX436507.1 |
96 |
Unconfirm | |
|
17 |
KLD.01 |
134 |
Streptococcus suis strain 9801, JX436507.1 |
96 |
Unconfirm | |
|
18 |
KLD.02 |
125 |
Streptococcus suis strain 9801, JX436507.1 |
96 |
Unconfirm | |
|
19 |
KLG.01 |
136 |
Streptococcus suis strain 9801, JX436507.1 |
96 |
Unconfirm | |
|
20 |
KLH.02 |
137 |
Streptococcus suis strain 9801, JX436507.1 |
96 |
Unconfirm | |
|
Antiga (Karan- |
21 |
KRN.01 |
136 |
Streptococcus suis strain 9801, JX436507.1 |
96 |
Unconfirm |
|
gasem) |
22 |
KRN.02 |
134 |
Streptococcus parasan-guinis, CP003122.1 |
93 |
Unconfirm |
|
23 |
KRL.01 |
136 |
Streptococcus suis strain 9801, JX436507.1 |
96 |
Unconfirm | |
|
24 |
KRG.01 |
146 |
Streptococcus suis strain 9801, JX436507.1 |
96 |
Unconfirm | |
|
25 |
KRE.01 |
146 |
Streptococcus suis strain 9801, JX436507.1 |
96 |
Unconfirm | |
|
26 |
KRP.01 |
136 |
Streptococcus suis strain 9801, JX436507.1 |
96 |
Unconfirm | |
|
27 |
KRM.01 |
137 |
Streptococcus suis strain |
96 |
Unconfirm |
9801, JX436507.1
Berdasarkan pada hasil BLAST (Tabel 1.), terdapat 2 sampel (yang berasal dari Buleleng) yang teridentifikasi sebagai Lactococcus lactis (dengan homologi sebesar 99-100%). Sisanya, sebanyak 25 sampel teridentifikasi sebagai genus Streptococcus (belum teridentifikasi sampai ke spesies karena data di GeneBank belum tersedia). Dari 25 sampel ini, terdapat sebanyak 24 sampel yang memiliki homologi sebesar 95-96% dengan Streptococcus suis, dan sisanya memiliki homologi sebesar 93% dengan Streptococcus parasanguinis. Sebaran Streptococcus sp. yang ditemukan pada ikan Kerapu yang diperjualbelikan di Bali dapat dilihat pada Tabel 2.
Tabel 2. Jumlah Isolat Streptococcus sp. yang Ditemukan Pada Ikan Kerapu dari Empat Pasar Ikan di Bali
|
No |
Genus Streptococcus |
KD |
Asal BL KL |
KR |
Total |
|
1 |
Streptococcus sp.isolat A |
6 |
48 |
6 |
24 |
|
2 |
Streptococcus sp.isolat B |
0 |
00 |
1 |
1 |
Keterangan: KD=Kedonganan, BL=Buleleng, KL=Klungkung, KR=Karangasem
Konstruksi pohon filogeni, yang dibuat dengan metode neighbor joining dan model Kimura 2 parameter, dan jarak evolusionernya dihitung dengan menggunakan metode Maximum Composite Likelihood (Tamura et al., 2011), diperoleh hasil seperti yang ditunjukkan pada Gambar 2.
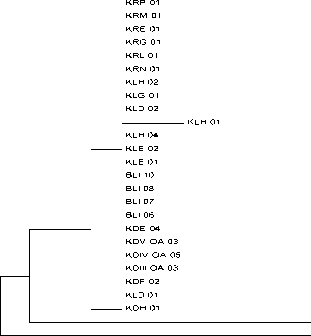
Streptococcus suis strain 9801 JX436507.1
KRN.02
0.005
Gambar 2. Pohon Filogeni Bakteri Streptococcus yang Diperoleh dari Hasil Penelitian yang Dibandingkan dengan Data yang ada pada GeneBank.
PEMBAHASAN
Berdasarkan pada Tabel 1 dan 2, untuk sementara sebanyak 24 isolat diberi nama Streptococus sp. isolat A dan Streptococus sp. isolat B. Isolat A ditemukan pada tujuh spesies ikan Kerapu (Epinephelus fuscoguttatus, Epinephelus polyphekadion, Cephalopholis cyanostigma, Cephalopholis miniata, Cephalopholis sonnerati, Cephalopholis leopardus, Variola albimarginata) yang berasal dari seluruh lokasi penelitian, dengan jarak genetik sesama isolat sebesar 0,000. Hasil ini
menunjukkan bahwa isolat-isolat tersebut merupakan spesies yang sama.
Sementara itu, isolat yang diberi nama Streptococcus sp. isolat B hanya ditemukan pada spesies ikan Kerapu Cephalopholis leopardus yang berasal dari Karangasem. Jarak genetik isolat ini dengan 24 isolat yang lain adalah 0,053. Ini menandakan bahwa isolat B berbeda spesies dengan yang lain.
Pada penelitian ini, Streptococcus sp. isolat A lebih mudah ditemukan pada sampel ikan Kerapu jika dibandingkan dengan isolat B. Hasil ini mengindikasikan bahwa isolat A kemungkinan besar merupakan flora normal pada ikan Kerapu. Streptococcus sp. merupakan salah satu mikroflora normal yang ditemukan pada saluran pencernaan ikan Gurami (Aslamyah et al., 2009).
Terbatasnya jumlah isolat B yang berhasil diisolasi pada penelitian ini disebabkan oleh keterbatasan pengetahuan peneliti tentang komposisi medium yang diperlukan oleh kelompok bakteri ini pada kultur in vitro. Hal serupa juga dilaporkan oleh Wang (1996) yang menyatakan bahwa tidak semua bakteri dapat tumbuh pada suatu media kultur sintetik. Oleh karena itu, penelitian ini perlu dilanjutkan dengan menggunakan metode kultur yang lebih disempurnakan, sehingga jumlah jenis bakteri yang berasal dari feses ikan Kerapu yang ditangkap dari laut (wild grouper) dapat ditingkatkan. Untuk mengatasi kesulitan identifikasi semua bakteri streptococcus yang berasosiasi dengan ikan Kerapu akibat dari kesulitan mengkultur secara in vitro, maka perlu diaplikasikan teknik PCR in situ atau culture independent PCR. Bakteri-bakteri yang tidak dapat dikultur secara in vitro akan terdeteksi dan teridentifikasi melalui aplikasi teknik ini (Kannan et al., 2001).
Analisis keragaman genetik dari bakteri Streptococcus sp. diperoleh nilai HD (Haplotype Diversity) isolat A yang tergolong rendah (0,163). Hal ini menunjukkan bahwa keragaman bakteri ini tergolong rendah (Rozas et al., 2009) atau secara implisit memberikan informasi bahwa telah terjadi genetic flow diantara mereka karena kelompok bakteri ini hanya menempati area saluran pencernaan ikan.
SIMPULAN
Dari 85 sampel ikan Kerapu yang didapat dari pasar-pasar ikan di Bali, teridentifikasi sebanyak 24 isolat Streptococcus yang sementara diberi nama Streptococus sp. isolat A dan isolat B. Isolat A ditemukan pada tujuh spesies ikan (Epinephelus fuscoguttatus, Epinephelus polyphekadion, Cephalopholis cyanostigma, Cephalopholis miniata, Cephalopholis sonnerati, Cephalopholis leopardus, Variola albimarginata) yang berasal dari seluruh lokasi penelitian. Sementara itu, Streptococcus sp. isolat B, hanya ditemukan pada Cephalopholis leopardus yang diperoleh dari pasar ikan di Karangasem.
KEPUSTAKAAN
Ajitha, S., M. Sridhar, N. Sridhar, I.S.B. Singh, V.Varghese. 2004. Probiotic Effects of Lactic Acid Bacteria Against Vibrio algi-nolyticus in Penaeus (Fenneropenaeus) indicus (H. Milne Edwards). Asian Fish. Sci.17: 71-80.
Aslamyah, S., H.Y. Azis, Sriwulan, K.G. Wiryawan. 2009. Mikroflora Saluran Pencernaan Ikan Gurame (Osphronemus gouramy Lacepede). Torani. 19(1): 66-73.
Barrow, G.I., R.K.A. Feltham. 1993. Cowan and Steel’s Manual for The Identification of Medical Bacteria. Cambridge: Cambridge University Press.
Cahill, M.M. 1990. Bacterial flora of fishes: a review. Microb. Ecol. 19: 21–41.
Feliatra., I. Efendi, E. Suryadi. 2004. Isolasi dan Identifikasi Bakteri Probiotik dari Ikan Kerapu Macan (Ephinephelus fuscogatus) dalam Upaya Efisiensi Pakan Ikan. Jurnal Natur Indonesia. 6(2): 75-80.
Gomez, G.D., J. L. Balcazar. 2008. A Review on The Interactions Between Gut Microbiota and Innate Immunity of Fish. FEMS. Immunol. Med. Microbiol. 52: 145-154.
Holt, J.G., N.R. Kreig, P.H.A. Sneath, J.T. Staley, S.T.Williams. 2000. Bergey’s Manual of Determinative Bacteriology. Ninth Edition. Lippincott Williams and Wilkins, Philadelphia.
Kannan S., P. S. Kanna, K. Karkuzhali, U.K. Chattopadhyay, D. Pal. 2001. Direct detection of diarrheagenic Aeromonas from faeces by polymerase chain reaction (PCR) targeting aerolysin toxin gene. European Review for Medical and Pharmacological Sciences 5: 91-94.
Murtidjo, B.A. 2002. Budi Daya Kerapu dalam Tambak. Kanisius. Yogyakarta.
Picard, F.J., D. Ke, D.K. Boudreau, M. Boissinot, A. Huletsky, D. Richard, M. Ouellette, P.H. Roy, M.G. Bergeron. 2004. Use of Tuf Sequences for Genus-Specific PCR Detection and Phylogenetic Analysis of 28 Streptococcal Species. J. Clin. Microbiol. 42: 3686–3695.
Pelczar, M.J.J., E.C.S. Chan. 1986. Dasar-Dasar Mikrobiologi. Penerjemah R.S. Hadioetomo, T. Imas, S.S. Tjitrosomo, S.L. Angka. Volume II. Jakarta: UI Press
Ringo, E., R.E. Olsen, T.M. Mayhew, R. Myklebust. 2003. Electron Microscopy of The Intestinal Microflora of Fish. Aquaculture. 227: 395–415.
Rozas, J., J.C. Sanchez-Delbarrio, X. Messeguer, R. Rozas. 2009. DNASp, DNA Polymorphism Analyses by Coaescent and Other Methods. Bioinformatics 19: 2496-2497.
Shickney, R. R. 2000. Encyclopedia of Aquaculture. John Wiley and Sons. Inc. 418 – 421.
Sugama, K., Wardoyo, D. Rohaniawan, H. Matsuda. 1988. Teknologi Pembenihan Ikan Kerapu Tikus. Prosiding Seminar Teknologi Perikanan Pantai. Jica ATA-379: 80-88.
Tamura, K., D. Peterson, N. Peterson, G. Stecher, M. Nei, S. Kumar. 2011. MEGA 5 : Molecular Evolutionary Genetics Analysis Using Maximum Likelihood, Evolutionary Distance and Maximum Parsimony Methods. Mol.Biol.Evol.10.1093/ molbev/msr121.
Walsh, P.S., D.A. Metzger, R. Higuchi. 1991. Chelex-100 as a Medium for Simple Extraction of DNA for PCR Based Typing from Forensic Material. Biotechniques 10: 506–513.
Wang, R., W. Cao, C. Cerniglia. 1996. PCR Detection and Quantitation of Predominant Anaerobic Bacteria in Human and Animal Fecal Samples. Applied and Environmental Microbiology. 62(4): 1242-1247.
13
Discussion and feedback